「伝令RNA」の版間の差分
→外部リンク: 脳科学辞典該当項目へのリンク作成 |
英語版 Messenger RNA の 2022-09-24T14:23:18(UTC)版を翻訳してマージした。(詳細はノート:伝令RNA#英語版による記事の強化についてを参照) タグ: サイズの大幅な増減 ビジュアルエディター 曖昧さ回避ページへのリンク |
||
| 1行目: | 1行目: | ||
{{Hatnote|[[ミトコンドリアDNA]] (mDNA) や、[[マイクロRNA]] (miRNA) と混同しないでください。}} |
|||
{{出典の明記|date=2011年12月}} |
|||
[[Image:MRNA-interaction.png|thumb|upright=1.2|[[真核細胞]]におけるmRNAのライフサイクルを示す模式図。mRNAは[[細胞核]]内で[[デオキシリボ核酸|DNA]]から[[転写 (生物学)|転写]]されて作られる。次に[[転写後修飾]](プロセシング)が行われ、[[細胞質]]へ輸送される。その後mRNAは[[リボソーム]]や[[転移RNA|tRNA]]と相互作用して[[翻訳 (生物学)|翻訳]]され、[[タンパク質]](または[[ペプチド]])分子が作られる。最終的にmRNAは分解される。]] |
|||
[[File:Peptide syn.png|thumb|right|400px|[[リボソーム]]は、一連の'''伝令RNA'''([[:en: Messenger RNA]])を読み取り、[[転移RNA]]([[:en: Transfer RNA]] (TRNA))に結びついた[[アミノ酸]]から所定の[[タンパク質]]を組み立てる。]] |
|||
[[File:Protein translation.gif|thumb|right|250px|[[小胞体]]に分泌されるタンパク質を組み立てている[[リボソーム]]]] |
|||
[[分子生物学]]において、{{読み仮名|'''伝令RNA'''|でんれいRNA|メッセンジャーRNA、{{lang-en-short|messenger RNA}}}}は、[[蛋白質]]に[[翻訳 (生物学)|翻訳]]され得る[[塩基配列]]情報と構造を持った[[リボ核酸|RNA]]のことであり、通常'''mRNA'''と表記される。[[DNA]]に比べてその長さは短い。DNAからコピーした遺伝情報を担っており、その遺伝情報は、特定のアミノ酸に対応する[[コドン]]と呼ばれる3塩基配列という形になっている。 |
|||
[[分子生物学]]において、'''伝令RNA'''(でんれいアールエヌエー、{{Lang-en-short|messenger ribonucleic acid}})は、'''mRNA'''または'''メッセンジャーリボ核酸'''(メッセンジャーリボかくさん)とも呼ばれ、[[遺伝子]]の[[遺伝子配列]]に対応する一本鎖の[[リボ核酸]](RNA)[[分子]]であり、[[タンパク質]]を[[タンパク質生合成|合成]]する過程で[[リボソーム]]によって読み取られる。 |
|||
mRNAはDNAから写し取られた遺伝情報に従い、タンパク質を合成する(詳しくは[[翻訳 (生物学)|翻訳]])。翻訳の役目を終えたmRNAは細胞に不要としてすぐに分解され、寿命が短く、分解しやすくするために1本鎖であるともいわれている。 |
|||
mRNAは、[[RNAポリメラーゼ]]という[[酵素]]が遺伝子を[[一次転写産物]]の[[mRNA前駆体]](pre-mRNAとよぶ)に変換する[[転写 (生物学)|転写]]の過程で作られる。このpre-mRNAには通常、最終的な[[アミノ酸配列]]を[[遺伝暗号|コード]]しない[[イントロン]]という領域が含まれている。これらは[[RNAスプライシング]]の過程で除去され、タンパク質をコードする領域である[[エクソン]]のみが残る。このエクソン配列が[[成熟mRNA]]を構成する。次に、成熟mRNAはリボソームによって読み取られ、リボソームは[[転移RNA]](tRNA)が運ぶ[[アミノ酸]]を利用して、タンパク質を作り出す。この過程は[[翻訳 (生物学)|翻訳]]として知られている。これらの過程はすべて、{{ill2|生物系|en|Biological system}}における遺伝情報の流れを説明する分子生物学の[[セントラルドグマ]]の一部を形成している。 |
|||
[[古細菌]]および[[細菌]]では[[転写 (生物学)|転写]]されたRNAはほぼそのままでmRNAとして機能する。一方[[真核生物]]では転写された[[mRNA前駆体]]はいくつかの切断(スプライシング)、修飾といったプロセシングを受けたのちに成熟mRNAになる。 |
|||
mRNAの遺伝情報は、[[デオキシリボ核酸]](DNA)と同様に[[ヌクレオチド]]配列に含まれ、おのおのが3連の[[リボヌクレオチド]]からなる[[コドン]]に配列されている。各コドンは、特定の[[アミノ酸]]をコードしているが、タンパク質合成を停止させる[[終止コドン]]は例外である。コドンからアミノ酸へ翻訳するためには、コドンを認識して対応するアミノ酸を供給する転移RNAと、リボソームに含まれるタンパク質製造装置の中心的な構成要素である[[リボソームRNA]](rRNA)の2種類のRNAが必要である。 |
|||
真核生物のmRNAは[[RNAポリメラーゼII]]によって転写されたRNAに由来する。5'末端にはm7Gキャップがあり、3'末端は一般にポリアデニル化される(poly (A)鎖で終了している)。これらの構造やmRNAの塩基配列は翻訳活性やmRNAの分解を制御する機能も持っている。古細菌、細菌も3'末端に短いpoly (A)鎖を持つが、5'末端のキャップ構造は持たない。 |
|||
mRNAの概念は、1960年に[[シドニー・ブレナー]]と[[フランシス・クリック]]によって発展した([[利用者:YasuakiH/sandbox#歴史|歴史]]を参照)。実験検証を行う過程で、[[フランソワ・ジャコブ]]と[[ジャック・モノー]]が「メッセンジャーRNA(''messenger RNA'')」という名称を作り出した。1961年、[[ジェームズ・ワトソン]]の研究チームと、ジャコブ、モノー、[[マシュー・メセルソン]]のチームによって、mRNAが単離され、独立して記述された。 |
|||
poly (A)鎖は[[rRNA]]や[[tRNA]]には存在しないmRNAの特徴であるとされており、このことを利用してmRNAを特異的に精製することができる。また、mRNAを鋳型にして[[デオキシリボ核酸|DNA]]を[[逆転写酵素]]によって合成することができ、これは[[相補的DNA|cDNA]]と呼ばれる。cDNAは[[遺伝子]]が働いていることの非常に信頼性の高い証拠であり、[[ゲノムプロジェクト]]によって得られた大量の[[シークエンス]]データの中から遺伝子を探す作業を補助することができる。 |
|||
{{toclimit|3}} |
|||
== {{Anchors|Synthesis, processing and function}}合成、プロセシング、働き == |
|||
伝令RNAの存在を最初に理論的に示唆したのは、1965年に揃って[[ノーベル生理学医学賞]]を受賞したフランスの生物学者[[ジャック・モノー]](1910年〜1976年)と[[フランソワ・ジャコブ]](1920年〜2013年)であった<ref>F. Jacob, J. Monod |
|||
[[File:DNA transcription.svg|thumb|RNAポリメラーゼがDNA鎖を転写してmRNAを形成する]] |
|||
"Genetic regulatory mechanisms in the synthesis of proteins". |
|||
mRNA分子は転写から始まり、最終的に分解されて短い生涯を終える。mRNA分子はその寿命の間、翻訳前に{{Ill2|生物学的過程|en|Biological process|label=プロセシング}}、編集、そして輸送されることもある。真核生物のmRNA分子は、しばしば広範なプロセシングや輸送を必要とするが、[[原核生物]]のmRNA分子はそうではない。[[真核生物]]のmRNA分子とそれに結合したタンパク質を合わせて{{Ill2|メッセンジャーRNP|en|Messenger RNP}}と呼ぶ。 |
|||
Journal of Molecular Biology, 3 (1961), pp. 318-356.</ref>。それに引き続いて1961年のうちに、フランソワ・ジャコブ自身や、[[南アフリカ連邦]]生まれの生物学者[[シドニー・ブレナー]](1927年〜2019年。2002年の[[ノーベル生理学医学賞]]受賞者)、アメリカ合衆国の[[遺伝学]]・[[分子生物学]]者[[マシュー・メセルソン]](1930年〜)らによって、その存在が実証された<ref>{{cite journal |last1=Cobb |first1=Matthew |authorlink1= Matthew Cobb |title=Who discovered messenger RNA? |journal=Current Biology |date=29 June 2015 |doi=10.1016/j.cub.2015.05.032 |url=https://www.sciencedirect.com/science/article/pii/S0960982215006065 |accessdate=2 July 2020 |volume=25 |issue=13 |pages=R526–R532|pmid=26126273 |doi-access=free }}</ref>。 |
|||
== |
=== 転写 === |
||
{{main|[[転写 (生物学)]]}} |
|||
遺伝子発現のプロセスの一つ、[[転写]]は[[細胞核]]内にて行われる。[[DNA]]に刻まれた[[遺伝情報]]([[遺伝子]])は、RNA合成酵素によりmRNAに転写される。DNAがすべて転写されるのではなく、必要な分だけ転写される。遺伝情報はmRNAの塩基によって[[コドン]]の形式でコードされ、全20種類の[[アミノ酸]]に対応している。遺伝情報を受け継いだmRNAは核から細胞質へ出て、[[リボソーム]]に付着する。ここでmRNAの遺伝情報に従い、特定のタンパク質が合成される。 |
|||
DNAからRNAをコピーすることを[[転写 (生物学)|転写]]という。転写の際、[[RNAポリメラーゼ]]は必要に応じてDNAからmRNAへの遺伝子コピーを作成する。この過程は真核生物と原核生物でわずかに相違する。顕著な相違の一つは、原核生物のRNAポリメラーゼは転写中にDNA処理酵素と結合し、転写中にプロセシングを進めることができる。それによって、新しいmRNA鎖はtRNA鎖と呼ばれる[[相補性 (分子生物学)#DNAとRNAの塩基対の相補性|相補鎖]]を生成して二本鎖となり、両者が結合すると[[塩基対]]形成による構造形成ができなくなる。さらに、mRNAの鋳型はtRNAの相補鎖であり、DNAが結合する[[アンチコドン]]配列と同じ配列である。短命で、未プロセシングあるいは部分的にプロセシングされた転写産物を前駆体mRNA、または[[Pre-mRNA|''pre-mRNA'']]と呼び、完全にプロセシングされると''[[成熟mRNA]]''と呼ぶ。 |
|||
=== 真核生物のpre-mRNAプロセシング === |
|||
== RNA 中の翻訳領域 == |
|||
{{main|転写後修飾}} |
|||
翻訳段階においてmRNA の情報は一部分しか解読されない。各mRNA の[[タンパク質]]翻訳領域は[[コーディング領域]]と呼ばれ、1つのコーディング領域は1つのタンパク質を指定している。翻訳は[[開始コドン]]から始まり、[[終止コドン]]で終了する。コーディング領域の両端がmRNA の両端に届くことはなく、開始コドン上流に[[5' 非翻訳領域]](five prime untranslated region:5' UTR)があり、終止コドン下流に[[3' 非翻訳領域]](three prime untranslated region:3' UTR)が存在する。 |
|||
[[File:Gene structure eukaryote 2 annotated.svg|thumb|(上段) DNA遺伝子はpre-mRNAに転写される。(中段) その後、pre-mRNAはプロセシングを経て成熟mRNAを形成する。(下段) 最終的に成熟mRNAはリボソームによって翻訳されてタンパク質が生成する。|440x440ピクセル]] |
|||
mRNAのプロセシングは、[[真核生物]]、[[細菌]]、および[[古細菌]]の間で大きく異なっている。非真核生物のmRNAは、本質的に転写された時点で成熟しており、まれな場合を除いてプロセシングを必要としない<ref>{{Cite book| vauthors = Watson JD |author-link=James Watson |title=Molecular Biology of the Gene, 7th edition|publisher=Pearson Higher Ed USA|date=February 22, 2013|isbn=9780321851499}}</ref>。しかし、真核生物のpre-mRNAは、細胞質へ輸送されリボソームにより翻訳される前に、一連のプロセシング段階を経る必要がある。 |
|||
==== スプライシング ==== |
|||
[[真核生物]]におけるmRNA の殆どは'''モノシストロン性のmRNA'''(monocistronic mRNA)で1つのタンパク質を翻訳する一方で、[[原核生物]]のmRNAの多くは'''ポリシストロン性のmRNA'''(polycistronic mRNA)で幾つかのタンパク質を翻訳する。 |
|||
{{main|{{ill2|RNAスプライシング|en|RNA splicing}}}} |
|||
[[RNAスプライシング]]は、真核生物のpre-mRNAが成熟mRNAに至る広範なプロセシングであり、[[イントロン]]や{{Ill2|アウトロン|en|Outron}}(非コード領域)が除去され、[[エクソン]](コード領域)が結合する機構である。 |
|||
==== 5'キャップの付加 ==== |
|||
また、近年ではpoly (A) 鎖や5' 末端のキャップ構造を持ちながら、コーディング領域を持たずに[[ノンコーディングRNA]]としてはたらく[[RNA]]も確認されている。 |
|||
{{main|5'キャップ}} |
|||
[[File:5' cap labeled.svg|thumb|5'キャップの構造]] |
|||
''[[5'キャップ]]''(''5' cap、''RNAキャップ、RNA [[7-メチルグアノシン]]キャップ、RNA m<sup>7</sup>Gキャップとも呼ばれる)とは、真核生物のメッセンジャーRNAの転写開始直後にその先端部つまり{{Ill2|5'末端|en|5' end}}に付加された修飾[[グアニン]]ヌクレオチドである。5'キャップは、末端の7-メチルグアノシン残基からなり、5'-5'-トリリン酸結合を介して最初の転写ヌクレオチドに結びつく。その存在は、[[リボソーム]]による認識と[[リボヌクレアーゼ]](RNase)酵素からの保護において重要である。 |
|||
キャップの付加は転写と連動しており、相互に影響を与えるように共転写的<!-- co-transcriptionally -->に行われる。転写開始の直後、合成されるmRNAの5'末端は、[[RNAポリメラーゼ]]に結合している{{Ill2|キャッピング酵素|en|Capping enzyme|label=キャップ結合複合体}}と結合する。この[[酵素]]複合体は、mRNAのキャッピングに必要な化学反応を[[触媒]]する。合成は多段階の[[生化学]]反応として進行する。 |
|||
== 脚注 == |
|||
{{脚注ヘルプ}} |
|||
==== 編集 ==== |
|||
{{Reflist}} |
|||
{{Main|RNAエディティング}} |
|||
場合によって、mRNAが[[RNAエディティング|編集]]されて、そのヌクレオチド組成が変化することがある。ヒトを例にとると、{{Ill2|アポリポタンパク質B|en|Apolipoprotein B}}のmRNAは、ある組織では編集されるが、他の組織では編集されない。この編集によって中途での終止コドン<!--early stop codon-->が作られ、翻訳時に短いタンパク質が生成する。 |
|||
==== ポリアデニル化 ==== |
|||
{{main|ポリアデニル化}} |
|||
[[File:Polyadenylation.png|thumb|ポリアデニル化の過程]] |
|||
[[ポリアデニル化]](''polyadenylation'')とは、メッセンジャーRNA分子にポリアデニリル部<!--polyadenylyl moiety-->を[[共有結合]]させることである。真核生物では、ほとんどのメッセンジャーRNA(mRNA)分子が3'末端でポリアデニル化されているが、最近の研究では、[[ウリジン]]の短い伸長(オリゴウリジル化<!-- oligouridylation -->)も一般的であることが示されている<ref name="Choi_2012">{{cite journal | vauthors = Choi YS, Patena W, Leavitt AD, McManus MT | title = Widespread RNA 3'-end oligouridylation in mammals | journal = RNA (New York, N.Y.) | volume = 18 | issue = 3 | pages = 394–401 | date = March 2012 | pmid = 22291204 | pmc = 3285928 | doi = 10.1261/rna.029306.111 }}</ref>。[[伝令RNA#Poly(A) tail|ポリ(A)テール]]とそれに結合したタンパク質は、エキソヌクレアーゼによる分解からmRNAを保護することを助ける。また、ポリアデニル化は、転写終結、mRNAの[[核外輸送]]、および翻訳にも重要である。原核生物では、mRNAがポリアデニル化されると、ポリ(A)テールがエキソヌクレアーゼ分解<!-- exonucleolytic degradation -->を妨げるのではなく、むしろ促進するように作用することもある。 |
|||
ポリアデニル化は、DNAからRNAへ転写される際、および(または)その直後に起こる。転写が終了すると、RNAポリメラーゼに結合するエンドヌクレアーゼ複合体の働きによって、mRNA鎖は切断される。mRNAが切断された後、切断部位の遊離3'末端に約250のアデノシン残基が付加される。この反応は、{{Ill2|ポリヌクレオチドアデニリルトランスフェラーゼ|en|Polynucleotide adenylyltransferase|label=ポリアデニル酸ポリメラーゼ}}によって触媒される。[[選択的スプライシング]]と同様に、1つのmRNAに複数種のポリアデニル化変異体が存在する可能性がある。 |
|||
また、ポリアデニル化部位の変異も起こる。遺伝子の一次RNA転写産物は、ポリA付加部位で切断され、RNAの3'末端に100-200個のアデノシン残基<!-- 訳注: 原文では単に"A"と書かれているが、前後の文よりアデノシン残基と判断した。 -->が付加される。この部位が変化すると、異常に長く、不安定なmRNAコンストラクトが形成される。 |
|||
=== 輸送 === |
|||
{{Main|核輸送#核外輸送}} |
|||
真核生物と原核生物のもう一つの違いは、mRNAの輸送に関するものである。真核生物では転写と翻訳は[[細胞内膜系|区画的に分割]]されているため、真核生物ではmRNAを[[細胞核]]から[[細胞質]]へ輸送しなくてはならない。この過程は、さまざまな[[シグナル伝達|シグナル伝達経路]]によって制御されている可能性がある<ref name="Quaresma2013">{{cite journal | vauthors = Quaresma AJ, Sievert R, Nickerson JA | title = Regulation of mRNA export by the PI3 kinase/AKT signal transduction pathway | journal = Molecular Biology of the Cell | volume = 24 | issue = 8 | pages = 1208–1221 | date = April 2013 | pmid = 23427269 | pmc = 3623641 | doi = 10.1091/mbc.E12-06-0450 }}</ref>。成熟mRNAは修飾の処理によって認識され、{{Ill2|キャップ結合複合体|en|Cap binding complex|label=キャップ結合タンパク質(CBC)}}であるCBP20およびCBP80<ref name="kierzkowski2009">{{cite journal | vauthors = Kierzkowski D, Kmieciak M, Piontek P, Wojtaszek P, Szweykowska-Kulinska Z, Jarmolowski A | title = The Arabidopsis CBP20 targets the cap-binding complex to the nucleus, and is stabilized by CBP80 | journal = The Plant Journal | volume = 59 | issue = 5 | pages = 814–825 | date = September 2009 | pmid = 19453442 | doi = 10.1111/j.1365-313X.2009.03915.x | doi-access = free }}</ref>、および[[核膜孔#RNAの核外輸送|転写/核外輸送複合体(TREX)]]に結合することによって、[[核膜孔]]から輸送される<ref name="strausser2002">{{cite journal | vauthors = Strässer K, Masuda S, Mason P, Pfannstiel J, Oppizzi M, Rodriguez-Navarro S, Rondón AG, Aguilera A, Struhl K, Reed R, Hurt E | title = TREX is a conserved complex coupling transcription with messenger RNA export | journal = Nature | volume = 417 | issue = 6886 | pages = 304–308 | date = May 2002 | pmid = 11979277 | doi = 10.1038/nature746 | bibcode = 2002Natur.417..304S | s2cid = 1112194 }}</ref><ref name="katahira2014">{{cite journal | vauthors = Katahira J, Yoneda Y | title = Roles of the TREX complex in nuclear export of mRNA | journal = RNA Biology | volume = 6 | issue = 2 | pages = 149–152 | date = 27 October 2014 | pmid = 19229134 | doi = 10.4161/rna.6.2.8046 | doi-access = free }}</ref>。真核生物では、複数のmRNA輸送経路が同定されている<ref name="Cenik2011">{{cite journal | vauthors = Cenik C, Chua HN, Zhang H, Tarnawsky SP, Akef A, Derti A, Tasan M, Moore MJ, Palazzo AF, Roth FP | title = Genome analysis reveals interplay between 5'UTR introns and nuclear mRNA export for secretory and mitochondrial genes | journal = PLOS Genetics | volume = 7 | issue = 4 | pages = e1001366 | date = April 2011 | pmid = 21533221 | pmc = 3077370 | doi = 10.1371/journal.pgen.1001366 }}</ref>。 |
|||
空間的に複雑な細胞では、いくつかのmRNAは特定の細胞内目的地<!--subcellular destinations-->に輸送される。成熟した[[神経細胞]]では、ある種のmRNAが[[細胞体|神経細胞体]]から[[樹状突起]]に輸送される。mRNA翻訳が行われる部位の一例は、シナプスの下に選択的に局在する[[ポリリボソーム]]である<ref>{{cite journal | vauthors = Steward O, Levy WB | title = Preferential localization of polyribosomes under the base of dendritic spines in granule cells of the dentate gyrus | journal = The Journal of Neuroscience | volume = 2 | issue = 3 | pages = 284–291 | date = March 1982 | pmid = 7062109 | pmc = 6564334 | doi = 10.1523/JNEUROSCI.02-03-00284.1982 }}</ref>。{{Ill2|活性調節細胞骨格関連タンパク質|en|Activity-regulated cytoskeleton-associated protein|label=Arc/Arg3.1}}のmRNAは、シナプス活動によって誘導され、[[NMDA受容体]]が生成するシグナルに基づいて、活動的なシナプス近傍に選択的に局在される<ref>{{cite journal | vauthors = Steward O, Worley PF | title = Selective targeting of newly synthesized Arc mRNA to active synapses requires NMDA receptor activation | journal = Neuron | volume = 30 | issue = 1 | pages = 227–240 | date = April 2001 | pmid = 11343657 | doi = 10.1016/s0896-6273(01)00275-6 | s2cid = 13395819 | doi-access = free }}</ref>。また、{{Ill2|βアクチン|en|Beta-actin|label=}}のmRNAのように、外部刺激に応答して樹状突起に移動するmRNAもある<ref name="Job1912">{{cite journal | vauthors = Job C, Eberwine J | title = Localization and translation of mRNA in dendrites and axons | journal = Nature Reviews. Neuroscience | volume = 2 | issue = 12 | pages = 889–898 | date = December 2001 | pmid = 11733796 | doi = 10.1038/35104069 | author-link2 = James Eberwine | s2cid = 5275219 }}</ref>。[[アクチン]]のmRNAは、細胞核から輸送されるときに、{{Ill2|ZBP1|en|ZBP1}}および{{Ill2|真核生物リボソーム小サブユニット|en|Eukaryotic small ribosomal subunit (40S)|label=40Sサブユニット}}と結合する。この複合体は[[モータータンパク質]]によって結合され、[[細胞骨格]]に沿って目的位置([[神経突起|神経突起伸長部]])に輸送される。最終的に、ZBP1が{{Ill2|Srcファミリーキナーゼ|en|Src family kinase|label=Src}}によって[[リン酸化]]され、翻訳が開始される<ref name="Hüttelmaier_2005">{{cite journal | vauthors = Hüttelmaier S, Zenklusen D, Lederer M, Dictenberg J, Lorenz M, Meng X, Bassell GJ, Condeelis J, Singer RH | display-authors = 6 | title = Spatial regulation of beta-actin translation by Src-dependent phosphorylation of ZBP1 | journal = Nature | volume = 438 | issue = 7067 | pages = 512–515 | date = November 2005 | pmid = 16306994 | doi = 10.1038/nature04115 | s2cid = 2453397 | bibcode = 2005Natur.438..512H }}</ref>。発達中の神経細胞では、mRNAは成長中の[[軸索]]、特に[[成長円錐]]にも輸送される。多くのmRNAには、特定の場所に輸送するために、いわゆる「ジップコード(郵便番号の意)」が付与されている<ref name="Ainger1997">{{cite journal | vauthors = Ainger K, Avossa D, Diana AS, Barry C, Barbarese E, Carson JH | title = Transport and localization elements in myelin basic protein mRNA | journal = The Journal of Cell Biology | volume = 138 | issue = 5 | pages = 1077–1087 | date = September 1997 | pmid = 9281585 | pmc = 2136761 | doi = 10.1083/jcb.138.5.1077 }}</ref>。mRNAは、[[細胞膜ナノチューブ]](トンネルナノチューブ)と呼ばれる構造体を通じて、[[哺乳動物]]細胞間でも移動することができる<ref>{{cite journal | vauthors = Haimovich G, Ecker CM, Dunagin MC, Eggan E, Raj A, Gerst JE, Singer RH | title = Intercellular mRNA trafficking via membrane nanotube-like extensions in mammalian cells | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 114 | issue = 46 | pages = E9873–E9882 | date = November 2017 | pmid = 29078295 | pmc = 5699038 | doi = 10.1073/pnas.1706365114 | doi-access = free }}</ref><ref>{{Cite journal | vauthors = Haimovich G, Dasgupta S, Gerst JE |title=RNA transfer through tunneling nanotubes | journal = Biochemical Society Transactions | date = February 2021| volume = 49 | issue = 1 | pages = 145–160 |url=https://portlandpress.com/biochemsoctrans/article-abstract/49/1/145/227426/RNA-transfer-through-tunneling-nanotubes |doi=10.1042/BST20200113|pmid=33367488 |s2cid=229689880 }}</ref>。 |
|||
=== 翻訳 === |
|||
{{main|翻訳 (生物学)|真核生物の翻訳}} |
|||
[[File:Peptide syn.svg|thumb|mRNAからタンパク質への翻訳を示す模式図。[[リボソーム]] (緑)は、一連の'''伝令RNA''' (Messanger RNA) (黄緑)と、[[転移RNA]] (TRNA) (黄) に結びついた[[アミノ酸]] (赤丸) から所定の[[タンパク質]] (赤丸の連鎖) を組み立てる。]] |
|||
原核生物のmRNAは、プロセシングや輸送を必要としないため、転写終了後すぐに[[リボソーム]]により翻訳を開始することができる。したがって、原核生物における翻訳は転写と共役しており、共転写的に行われていると言える。 |
|||
真核生物のmRNAは、プロセシングされて細胞質に輸送された後(すなわち成熟mRNA)、リボソームによって翻訳することができる。翻訳は、細胞質内を自由に浮遊しているリボソームで起こる場合と、[[シグナル認識粒子]]によって誘導されて、[[小胞体]]に結合したリボソームで起こる場合がある。したがって、原核生物とは異なり、真核生物における翻訳は転写と直接的に結びついていない。[[乳癌]](がん)で監視される{{Ill2|真核生物翻訳伸長因子1α1|en|Eukaryotic translation elongation factor 1 alpha 1|label=EEF1A1}}のmRNA/タンパク質レベルのように、mRNAレベルの低下がタンパク質レベルの上昇を伴うこともある<ref name="Lin2018">{{cite journal | vauthors = Lin CY, Beattie A, Baradaran B, Dray E, Duijf PH | title = Contradictory mRNA and protein misexpression of EEF1A1 in ductal breast carcinoma due to cell cycle regulation and cellular stress | journal = Scientific Reports | volume = 8 | issue = 1 | pages = 13904 | date = September 2018 | pmid = 30224719 | pmc = 6141510 | doi = 10.1038/s41598-018-32272-x | bibcode = 2018NatSR...813904L }}</ref>{{Primary source inline|date=June 2021}}。 |
|||
== {{Anchors|Structure}}構造 == |
|||
[[File:MRNA structure.svg|thumb|700px|center|成熟した真核生物のmRNAの構造。完全にプロセシングされたmRNAは、(左から右へ) [[5'キャップ]]、[[5' UTR]]、[[コーディング領域]]、[[3' UTR]]、および[[ポリ(A)テール]]から構成される。]] |
|||
=== コーディング領域 === |
|||
{{main|コーディング領域}} |
|||
[[コーディング領域]](''coding regions'')は[[コドン]](遺伝暗号ともいう)で構成され、リボソームによって解読され、さらにタンパク質へ翻訳される。これは、真核生物では通常1つなのに対し、原核生物では通常複数である。コーディング領域は、[[開始コドン]]で始まり、[[終止コドン]]で終わる。一般に、開始コドンはAUGトリプレットで、終止コドンはUAG(アンバー)、UAA(オーカー)、またはUGA(オパール)である。コーディング領域は内部の塩基対によって安定化する傾向があり、これが分解を妨げている<ref>{{cite journal | vauthors = Shabalina SA, Ogurtsov AY, Spiridonov NA | title = A periodic pattern of mRNA secondary structure created by the genetic code | journal = Nucleic Acids Research | volume = 34 | issue = 8 | pages = 2428–2437 | year = 2006 | pmid = 16682450 | pmc = 1458515 | doi = 10.1093/nar/gkl287 }}</ref><ref>{{cite journal | vauthors = Katz L, Burge CB | title = Widespread selection for local RNA secondary structure in coding regions of bacterial genes | journal = Genome Research | volume = 13 | issue = 9 | pages = 2042–2051 | date = September 2003 | pmid = 12952875 | pmc = 403678 | doi = 10.1101/gr.1257503 }}</ref>。コーディング領域は、タンパク質を[[遺伝暗号|コードする]]ことに加え、その一部は{{Ill2|エクソン性スプライシングエンハンサー|en|Exonic splicing enhancer}}または{{Ill2|エクソン性スプライシングサイレンサー|en|Exonic splicing silencer}}として、[[pre-mRNA]]中の{{Ill2|制御配列|en|Regulatory sequence}}として機能することがある。 |
|||
=== 非翻訳領域 === |
|||
{{main|非翻訳領域|エクソン}} |
|||
[[File:Fbioe-09-718753-g002.jpg|thumb|真核生物mRNAにおける5' 非翻訳領域 (5' UTR) および3' 非翻訳領域 (3' UTR) の一般的な構造を示す。]] |
|||
[[非翻訳領域]](''untranslated regions''、UTR)は、mRNAのうち、開始コドンの前および停止コドンの後で翻訳されない領域のことで、それぞれ[[5' 非翻訳領域]](5' UTR)と[[3' 非翻訳領域]](3' UTR)と呼ばれる。これらの領域はコーディング領域と一緒に転写されるため、成熟mRNA中にそのまま存在することから[[エクソン|エクソン性]](''exonic'')という。[[遺伝子発現]]に関わる非翻訳領域のいくつかの役割は、mRNAの安定性、mRNAの局在化、{{Ill2|翻訳効率|en|Translational efficiency|label=翻訳効率}}へ起因するとされている。UTRがこれらの機能を果たすかどうかはUTRの配列に依存し、mRNAの種類によって異なる可能性がある。また、3' UTRの遺伝子変異は、RNAの構造やタンパク質への翻訳を変化させるため、疾患感受性にも関与すると考えられている<ref>{{cite journal | vauthors = Lu YF, Mauger DM, Goldstein DB, Urban TJ, Weeks KM, Bradrick SS | title = IFNL3 mRNA structure is remodeled by a functional non-coding polymorphism associated with hepatitis C virus clearance | journal = Scientific Reports | volume = 5 | pages = 16037 | date = November 2015 | pmid = 26531896 | pmc = 4631997 | doi = 10.1038/srep16037 | bibcode = 2015NatSR...516037L }}</ref>。 |
|||
mRNAの安定性は、[[リボヌクレアーゼ]]というRNA分解酵素や、RNA分解を促進または阻害する補助タンパク質に対する親和性が異なるため、5' UTRおよび(または)3' UTRによって制御されている可能性がある ({{ill2|Cリッチ安定化配列|en|C-rich stability element}}も参照)。 |
|||
翻訳効率は、時には翻訳を完全に阻害することも含め、UTRによって制御することができる。3' UTRまたは5' UTRに結合するタンパク質は、リボソームがmRNAに結合する能力に働きかけることで、翻訳に影響を及ぼす可能性がある。また、3' UTRに結合した[[マイクロRNA]](miRNA)も、翻訳効率やmRNAの安定性に影響を及ぼす可能性がある。 |
|||
mRNAの細胞質局在性は、3' UTRの機能であると考えられている。細胞内の特定の領域で必要とされるタンパク質は、その場所で翻訳されることもある。このような場合、3' UTRには、転写産物を翻訳するためにこの領域に局在化させる配列が含まれている可能性がある。 |
|||
非翻訳領域に含まれる配列<!-- elements -->の中には、RNAに転写されると特徴的な[[二次構造]]を形成するものがある。これらの構造的なmRNA配列は、mRNAの調節に関与している。{{Ill2|SECIS配列|en|SECIS element}}のようにタンパク質が結合する標的となるものもある。mRNA配列の一種である[[リボスイッチ]]は、[[小分子]]と直接結合してその折りたたみを変化させて転写や翻訳のレベルを変更する。こうした場合、mRNAはそれ自身を制御している。 |
|||
=== {{Anchors|Poly(A) tail}}ポリ(A)テール === |
|||
{{main|ポリアデニル化}} |
|||
3'ポリ(A)テール(''3' poly(A) tail'')は、pre-mRNAの3'末端に付加された[[アデニン]]ヌクレオチドの長い配列である(配列長は数100個が多い)。このテール(尾部)は、細胞核からの輸送と翻訳を促進するとともに、mRNAを分解から保護する役割を持つ。 |
|||
=== モノシストロン型とポリシストロン型の違い === |
|||
{{see also|{{ill2|シストロン (遺伝学)|en|Cistron}}}} |
|||
mRNA分子が、単一の[[タンパク質構造|タンパク質鎖]](ポリペプチド)のみを[[翻訳 (生物学)|翻訳]]するための遺伝情報を含む場合、モノシストロン型(''monocistronic mRNA'')であるという。ほとんどの[[真核生物]]のmRNAはこのようなケースである<ref name="Kozak_1983">{{cite journal | vauthors = Kozak M | title = Comparison of initiation of protein synthesis in procaryotes, eucaryotes, and organelles | journal = Microbiological Reviews | volume = 47 | issue = 1 | pages = 1–45 | date = March 1983 | pmid = 6343825 | pmc = 281560 | doi = 10.1128/MMBR.47.1.1-45.1983}}</ref><ref>{{cite journal | vauthors = Niehrs C, Pollet N | title = Synexpression groups in eukaryotes | journal = Nature | volume = 402 | issue = 6761 | pages = 483–487 | date = December 1999 | pmid = 10591207 | doi = 10.1038/990025 | bibcode = 1999Natur.402..483N | s2cid = 4349134 }}</ref>。一方、ポリシストロン型(''polycistronic mRNA'')mRNAは、複数の[[オープンリーディングフレーム|オープン・リーディング・フレーム]](ORF)を持ち、それぞれがポリペプチドに翻訳される。これらのポリペプチドは通常、関連する機能を持ち(多くは最終的な複合タンパク質を構成するサブユニット)、それらのコード配列(''coding sequence''、CDS)は[[プロモーター]]と[[オペレーター (生物学)|オペレーター]]を含む制御領域にまとめられて全体として制御される。[[細菌]]や[[古細菌]]に見られるmRNAのほとんどはポリシストロン型で、ヒトの[[ミトコンドリア]]・[[ゲノム]]も同様である<ref name="Kozak_1983" />。ジシストロン型(''dicistronic'')またはバイシストロン型(''bicistronic'')のmRNAは、2つの[[タンパク質]]のみをコードしている<ref name="MercerNeph2011">{{cite journal | vauthors = Mercer TR, Neph S, Dinger ME, Crawford J, Smith MA, Shearwood AM, Haugen E, Bracken CP, Rackham O, Stamatoyannopoulos JA, Filipovska A, Mattick JS |author-link10=John Stamatoyannopoulos | title = The human mitochondrial transcriptome | journal = Cell | volume = 146 | issue = 4 | pages = 645–658 | date = August 2011 | pmid = 21854988 | pmc = 3160626 | doi = 10.1016/j.cell.2011.06.051 }}</ref>。 |
|||
=== mRNAの環状化 === |
|||
[[File:Fgene-10-00006-g001.jpg|thumb|mRNAの環状化と調節。(詳細は[[:ファイル:Fgene-10-00006-g001.jpg|画像の概要]]を参照)]] |
|||
真核生物では、[[eIF4E]]と[[ポリ(A)結合タンパク質]](PABP)が相互作用し、両者が足場タンパク質の{{Ill2|eIF4G|en|eIF4G}}に結合してmRNA-タンパク質-mRNAの橋渡しをすることで、mRNA分子は環状構造を形成する<ref>{{cite journal | vauthors = Wells SE, Hillner PE, Vale RD, Sachs AB | title = Circularization of mRNA by eukaryotic translation initiation factors | journal = Molecular Cell | volume = 2 | issue = 1 | pages = 135–140 | date = July 1998 | pmid = 9702200 | doi = 10.1016/S1097-2765(00)80122-7 | citeseerx = 10.1.1.320.5704 }}</ref>。環状化は、mRNA上のリボソームの循環を促進し、時間効率のよい翻訳をもたらすと考えられており、また、無傷のmRNAのみを翻訳するように機能する可能性もある(部分的に分解したmRNAは、m<sup>7</sup>Gキャップやポリ(A)テールの欠失を特徴とする)<ref>{{cite journal | vauthors = López-Lastra M, Rivas A, Barría MI | title = Protein synthesis in eukaryotes: the growing biological relevance of cap-independent translation initiation | journal = Biological Research | volume = 38 | issue = 2–3 | pages = 121–146 | year = 2005 | pmid = 16238092 | doi = 10.4067/S0716-97602005000200003 | doi-access = free }}</ref>。 |
|||
この他に、特にウイルスmRNAで、環状化の機構が知られている。[[ポリオウイルス]]のmRNAは、その5'末端方向のクローバーリーフ部分を利用してヒトタンパク質{{Ill2|PCBP2|en|PCBP2}}と結合し、PCBP2はポリ(A)結合タンパク質と結合して、よく知られたmRNA-タンパク質-mRNAの輪を形成する。{{Ill2|オオムギ黄化萎縮病|en|Barley yellow dwarf|label=オオムギ黄化萎縮ウイルス}}は、5'末端と3'末端のmRNAセグメント間で結合し({{Ill2|キッシングステムループ|en|Kissing stem-loop}}と呼ばれる)、タンパク質を介さずにmRNAを環状化する。 |
|||
RNAウイルスゲノム(その+鎖がmRNAとして翻訳される)も一般に環状化している{{Citation needed|date=October 2012}}。ゲノム複製の際、環状化はゲノム複製速度を高めるように作用し、リボソームが循環している仮説とほぼ同様に、ウイルス[[RNA依存性RNAポリメラーゼ]]を循環させる。 |
|||
== {{Anchors|Degradation}}分解 == |
|||
同じ細胞内でも、mRNAの寿命(安定性)はそれぞれ異なる。[[細菌]]細胞では、個々のmRNAは数秒から1時間以上生存することができる。しかしその寿命は平均して1-3分であり、細菌のmRNAは真核生物のmRNAよりもはるかに安定性が低い<ref>{{Cite book|title=Lewin's genes X|date=2011|publisher=Jones and Bartlett| veditors = Lewin B, Krebs JE, Kilpatrick ST, Goldstein ES |editor-link1=Benjamin Lewin |isbn=9780763766320|edition=10th|location=Sudbury, Mass.|oclc=456641931|url-access=registration|url=https://archive.org/details/lewinsgenesx0000unse}}</ref>。哺乳動物細胞では、mRNAの寿命は数分から数日にまで及ぶ<ref>{{cite journal | vauthors = Yu J, Russell JE | title = Structural and functional analysis of an mRNP complex that mediates the high stability of human beta-globin mRNA | journal = Molecular and Cellular Biology | volume = 21 | issue = 17 | pages = 5879–5888 | date = September 2001 | pmid = 11486027 | pmc = 87307 | doi = 10.1128/mcb.21.17.5879-5888.2001 }}</ref>。mRNAの安定性が高いほどそのmRNAからより多くのタンパク質が生成される可能性がある。mRNAの寿命が限られているので、細胞は変化する需要に応じてタンパク質合成を速やかに変更することができる。mRNAの破壊をもたらす多くの機構があり、そのいくつかを次に説明する。 |
|||
=== 原核生物のmRNA分解 === |
|||
[[File:1-s2.0-S1874939913000436-gr1 lrg.jpg|thumb|さまざまな生命ドメインにおけるRNA分解経路の概要。グラム陰性の大腸菌とグラム陽性の枯草菌 (それぞれ左上と右上) に代表される2つの主要な細菌系統では、経路は類似しているが、酵素は異なっている。グラム陰性とグラム陽性の両方で、エンドリボヌクレアーゼは基質を繰り返し切断することができ、生成物はエキソリボヌクレアーゼによって攻撃される。比較のため、真核生物の分解経路を示す (右上)。]] |
|||
一般的に、原核生物では、真核生物よりもmRNAの寿命がはるかに短い。原核生物は、[[エンドヌクレアーゼ]]、3'[[エキソヌクレアーゼ]]、および5'エキソヌクレアーゼを含むリボヌクレアーゼの組み合わせて、メッセージ(mRNAの意)を分解する。また、数十から数百ヌクレオチド長の{{Ill2|小型RNA|en|Small RNA}}(sRNA)が相補的な配列と塩基対を形成し、{{Ill2|リボヌクレアーゼIII|en|Ribonuclease III|label=RNase III}}によるリボヌクレアーゼ切断を促進することによって、特定のmRNAの分解を促す場合がある。最近、細菌も5'末端に三リン酸からなる一種の5'キャップを持っていることが明らかになった<ref name="Deana2008">{{cite journal | vauthors = Deana A, Celesnik H, Belasco JG | title = The bacterial enzyme RppH triggers messenger RNA degradation by 5' pyrophosphate removal | journal = Nature | volume = 451 | issue = 7176 | pages = 355–358 | date = January 2008 | pmid = 18202662 | doi = 10.1038/nature06475 | bibcode = 2008Natur.451..355D | s2cid = 4321451 }}</ref>。このリン酸を2つ除去すると5'-リン酸が残り、5'を3'に分解するエキソヌクレアーゼRNase Jによってメッセージが破壊される。 |
|||
=== 真核生物のmRNAターンオーバー === |
|||
真核細胞内では、[[翻訳 (生物学)|翻訳]]とmRNA分解のプロセス間で[[ターンオーバー (生物)|釣り合いが保たれている]]。活発に翻訳されているメッセージは、[[リボソーム]]、{{Ill2|真核生物翻訳開始因子|en|Eukaryotic initiation factor}}[[eIF4E]]および{{Ill2|EIF4G|en|EIF4G|label=eIF4G}}、[[ポリ(A)結合タンパク質]]によって結合されている。eIF4EとeIF4Gはデキャッピング酵素({{Ill2|DCP2|en|DCP2}})を阻害し、ポリ(A)結合タンパク質は[[エキソソーム複合体]]を阻害して、メッセージの末端を保護する。翻訳と分解の釣り合いは、{{Ill2|Pボディ|en|P-bodies}}(''P-bodies'')という細胞質構造の大きさと存在量に反映される<ref name="Parker2007">{{cite journal | vauthors = Parker R, Sheth U | title = P bodies and the control of mRNA translation and degradation | journal = Molecular Cell | volume = 25 | issue = 5 | pages = 635–646 | date = March 2007 | pmid = 17349952 | doi = 10.1016/j.molcel.2007.02.011 | doi-access = free }}</ref>。mRNAの[[ポリアデニル化|ポリ(A)テール]]は、RNA上のシス制御配列とトランス作用性RNA結合タンパク質の組み合わせによって、特定のメッセンジャーRNAを標的とする特殊なエキソヌクレアーゼによって短縮される。ポリ(A)テールの除去は、メッセージの環状構造を破壊し、{{Ill2|キャップ結合複合体|en|Cap binding complex}}を不安定化すると考えられている。その後、メッセージはエキソソーム複合体または{{Ill2|デキャッピング複合体|en|Decapping complex}}のいずれかによって分解される。このようにして、翻訳的に不活性なメッセージを速やかに破壊し、活性なメッセージを無傷のまま残すことができる。翻訳を停止してメッセージが崩壊複合体に渡される機構は詳しくは分かっていない。 |
|||
=== AUリッチエレメント分解 === |
|||
一部の哺乳類では、mRNA中に{{Ill2|AUリッチエレメント|en|AU-rich element}}(ARE)が存在すると、この配列に結合して[[ポリ(A)テール]]の除去を促す細胞タンパク質の作用によって、これらの転写産物を不安定化する傾向がある。ポリ(A)テールの欠失は、[[エキソソーム複合体]]<ref name="Chen2001">{{cite journal | vauthors = Chen CY, Gherzi R, Ong SE, Chan EL, Raijmakers R, Pruijn GJ, Stoecklin G, Moroni C, Mann M, Karin M | title = AU binding proteins recruit the exosome to degrade ARE-containing mRNAs | journal = Cell | volume = 107 | issue = 4 | pages = 451–464 | date = November 2001 | pmid = 11719186 | doi = 10.1016/S0092-8674(01)00578-5 | s2cid = 14817671 | doi-access = free }}</ref>と{{Ill2|デキャッピング複合体|en|Decapping complex}}<ref>{{cite journal | vauthors = Fenger-Grøn M, Fillman C, Norrild B, Lykke-Andersen J | title = Multiple processing body factors and the ARE binding protein TTP activate mRNA decapping | journal = Molecular Cell | volume = 20 | issue = 6 | pages = 905–915 | date = December 2005 | pmid = 16364915 | doi = 10.1016/j.molcel.2005.10.031 | doi-access = free }}</ref>の両方による攻撃を促進することにより、mRNAの分解を促進すると考えられている。AUリッチエレメントを介した速やかなmRNA分解は、[[腫瘍壊死因子]](TNF)や[[顆粒球マクロファージコロニー刺激因子]](GM-CSF)のような強力な[[サイトカイン]]の過剰産生を防ぐための重要な機構である<ref name="Shaw1986">{{cite journal | vauthors = Shaw G, Kamen R | title = A conserved AU sequence from the 3' untranslated region of GM-CSF mRNA mediates selective mRNA degradation | journal = Cell | volume = 46 | issue = 5 | pages = 659–667 | date = August 1986 | pmid = 3488815 | doi = 10.1016/0092-8674(86)90341-7 | s2cid = 40332253 }}</ref>。また、AUリッチエレメントは、{{Ill2|Transcription factor Jun|en|Transcription factor Jun|label=c-Jun}}や[[c-Fos]]などの発がん性転写因子の生合成も調節する<ref name="Chen1995">{{cite journal | vauthors = Chen CY, Shyu AB | title = AU-rich elements: characterization and importance in mRNA degradation | journal = Trends in Biochemical Sciences | volume = 20 | issue = 11 | pages = 465–470 | date = November 1995 | pmid = 8578590 | doi = 10.1016/S0968-0004(00)89102-1 }}</ref>。 |
|||
=== ナンセンス変異依存mRNA分解機構 === |
|||
{{main|ナンセンス変異依存mRNA分解機構}} |
|||
真核生物のメッセージは、メッセージ中の中途での終止コドン([[ナンセンスコドン]])の存在をチェックする[[ナンセンス変異依存mRNA分解機構]](NMD)による監視を受けている。ナンセンスコドンは、不完全なスプライシング、[[適応免疫系]]における[[V(D)J遺伝子再構成]]、DNAの[[遺伝的変異|変異]]、転写エラー、{{Ill2|リボソームフレームシフト|en|Ribosomal frameshift|label=フレームシフト}}を引き起こすリボソームによる{{Ill2|漏出スキャン|en|Leaky scanning}}、およびその他の原因によって発生する可能性がある。中途での終止コドンが検出されると、5'キャップ除去、3'[[ポリ(A)]]テール除去、または[[エンドヌクレアーゼ|ヌクレオチド鎖切断]]による分解を引き起こす<ref name="Isken2007">{{cite journal | vauthors = Isken O, Maquat LE | title = Quality control of eukaryotic mRNA: safeguarding cells from abnormal mRNA function | journal = Genes & Development | volume = 21 | issue = 15 | pages = 1833–1856 | date = August 2007 | pmid = 17671086 | doi = 10.1101/gad.1566807 | doi-access = free }}</ref>。 |
|||
=== 低分子干渉RNA (siRNA) === |
|||
{{main|siRNA}} |
|||
[[動物|後生動物]]では、酵素である[[Dicer]]によって処理された[[低分子干渉RNA]](siRNA)は、[[RNA誘導サイレンシング複合体]]またはRISC(RNA-induced silencing complex)として知られる複合体に取り込まれる。この複合は[[エンドヌクレアーゼ]]を含んでおり、siRNAが結合する完全に相補的なメッセージを切断する。その結果として生じたmRNA断片は、[[エキソヌクレアーゼ]]によって破壊される。siRNAは、細胞培養において遺伝子の機能を阻害するために、実験室で一般的に使用されている。これは[[二本鎖RNAウイルス]]に対する防御としての[[自然免疫系]]の一部であると考えられている<ref name="Obbard2009">{{cite journal | vauthors = Obbard DJ, Gordon KH, Buck AH, Jiggins FM | title = The evolution of RNAi as a defence against viruses and transposable elements | journal = Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences | volume = 364 | issue = 1513 | pages = 99–115 | date = January 2009 | pmid = 18926973 | pmc = 2592633 | doi = 10.1098/rstb.2008.0168 }}</ref>。 |
|||
=== マイクロRNA (miRNA) === |
|||
{{main|マイクロRNA}} |
|||
[[マイクロRNA]](miRNA)は、通常、後生動物のメッセンジャーRNAと部分相補的な配列を持つ小型RNAである<ref>Robert E. Farrell, Jr. RNA Methodologies, 5th Edition. Academic Press, 2017</ref><ref>{{cite journal | vauthors = Brennecke J, Stark A, Russell RB, Cohen SM | title = Principles of microRNA-target recognition | journal = PLOS Biology | volume = 3 | issue = 3 | pages = e85 | date = March 2005 | pmid = 15723116 | pmc = 1043860 | doi = 10.1371/journal.pbio.0030085 }}</ref>。miRNAがメッセージに結合すると、そのメッセージの翻訳は抑制されまたポリ(A)テールの除去が促進されるため、mRNAの分解は早められる。miRNAの作用機序は活発な研究対象となっている<ref>Tasuku Honjo, Michael Reth, Andreas Radbruch, Frederick Alt. Molecular Biology of B Cells, 2nd Edition. Academic Press, 2014 (including "updated research on microRNAs")</ref><ref name="Eulalio2009">{{cite journal | vauthors = Eulalio A, Huntzinger E, Nishihara T, Rehwinkel J, Fauser M, Izaurralde E | title = Deadenylation is a widespread effect of miRNA regulation | journal = RNA | volume = 15 | issue = 1 | pages = 21–32 | date = January 2009 | pmid = 19029310 | pmc = 2612776 | doi = 10.1261/rna.1399509 }}</ref>。 |
|||
=== その他の分解機構 === |
|||
メッセージが分解される機構は他にも、{{Ill2|ノンストップ分解|en|Non-stop decay}}(''non-stop decay''、NSD)や、{{Ill2|Piwi結合RNA|en|Piwi-interacting RNA}}(''Piwi-interacting RNA''、piRNA)による[[サイレンシング]]など、さまざまなものがある。 |
|||
== {{Anchors|Applications}}応用例 == |
|||
{{see also|RNAワクチン|RNA治療}} |
|||
[[ヌクレオシド修飾メッセンジャーRNA]](modRNA)配列を投与することで、細胞にタンパク質を作らせることができ、直接的にはそのタンパク質が病気を治療したり、[[ワクチン]]として機能する可能性がある。より間接的には、このタンパク質が[[内在性]][[幹細胞]]を望ましい方法で分化させる可能性がある<ref name="NatRevMat">{{cite journal| vauthors = Hajj KA, Whitehead KA |title=Tools for translation: non-viral materials for therapeutic mRNA delivery|journal=Nature Reviews Materials|date=12 September 2017|volume=2|issue=10|pages=17056|doi=10.1038/natrevmats.2017.56|bibcode=2017NatRM...217056H|doi-access=free}}</ref><ref name="GEN">{{cite news| vauthors = Gousseinov E, Kozlov M, Scanlan C |title=RNA-Based Therapeutics and Vaccines|url=https://www.genengnews.com/gen-exclusives/rna-based-therapeutics-and-vaccines/77900520|work=Genetic Engineering News|date=September 15, 2015}}</ref>。 |
|||
RNA治療の主な課題は、RNAを適切な細胞に送達することにある<ref name="genemed">{{cite journal|vauthors=Kaczmarek JC, Kowalski PS, Anderson DG|date=June 2017|title=Advances in the delivery of RNA therapeutics: from concept to clinical reality|journal=Genome Medicine|volume=9|issue=1|pages=60|doi=10.1186/s13073-017-0450-0|pmc=5485616|pmid=28655327}}</ref>。課題にはさらに、裸のRNA配列が調剤後に自然に分解されること、身体の[[免疫系]]がRNAを侵入者として攻撃する可能性があること、細胞膜を[[半透膜|通過]]しないことといった事実も含まれる<ref name="GEN" />。RNAが細胞内に入った後、必要な[[リボソーム]]がある[[細胞質]]で活動するためには、細胞の輸送機構を離れなくてはならない<ref name="NatRevMat" />。 |
|||
これらの課題を克服し、1989年に『広く適用可能な''[[in vitro]]''[[トランスフェクション|トランスフェクション技術]]が開発された後』<ref>{{cite journal | vauthors = Schlake T, Thess A, Fotin-Mleczek M, Kallen KJ | title = Developing mRNA-vaccine technologies | journal = RNA Biology | volume = 9 | issue = 11 | pages = 1319–30 | date = November 2012 | pmid = 23064118 | pmc = 3597572 | doi = 10.4161/rna.22269 }}</ref>、治療薬としてのmRNAが初めて提唱された。1990年代に、非ヌクレオシド修飾mRNA<!-- non-nucleoside modified mRNA -->に依存した、{{Ill2|個別化がん免疫療法|en|Individualized cancer immunotherapy|label=個別化がん}}に対するmRNAワクチンが開発された。mRNAを用いた治療法は、がんだけでなく、[[自己免疫疾患]]、[[代謝疾患|代謝性疾患]]、および呼吸器炎症性疾患に対する治療法と処置法の両面で研究が続けられている。{{Ill2|CRISPR遺伝子編集|en|CRISPR gene editing|label=CRISPR}}のような[[遺伝子治療|遺伝子編集療法]]も、目的の[[Cas9|Cas]]タンパク質を作るよう細胞を誘導するためにmRNAを使用することで、有益となる可能性がある<ref>{{Cite web| vauthors = Haridi R |date=2021-04-23|title=The mRNA revolution: How COVID-19 hit fast-forward on an experimental technology|url=https://newatlas.com/science/mrna-revolution-vaccine-covid-therapy-pandemic-future-cancer/|url-status=live|access-date=2021-04-26|website=New Atlas|language=en-US}}</ref>。 |
|||
2010年代以降、RNAワクチンやその他のRNA治療薬は「新しいクラスの医薬品」と見なされている<ref>{{Citation|title=mRNA-based therapeutics–developing a new class of drugs.|date=2014|work=[[:en:Nature Reviews Drug Discovery|Nature Reviews Drug Discovery]]|volume=13|issue=10|pages=759–780|language=en|pmid=25150148|vauthors=Kowalska J, Wypijewska del Nogal A, Darzynkiewicz ZM, Buck J, Nicola C, Kuhn AN, Lukaszewicz M, Zuberek J, Strenkowska M, Ziemniak M, Maciejczyk M, Bojarska E, Rhoads RE, Darzynkiewicz E, Sahin U, Jemielity J |doi=10.1093/nar/gku757 |doi-access=free |pmc=4176373}}</ref>。最初のmRNAに基づくワクチンは制限付き承認を受け、[[COVID-19パンデミック]]の間に、たとえば[[ファイザー - バイオンテック COVID-19 ワクチン|ファイザー - バイオンテック]]や[[モデルナCOVID-19ワクチン|モデルナ]]による[[COVID-19ワクチン]]が世界中で展開された<ref name="pmid35534554">{{cite journal | vauthors = Barbier AJ, Jiang AY, Zhang P, Wooster R, Anderson DG | title = The clinical progress of mRNA vaccines and immunotherapies | journal = Nature Biotechnology | volume = 40 | issue = 6 | pages = 840–854 | date = June 2022 | pmid = 35534554 | doi = 10.1038/s41587-022-01294-2 | s2cid = 248667843 }}</ref>。 |
|||
== {{Anchors|History}}歴史 == |
|||
1950年代初頭から、分子生物学の研究によって、タンパク質合成の際にRNAに関連する分子が存在することが示唆された。たとえば、最も古い報告の1つで、[[ジャック・モノー]]と彼のチームは、RNA合成がタンパク質合成に必要であることを示し、特に細菌の[[大腸菌]]で酵素である[[Β-ガラクトシダーゼ|βガラクトシダーゼ]]を産生する時に必要なことを示した<ref>{{Cite journal | vauthors = Monod J, Pappenheimer AM, Cohen-Bazire G |date=1952 |title=La cinétique de la biosynthèse de la β-galactosidase chez E. coli considérée comme fonction de la croissance |journal=Biochimica et Biophysica Acta |language=fr |volume=9 |issue=6 |pages=648–660 |doi=10.1016/0006-3002(52)90227-8|pmid=13032175 }}</ref>。また、1954年に{{Ill2|アーサー・パーディー|en|Arthur Pardee}}も同様のRNA蓄積を発見した<ref>{{cite journal | vauthors = Pardee AB | title = Nucleic Acid Precursors and Protein Synthesis | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 40 | issue = 5 | pages = 263–270 | date = May 1954 | pmid = 16589470 | pmc = 534118 | doi = 10.1073/pnas.40.5.263 | bibcode = 1954PNAS...40..263P | doi-access = free }}</ref>。1953年、[[アルフレッド・ハーシー]]、[[ジューン・ディクソン]]<!-- June Dixon -->、[[マーサ・チェイス]]は、大腸菌内で合成後すぐに消失する特定のシトシン含有DNA(RNAであることを示す)について報告した<ref>{{cite journal | vauthors = Hershey AD, Dixon J, Chase M | title = Nucleic acid economy in bacteria infected with bacteriophage T2. I. Purine and pyrimidine composition | journal = The Journal of General Physiology | volume = 36 | issue = 6 | pages = 777–789 | date = July 1953 | pmid = 13069681 | pmc = 2147416 | doi = 10.1085/jgp.36.6.777 }}</ref>。これは、mRNAの存在を示す最初の記録であったが、mRNAとしては特定されなかった<ref name="Cobb" />。 |
|||
mRNAのアイディアは、1960年4月15日、ケンブリッジの[[キングス・カレッジ (ケンブリッジ大学)|キングス・カレッジ]]で、[[シドニー・ブレナー]]とフランシス・クリックによって最初に着想され、[[フランソワ・ジャコブ]]が、アーサー・パーディー、ジャコブ、そしてモノーが最近行った実験について話しをしているときだった。クリックの励ましを受け、ブレナーとジャコブはすぐにこの新しい仮説の検証に着手し、[[カリフォルニア工科大学]]の[[マシュー・メセルソン]]に連絡を取った。1960年の夏、ブレナー、ジャコブ、メセルソンの3人は、カリフォルニア工科大学のメセルソンの研究室で実験を行い、mRNAの存在を証明した。その年の秋に、ジャコブとモノーは「''メッセンジャーRNA''(''messenger RNA'')」と命名し、その機能を説明する最初の理論的枠組みを構築した<ref name="Cobb">{{cite journal |author-link1=Matthew Cobb |vauthors=Cobb M |date=29 June 2015 |title=Who discovered messenger RNA? |journal=Current Biology |volume=25 |issue=13 |pages=R526–R532 |doi=10.1016/j.cub.2015.05.032 |pmid=26126273 |doi-access=free}}</ref>。 |
|||
1961年2月、[[ジェームズ・ワトソン]]は、自身の研究グループが彼らのすぐ後を、ほぼ同じ方向で同様の実験を行っていることを明らかにした。ブレナーと他の人たちは、彼らの研究知見の論文発表を遅らせるというワトソンからの要請に同意した。その結果、1961年5月の『[[ネイチャー|''ネイチャー'']]』誌にブレナーとワトソンの論文が同時に掲載され、同じ月の『''{{Ill2|ジャーナル・オブ・モレキュラー・バイオロジー|en|Journal of Molecular Biology}}(Journal of Molecular Biology)''』誌にジャコブとモノーはmRNAの理論的枠組みを発表した<ref>F. Jacob, J. Monod "Genetic regulatory mechanisms in the synthesis of proteins". Journal of Molecular Biology, 3 (1961), pp. 318-356.</ref><ref name="Cobb" />。 |
|||
== 関連項目 == |
== 関連項目 == |
||
{{Commonscat|mRNA}} |
{{Commonscat|mRNA}} |
||
* [[リボ核酸]](RNA) |
* [[リボ核酸]](RNA) |
||
* [[転写減衰]] |
|||
* [[イントロン]] |
|||
* [[エクソン]] |
|||
* [[スプライシング]] |
|||
* [[pre-mRNA スプライシング]] |
|||
* [[RNAエディティング]] |
|||
* [[H-Invitational Database]] |
|||
* [[COVID-19ワクチン]] |
|||
* [[タンパク質生合成]] - 細胞内でタンパク質を生成する過程 |
|||
==外部リンク== |
|||
* [[転写 (生物学)]] - 細胞で遺伝情報がmRNAに転写されてタンパク質を合成する過程 |
|||
* [[翻訳 (生物学)]] - DNAの断片をRNAにコピーする過程 |
|||
* [[リボソーム]] - 細胞内で生体タンパク質の合成(mRNAの翻訳)を行う高分子 |
|||
* {{Ill2|ミスセンスmRNA|en|Missense mRNA}} - 変異したコドンを持つメッセンジャーRNA |
|||
* [[ノンコーディングRNA]] - タンパク質へ翻訳されないRNA分子 |
|||
* {{Ill2|mRNA監視機構|en|mRNA surveillance}} - 生物がmRNA分子の忠実度と品質を確保するための仕組み |
|||
* [[トランスクリプトーム]] - 個体または細胞集団に存在するすべてのRNA転写産物の集まり |
|||
* [[RNAワクチン]] - メッセンジャーRNA (mRNA) 分子のコピーを用いて免疫応答を引き起こすワクチンの一種 |
|||
* [[RNA治療]] - リボ核酸(RNA)に基づく薬物の一群と、それによる治療法 |
|||
* [[H-Invitational Database]] - ヒトnRNA配列の情報注釈データベース |
|||
== 脚注 == |
|||
{{脚注ヘルプ}} |
|||
{{reflist}} |
|||
=== 推薦文献 === |
|||
{{refbegin}} |
|||
* {{cite journal | vauthors = Alsaweed M, Lai CT, Hartmann PE, Geddes DT, Kakulas F | title = Human milk miRNAs primarily originate from the mammary gland resulting in unique miRNA profiles of fractionated milk | journal = Scientific Reports | volume = 6 | issue = 1 | pages = 20680 | date = February 2016 | pmid = 26854194 | pmc = 4745068 | doi = 10.1038/srep20680 | bibcode = 2016NatSR...620680A }} |
|||
* {{cite journal | vauthors = Lillycrop KA, Burdge GC | title = Epigenetic mechanisms linking early nutrition to long term health | journal = Best Practice & Research. Clinical Endocrinology & Metabolism | volume = 26 | issue = 5 | pages = 667–676 | date = October 2012 | pmid = 22980048 | doi = 10.1016/j.beem.2012.03.009 }} |
|||
* {{cite journal | vauthors = Melnik BC, Kakulas F, Geddes DT, Hartmann PE, John SM, Carrera-Bastos P, Cordain L, Schmitz G | title = Milk miRNAs: simple nutrients or systemic functional regulators? | journal = Nutrition & Metabolism | volume = 13 | issue = 1 | pages = 42 | date = 21 June 2016 | pmid = 27330539 | pmc = 4915038 | doi = 10.1186/s12986-016-0101-2 }} |
|||
* {{cite journal | vauthors = Vickers MH | title = Early life nutrition, epigenetics and programming of later life disease | journal = Nutrients | volume = 6 | issue = 6 | pages = 2165–2178 | date = June 2014 | pmid = 24892374 | pmc = 4073141 | doi = 10.3390/nu6062165 | doi-access = free }} |
|||
* {{cite journal | vauthors = Zhou Q, Li M, Wang X, Li Q, Wang T, Zhu Q, Zhou X, Wang X, Gao X, Li X | title = Immune-related microRNAs are abundant in breast milk exosomes | journal = International Journal of Biological Sciences | volume = 8 | issue = 1 | pages = 118–123 | date = 2012 | pmid = 22211110 | pmc = 3248653 | doi = 10.7150/ijbs.8.118 }} |
|||
{{refend}} |
|||
== 外部リンク == |
|||
{{scholia}} |
|||
{{Commons category|MRNA|lcfirst=yes}} |
|||
* {{脳科学辞典|伝令RNA}} |
* {{脳科学辞典|伝令RNA}} |
||
{{核酸}} |
{{核酸}} |
||
{{遺伝子発現}} |
{{遺伝子発現}} |
||
{{Biosci-stub}} |
|||
{{DEFAULTSORT:てんれいああるえぬええ}} |
{{DEFAULTSORT:てんれいああるえぬええ}} |
||
| 56行目: | 190行目: | ||
[[Category:スプライセオソーム]] |
[[Category:スプライセオソーム]] |
||
[[Category:RNAスプライシング]] |
[[Category:RNAスプライシング]] |
||
[[Category:ライフサイエンス産業]] |
|||
2022年10月15日 (土) 07:18時点における版
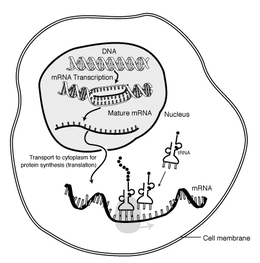
分子生物学において、伝令RNA(でんれいアールエヌエー、英: messenger ribonucleic acid)は、mRNAまたはメッセンジャーリボ核酸(メッセンジャーリボかくさん)とも呼ばれ、遺伝子の遺伝子配列に対応する一本鎖のリボ核酸(RNA)分子であり、タンパク質を合成する過程でリボソームによって読み取られる。
mRNAは、RNAポリメラーゼという酵素が遺伝子を一次転写産物のmRNA前駆体(pre-mRNAとよぶ)に変換する転写の過程で作られる。このpre-mRNAには通常、最終的なアミノ酸配列をコードしないイントロンという領域が含まれている。これらはRNAスプライシングの過程で除去され、タンパク質をコードする領域であるエクソンのみが残る。このエクソン配列が成熟mRNAを構成する。次に、成熟mRNAはリボソームによって読み取られ、リボソームは転移RNA(tRNA)が運ぶアミノ酸を利用して、タンパク質を作り出す。この過程は翻訳として知られている。これらの過程はすべて、生物系における遺伝情報の流れを説明する分子生物学のセントラルドグマの一部を形成している。
mRNAの遺伝情報は、デオキシリボ核酸(DNA)と同様にヌクレオチド配列に含まれ、おのおのが3連のリボヌクレオチドからなるコドンに配列されている。各コドンは、特定のアミノ酸をコードしているが、タンパク質合成を停止させる終止コドンは例外である。コドンからアミノ酸へ翻訳するためには、コドンを認識して対応するアミノ酸を供給する転移RNAと、リボソームに含まれるタンパク質製造装置の中心的な構成要素であるリボソームRNA(rRNA)の2種類のRNAが必要である。
mRNAの概念は、1960年にシドニー・ブレナーとフランシス・クリックによって発展した(歴史を参照)。実験検証を行う過程で、フランソワ・ジャコブとジャック・モノーが「メッセンジャーRNA(messenger RNA)」という名称を作り出した。1961年、ジェームズ・ワトソンの研究チームと、ジャコブ、モノー、マシュー・メセルソンのチームによって、mRNAが単離され、独立して記述された。
合成、プロセシング、働き
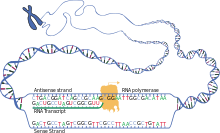
mRNA分子は転写から始まり、最終的に分解されて短い生涯を終える。mRNA分子はその寿命の間、翻訳前にプロセシング、編集、そして輸送されることもある。真核生物のmRNA分子は、しばしば広範なプロセシングや輸送を必要とするが、原核生物のmRNA分子はそうではない。真核生物のmRNA分子とそれに結合したタンパク質を合わせてメッセンジャーRNPと呼ぶ。
転写
DNAからRNAをコピーすることを転写という。転写の際、RNAポリメラーゼは必要に応じてDNAからmRNAへの遺伝子コピーを作成する。この過程は真核生物と原核生物でわずかに相違する。顕著な相違の一つは、原核生物のRNAポリメラーゼは転写中にDNA処理酵素と結合し、転写中にプロセシングを進めることができる。それによって、新しいmRNA鎖はtRNA鎖と呼ばれる相補鎖を生成して二本鎖となり、両者が結合すると塩基対形成による構造形成ができなくなる。さらに、mRNAの鋳型はtRNAの相補鎖であり、DNAが結合するアンチコドン配列と同じ配列である。短命で、未プロセシングあるいは部分的にプロセシングされた転写産物を前駆体mRNA、またはpre-mRNAと呼び、完全にプロセシングされると成熟mRNAと呼ぶ。
真核生物のpre-mRNAプロセシング

mRNAのプロセシングは、真核生物、細菌、および古細菌の間で大きく異なっている。非真核生物のmRNAは、本質的に転写された時点で成熟しており、まれな場合を除いてプロセシングを必要としない[1]。しかし、真核生物のpre-mRNAは、細胞質へ輸送されリボソームにより翻訳される前に、一連のプロセシング段階を経る必要がある。
スプライシング
RNAスプライシングは、真核生物のpre-mRNAが成熟mRNAに至る広範なプロセシングであり、イントロンやアウトロン(非コード領域)が除去され、エクソン(コード領域)が結合する機構である。
5'キャップの付加
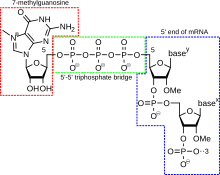
5'キャップ(5' cap、RNAキャップ、RNA 7-メチルグアノシンキャップ、RNA m7Gキャップとも呼ばれる)とは、真核生物のメッセンジャーRNAの転写開始直後にその先端部つまり5'末端に付加された修飾グアニンヌクレオチドである。5'キャップは、末端の7-メチルグアノシン残基からなり、5'-5'-トリリン酸結合を介して最初の転写ヌクレオチドに結びつく。その存在は、リボソームによる認識とリボヌクレアーゼ(RNase)酵素からの保護において重要である。
キャップの付加は転写と連動しており、相互に影響を与えるように共転写的に行われる。転写開始の直後、合成されるmRNAの5'末端は、RNAポリメラーゼに結合しているキャップ結合複合体と結合する。この酵素複合体は、mRNAのキャッピングに必要な化学反応を触媒する。合成は多段階の生化学反応として進行する。
編集
場合によって、mRNAが編集されて、そのヌクレオチド組成が変化することがある。ヒトを例にとると、アポリポタンパク質BのmRNAは、ある組織では編集されるが、他の組織では編集されない。この編集によって中途での終止コドンが作られ、翻訳時に短いタンパク質が生成する。
ポリアデニル化

ポリアデニル化(polyadenylation)とは、メッセンジャーRNA分子にポリアデニリル部を共有結合させることである。真核生物では、ほとんどのメッセンジャーRNA(mRNA)分子が3'末端でポリアデニル化されているが、最近の研究では、ウリジンの短い伸長(オリゴウリジル化)も一般的であることが示されている[2]。ポリ(A)テールとそれに結合したタンパク質は、エキソヌクレアーゼによる分解からmRNAを保護することを助ける。また、ポリアデニル化は、転写終結、mRNAの核外輸送、および翻訳にも重要である。原核生物では、mRNAがポリアデニル化されると、ポリ(A)テールがエキソヌクレアーゼ分解を妨げるのではなく、むしろ促進するように作用することもある。
ポリアデニル化は、DNAからRNAへ転写される際、および(または)その直後に起こる。転写が終了すると、RNAポリメラーゼに結合するエンドヌクレアーゼ複合体の働きによって、mRNA鎖は切断される。mRNAが切断された後、切断部位の遊離3'末端に約250のアデノシン残基が付加される。この反応は、ポリアデニル酸ポリメラーゼによって触媒される。選択的スプライシングと同様に、1つのmRNAに複数種のポリアデニル化変異体が存在する可能性がある。
また、ポリアデニル化部位の変異も起こる。遺伝子の一次RNA転写産物は、ポリA付加部位で切断され、RNAの3'末端に100-200個のアデノシン残基が付加される。この部位が変化すると、異常に長く、不安定なmRNAコンストラクトが形成される。
輸送
真核生物と原核生物のもう一つの違いは、mRNAの輸送に関するものである。真核生物では転写と翻訳は区画的に分割されているため、真核生物ではmRNAを細胞核から細胞質へ輸送しなくてはならない。この過程は、さまざまなシグナル伝達経路によって制御されている可能性がある[3]。成熟mRNAは修飾の処理によって認識され、キャップ結合タンパク質(CBC)であるCBP20およびCBP80[4]、および転写/核外輸送複合体(TREX)に結合することによって、核膜孔から輸送される[5][6]。真核生物では、複数のmRNA輸送経路が同定されている[7]。
空間的に複雑な細胞では、いくつかのmRNAは特定の細胞内目的地に輸送される。成熟した神経細胞では、ある種のmRNAが神経細胞体から樹状突起に輸送される。mRNA翻訳が行われる部位の一例は、シナプスの下に選択的に局在するポリリボソームである[8]。Arc/Arg3.1のmRNAは、シナプス活動によって誘導され、NMDA受容体が生成するシグナルに基づいて、活動的なシナプス近傍に選択的に局在される[9]。また、βアクチンのmRNAのように、外部刺激に応答して樹状突起に移動するmRNAもある[10]。アクチンのmRNAは、細胞核から輸送されるときに、ZBP1および40Sサブユニットと結合する。この複合体はモータータンパク質によって結合され、細胞骨格に沿って目的位置(神経突起伸長部)に輸送される。最終的に、ZBP1がSrcによってリン酸化され、翻訳が開始される[11]。発達中の神経細胞では、mRNAは成長中の軸索、特に成長円錐にも輸送される。多くのmRNAには、特定の場所に輸送するために、いわゆる「ジップコード(郵便番号の意)」が付与されている[12]。mRNAは、細胞膜ナノチューブ(トンネルナノチューブ)と呼ばれる構造体を通じて、哺乳動物細胞間でも移動することができる[13][14]。
翻訳
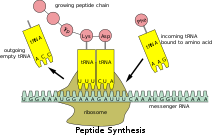
原核生物のmRNAは、プロセシングや輸送を必要としないため、転写終了後すぐにリボソームにより翻訳を開始することができる。したがって、原核生物における翻訳は転写と共役しており、共転写的に行われていると言える。
真核生物のmRNAは、プロセシングされて細胞質に輸送された後(すなわち成熟mRNA)、リボソームによって翻訳することができる。翻訳は、細胞質内を自由に浮遊しているリボソームで起こる場合と、シグナル認識粒子によって誘導されて、小胞体に結合したリボソームで起こる場合がある。したがって、原核生物とは異なり、真核生物における翻訳は転写と直接的に結びついていない。乳癌(がん)で監視されるEEF1A1のmRNA/タンパク質レベルのように、mRNAレベルの低下がタンパク質レベルの上昇を伴うこともある[15][要非一次資料]。
構造

コーディング領域
コーディング領域(coding regions)はコドン(遺伝暗号ともいう)で構成され、リボソームによって解読され、さらにタンパク質へ翻訳される。これは、真核生物では通常1つなのに対し、原核生物では通常複数である。コーディング領域は、開始コドンで始まり、終止コドンで終わる。一般に、開始コドンはAUGトリプレットで、終止コドンはUAG(アンバー)、UAA(オーカー)、またはUGA(オパール)である。コーディング領域は内部の塩基対によって安定化する傾向があり、これが分解を妨げている[16][17]。コーディング領域は、タンパク質をコードすることに加え、その一部はエクソン性スプライシングエンハンサーまたはエクソン性スプライシングサイレンサーとして、pre-mRNA中の制御配列として機能することがある。
非翻訳領域

非翻訳領域(untranslated regions、UTR)は、mRNAのうち、開始コドンの前および停止コドンの後で翻訳されない領域のことで、それぞれ5' 非翻訳領域(5' UTR)と3' 非翻訳領域(3' UTR)と呼ばれる。これらの領域はコーディング領域と一緒に転写されるため、成熟mRNA中にそのまま存在することからエクソン性(exonic)という。遺伝子発現に関わる非翻訳領域のいくつかの役割は、mRNAの安定性、mRNAの局在化、翻訳効率へ起因するとされている。UTRがこれらの機能を果たすかどうかはUTRの配列に依存し、mRNAの種類によって異なる可能性がある。また、3' UTRの遺伝子変異は、RNAの構造やタンパク質への翻訳を変化させるため、疾患感受性にも関与すると考えられている[18]。
mRNAの安定性は、リボヌクレアーゼというRNA分解酵素や、RNA分解を促進または阻害する補助タンパク質に対する親和性が異なるため、5' UTRおよび(または)3' UTRによって制御されている可能性がある (Cリッチ安定化配列も参照)。
翻訳効率は、時には翻訳を完全に阻害することも含め、UTRによって制御することができる。3' UTRまたは5' UTRに結合するタンパク質は、リボソームがmRNAに結合する能力に働きかけることで、翻訳に影響を及ぼす可能性がある。また、3' UTRに結合したマイクロRNA(miRNA)も、翻訳効率やmRNAの安定性に影響を及ぼす可能性がある。
mRNAの細胞質局在性は、3' UTRの機能であると考えられている。細胞内の特定の領域で必要とされるタンパク質は、その場所で翻訳されることもある。このような場合、3' UTRには、転写産物を翻訳するためにこの領域に局在化させる配列が含まれている可能性がある。
非翻訳領域に含まれる配列の中には、RNAに転写されると特徴的な二次構造を形成するものがある。これらの構造的なmRNA配列は、mRNAの調節に関与している。SECIS配列のようにタンパク質が結合する標的となるものもある。mRNA配列の一種であるリボスイッチは、小分子と直接結合してその折りたたみを変化させて転写や翻訳のレベルを変更する。こうした場合、mRNAはそれ自身を制御している。
ポリ(A)テール
3'ポリ(A)テール(3' poly(A) tail)は、pre-mRNAの3'末端に付加されたアデニンヌクレオチドの長い配列である(配列長は数100個が多い)。このテール(尾部)は、細胞核からの輸送と翻訳を促進するとともに、mRNAを分解から保護する役割を持つ。
モノシストロン型とポリシストロン型の違い
mRNA分子が、単一のタンパク質鎖(ポリペプチド)のみを翻訳するための遺伝情報を含む場合、モノシストロン型(monocistronic mRNA)であるという。ほとんどの真核生物のmRNAはこのようなケースである[19][20]。一方、ポリシストロン型(polycistronic mRNA)mRNAは、複数のオープン・リーディング・フレーム(ORF)を持ち、それぞれがポリペプチドに翻訳される。これらのポリペプチドは通常、関連する機能を持ち(多くは最終的な複合タンパク質を構成するサブユニット)、それらのコード配列(coding sequence、CDS)はプロモーターとオペレーターを含む制御領域にまとめられて全体として制御される。細菌や古細菌に見られるmRNAのほとんどはポリシストロン型で、ヒトのミトコンドリア・ゲノムも同様である[19]。ジシストロン型(dicistronic)またはバイシストロン型(bicistronic)のmRNAは、2つのタンパク質のみをコードしている[21]。
mRNAの環状化

真核生物では、eIF4Eとポリ(A)結合タンパク質(PABP)が相互作用し、両者が足場タンパク質のeIF4Gに結合してmRNA-タンパク質-mRNAの橋渡しをすることで、mRNA分子は環状構造を形成する[22]。環状化は、mRNA上のリボソームの循環を促進し、時間効率のよい翻訳をもたらすと考えられており、また、無傷のmRNAのみを翻訳するように機能する可能性もある(部分的に分解したmRNAは、m7Gキャップやポリ(A)テールの欠失を特徴とする)[23]。
この他に、特にウイルスmRNAで、環状化の機構が知られている。ポリオウイルスのmRNAは、その5'末端方向のクローバーリーフ部分を利用してヒトタンパク質PCBP2と結合し、PCBP2はポリ(A)結合タンパク質と結合して、よく知られたmRNA-タンパク質-mRNAの輪を形成する。オオムギ黄化萎縮ウイルスは、5'末端と3'末端のmRNAセグメント間で結合し(キッシングステムループと呼ばれる)、タンパク質を介さずにmRNAを環状化する。
RNAウイルスゲノム(その+鎖がmRNAとして翻訳される)も一般に環状化している[要出典]。ゲノム複製の際、環状化はゲノム複製速度を高めるように作用し、リボソームが循環している仮説とほぼ同様に、ウイルスRNA依存性RNAポリメラーゼを循環させる。
分解
同じ細胞内でも、mRNAの寿命(安定性)はそれぞれ異なる。細菌細胞では、個々のmRNAは数秒から1時間以上生存することができる。しかしその寿命は平均して1-3分であり、細菌のmRNAは真核生物のmRNAよりもはるかに安定性が低い[24]。哺乳動物細胞では、mRNAの寿命は数分から数日にまで及ぶ[25]。mRNAの安定性が高いほどそのmRNAからより多くのタンパク質が生成される可能性がある。mRNAの寿命が限られているので、細胞は変化する需要に応じてタンパク質合成を速やかに変更することができる。mRNAの破壊をもたらす多くの機構があり、そのいくつかを次に説明する。
原核生物のmRNA分解
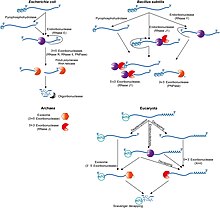
一般的に、原核生物では、真核生物よりもmRNAの寿命がはるかに短い。原核生物は、エンドヌクレアーゼ、3'エキソヌクレアーゼ、および5'エキソヌクレアーゼを含むリボヌクレアーゼの組み合わせて、メッセージ(mRNAの意)を分解する。また、数十から数百ヌクレオチド長の小型RNA(sRNA)が相補的な配列と塩基対を形成し、RNase IIIによるリボヌクレアーゼ切断を促進することによって、特定のmRNAの分解を促す場合がある。最近、細菌も5'末端に三リン酸からなる一種の5'キャップを持っていることが明らかになった[26]。このリン酸を2つ除去すると5'-リン酸が残り、5'を3'に分解するエキソヌクレアーゼRNase Jによってメッセージが破壊される。
真核生物のmRNAターンオーバー
真核細胞内では、翻訳とmRNA分解のプロセス間で釣り合いが保たれている。活発に翻訳されているメッセージは、リボソーム、真核生物翻訳開始因子eIF4EおよびeIF4G、ポリ(A)結合タンパク質によって結合されている。eIF4EとeIF4Gはデキャッピング酵素(DCP2)を阻害し、ポリ(A)結合タンパク質はエキソソーム複合体を阻害して、メッセージの末端を保護する。翻訳と分解の釣り合いは、Pボディ(P-bodies)という細胞質構造の大きさと存在量に反映される[27]。mRNAのポリ(A)テールは、RNA上のシス制御配列とトランス作用性RNA結合タンパク質の組み合わせによって、特定のメッセンジャーRNAを標的とする特殊なエキソヌクレアーゼによって短縮される。ポリ(A)テールの除去は、メッセージの環状構造を破壊し、キャップ結合複合体を不安定化すると考えられている。その後、メッセージはエキソソーム複合体またはデキャッピング複合体のいずれかによって分解される。このようにして、翻訳的に不活性なメッセージを速やかに破壊し、活性なメッセージを無傷のまま残すことができる。翻訳を停止してメッセージが崩壊複合体に渡される機構は詳しくは分かっていない。
AUリッチエレメント分解
一部の哺乳類では、mRNA中にAUリッチエレメント(ARE)が存在すると、この配列に結合してポリ(A)テールの除去を促す細胞タンパク質の作用によって、これらの転写産物を不安定化する傾向がある。ポリ(A)テールの欠失は、エキソソーム複合体[28]とデキャッピング複合体[29]の両方による攻撃を促進することにより、mRNAの分解を促進すると考えられている。AUリッチエレメントを介した速やかなmRNA分解は、腫瘍壊死因子(TNF)や顆粒球マクロファージコロニー刺激因子(GM-CSF)のような強力なサイトカインの過剰産生を防ぐための重要な機構である[30]。また、AUリッチエレメントは、c-Junやc-Fosなどの発がん性転写因子の生合成も調節する[31]。
ナンセンス変異依存mRNA分解機構
真核生物のメッセージは、メッセージ中の中途での終止コドン(ナンセンスコドン)の存在をチェックするナンセンス変異依存mRNA分解機構(NMD)による監視を受けている。ナンセンスコドンは、不完全なスプライシング、適応免疫系におけるV(D)J遺伝子再構成、DNAの変異、転写エラー、フレームシフトを引き起こすリボソームによる漏出スキャン、およびその他の原因によって発生する可能性がある。中途での終止コドンが検出されると、5'キャップ除去、3'ポリ(A)テール除去、またはヌクレオチド鎖切断による分解を引き起こす[32]。
低分子干渉RNA (siRNA)
後生動物では、酵素であるDicerによって処理された低分子干渉RNA(siRNA)は、RNA誘導サイレンシング複合体またはRISC(RNA-induced silencing complex)として知られる複合体に取り込まれる。この複合はエンドヌクレアーゼを含んでおり、siRNAが結合する完全に相補的なメッセージを切断する。その結果として生じたmRNA断片は、エキソヌクレアーゼによって破壊される。siRNAは、細胞培養において遺伝子の機能を阻害するために、実験室で一般的に使用されている。これは二本鎖RNAウイルスに対する防御としての自然免疫系の一部であると考えられている[33]。
マイクロRNA (miRNA)
マイクロRNA(miRNA)は、通常、後生動物のメッセンジャーRNAと部分相補的な配列を持つ小型RNAである[34][35]。miRNAがメッセージに結合すると、そのメッセージの翻訳は抑制されまたポリ(A)テールの除去が促進されるため、mRNAの分解は早められる。miRNAの作用機序は活発な研究対象となっている[36][37]。
その他の分解機構
メッセージが分解される機構は他にも、ノンストップ分解(non-stop decay、NSD)や、Piwi結合RNA(Piwi-interacting RNA、piRNA)によるサイレンシングなど、さまざまなものがある。
応用例
ヌクレオシド修飾メッセンジャーRNA(modRNA)配列を投与することで、細胞にタンパク質を作らせることができ、直接的にはそのタンパク質が病気を治療したり、ワクチンとして機能する可能性がある。より間接的には、このタンパク質が内在性幹細胞を望ましい方法で分化させる可能性がある[38][39]。
RNA治療の主な課題は、RNAを適切な細胞に送達することにある[40]。課題にはさらに、裸のRNA配列が調剤後に自然に分解されること、身体の免疫系がRNAを侵入者として攻撃する可能性があること、細胞膜を通過しないことといった事実も含まれる[39]。RNAが細胞内に入った後、必要なリボソームがある細胞質で活動するためには、細胞の輸送機構を離れなくてはならない[38]。
これらの課題を克服し、1989年に『広く適用可能なin vitroトランスフェクション技術が開発された後』[41]、治療薬としてのmRNAが初めて提唱された。1990年代に、非ヌクレオシド修飾mRNAに依存した、個別化がんに対するmRNAワクチンが開発された。mRNAを用いた治療法は、がんだけでなく、自己免疫疾患、代謝性疾患、および呼吸器炎症性疾患に対する治療法と処置法の両面で研究が続けられている。CRISPRのような遺伝子編集療法も、目的のCasタンパク質を作るよう細胞を誘導するためにmRNAを使用することで、有益となる可能性がある[42]。
2010年代以降、RNAワクチンやその他のRNA治療薬は「新しいクラスの医薬品」と見なされている[43]。最初のmRNAに基づくワクチンは制限付き承認を受け、COVID-19パンデミックの間に、たとえばファイザー - バイオンテックやモデルナによるCOVID-19ワクチンが世界中で展開された[44]。
歴史
1950年代初頭から、分子生物学の研究によって、タンパク質合成の際にRNAに関連する分子が存在することが示唆された。たとえば、最も古い報告の1つで、ジャック・モノーと彼のチームは、RNA合成がタンパク質合成に必要であることを示し、特に細菌の大腸菌で酵素であるβガラクトシダーゼを産生する時に必要なことを示した[45]。また、1954年にアーサー・パーディーも同様のRNA蓄積を発見した[46]。1953年、アルフレッド・ハーシー、ジューン・ディクソン、マーサ・チェイスは、大腸菌内で合成後すぐに消失する特定のシトシン含有DNA(RNAであることを示す)について報告した[47]。これは、mRNAの存在を示す最初の記録であったが、mRNAとしては特定されなかった[48]。
mRNAのアイディアは、1960年4月15日、ケンブリッジのキングス・カレッジで、シドニー・ブレナーとフランシス・クリックによって最初に着想され、フランソワ・ジャコブが、アーサー・パーディー、ジャコブ、そしてモノーが最近行った実験について話しをしているときだった。クリックの励ましを受け、ブレナーとジャコブはすぐにこの新しい仮説の検証に着手し、カリフォルニア工科大学のマシュー・メセルソンに連絡を取った。1960年の夏、ブレナー、ジャコブ、メセルソンの3人は、カリフォルニア工科大学のメセルソンの研究室で実験を行い、mRNAの存在を証明した。その年の秋に、ジャコブとモノーは「メッセンジャーRNA(messenger RNA)」と命名し、その機能を説明する最初の理論的枠組みを構築した[48]。
1961年2月、ジェームズ・ワトソンは、自身の研究グループが彼らのすぐ後を、ほぼ同じ方向で同様の実験を行っていることを明らかにした。ブレナーと他の人たちは、彼らの研究知見の論文発表を遅らせるというワトソンからの要請に同意した。その結果、1961年5月の『ネイチャー』誌にブレナーとワトソンの論文が同時に掲載され、同じ月の『ジャーナル・オブ・モレキュラー・バイオロジー(Journal of Molecular Biology)』誌にジャコブとモノーはmRNAの理論的枠組みを発表した[49][48]。
関連項目
- リボ核酸(RNA)
- タンパク質生合成 - 細胞内でタンパク質を生成する過程
- 転写 (生物学) - 細胞で遺伝情報がmRNAに転写されてタンパク質を合成する過程
- 翻訳 (生物学) - DNAの断片をRNAにコピーする過程
- リボソーム - 細胞内で生体タンパク質の合成(mRNAの翻訳)を行う高分子
- ミスセンスmRNA - 変異したコドンを持つメッセンジャーRNA
- ノンコーディングRNA - タンパク質へ翻訳されないRNA分子
- mRNA監視機構 - 生物がmRNA分子の忠実度と品質を確保するための仕組み
- トランスクリプトーム - 個体または細胞集団に存在するすべてのRNA転写産物の集まり
- RNAワクチン - メッセンジャーRNA (mRNA) 分子のコピーを用いて免疫応答を引き起こすワクチンの一種
- RNA治療 - リボ核酸(RNA)に基づく薬物の一群と、それによる治療法
- H-Invitational Database - ヒトnRNA配列の情報注釈データベース
脚注
- ^ Molecular Biology of the Gene, 7th edition. Pearson Higher Ed USA. (February 22, 2013). ISBN 9780321851499
- ^ “Widespread RNA 3'-end oligouridylation in mammals”. RNA (New York, N.Y.) 18 (3): 394–401. (March 2012). doi:10.1261/rna.029306.111. PMC 3285928. PMID 22291204.
- ^ “Regulation of mRNA export by the PI3 kinase/AKT signal transduction pathway”. Molecular Biology of the Cell 24 (8): 1208–1221. (April 2013). doi:10.1091/mbc.E12-06-0450. PMC 3623641. PMID 23427269.
- ^ “The Arabidopsis CBP20 targets the cap-binding complex to the nucleus, and is stabilized by CBP80”. The Plant Journal 59 (5): 814–825. (September 2009). doi:10.1111/j.1365-313X.2009.03915.x. PMID 19453442.
- ^ “TREX is a conserved complex coupling transcription with messenger RNA export”. Nature 417 (6886): 304–308. (May 2002). Bibcode: 2002Natur.417..304S. doi:10.1038/nature746. PMID 11979277.
- ^ “Roles of the TREX complex in nuclear export of mRNA”. RNA Biology 6 (2): 149–152. (27 October 2014). doi:10.4161/rna.6.2.8046. PMID 19229134.
- ^ “Genome analysis reveals interplay between 5'UTR introns and nuclear mRNA export for secretory and mitochondrial genes”. PLOS Genetics 7 (4): e1001366. (April 2011). doi:10.1371/journal.pgen.1001366. PMC 3077370. PMID 21533221.
- ^ “Preferential localization of polyribosomes under the base of dendritic spines in granule cells of the dentate gyrus”. The Journal of Neuroscience 2 (3): 284–291. (March 1982). doi:10.1523/JNEUROSCI.02-03-00284.1982. PMC 6564334. PMID 7062109.
- ^ “Selective targeting of newly synthesized Arc mRNA to active synapses requires NMDA receptor activation”. Neuron 30 (1): 227–240. (April 2001). doi:10.1016/s0896-6273(01)00275-6. PMID 11343657.
- ^ “Localization and translation of mRNA in dendrites and axons”. Nature Reviews. Neuroscience 2 (12): 889–898. (December 2001). doi:10.1038/35104069. PMID 11733796.
- ^ “Spatial regulation of beta-actin translation by Src-dependent phosphorylation of ZBP1”. Nature 438 (7067): 512–515. (November 2005). Bibcode: 2005Natur.438..512H. doi:10.1038/nature04115. PMID 16306994.
- ^ “Transport and localization elements in myelin basic protein mRNA”. The Journal of Cell Biology 138 (5): 1077–1087. (September 1997). doi:10.1083/jcb.138.5.1077. PMC 2136761. PMID 9281585.
- ^ “Intercellular mRNA trafficking via membrane nanotube-like extensions in mammalian cells”. Proceedings of the National Academy of Sciences of the United States of America 114 (46): E9873–E9882. (November 2017). doi:10.1073/pnas.1706365114. PMC 5699038. PMID 29078295.
- ^ “RNA transfer through tunneling nanotubes”. Biochemical Society Transactions 49 (1): 145–160. (February 2021). doi:10.1042/BST20200113. PMID 33367488.
- ^ “Contradictory mRNA and protein misexpression of EEF1A1 in ductal breast carcinoma due to cell cycle regulation and cellular stress”. Scientific Reports 8 (1): 13904. (September 2018). Bibcode: 2018NatSR...813904L. doi:10.1038/s41598-018-32272-x. PMC 6141510. PMID 30224719.
- ^ “A periodic pattern of mRNA secondary structure created by the genetic code”. Nucleic Acids Research 34 (8): 2428–2437. (2006). doi:10.1093/nar/gkl287. PMC 1458515. PMID 16682450.
- ^ “Widespread selection for local RNA secondary structure in coding regions of bacterial genes”. Genome Research 13 (9): 2042–2051. (September 2003). doi:10.1101/gr.1257503. PMC 403678. PMID 12952875.
- ^ “IFNL3 mRNA structure is remodeled by a functional non-coding polymorphism associated with hepatitis C virus clearance”. Scientific Reports 5: 16037. (November 2015). Bibcode: 2015NatSR...516037L. doi:10.1038/srep16037. PMC 4631997. PMID 26531896.
- ^ a b “Comparison of initiation of protein synthesis in procaryotes, eucaryotes, and organelles”. Microbiological Reviews 47 (1): 1–45. (March 1983). doi:10.1128/MMBR.47.1.1-45.1983. PMC 281560. PMID 6343825.
- ^ “Synexpression groups in eukaryotes”. Nature 402 (6761): 483–487. (December 1999). Bibcode: 1999Natur.402..483N. doi:10.1038/990025. PMID 10591207.
- ^ “The human mitochondrial transcriptome”. Cell 146 (4): 645–658. (August 2011). doi:10.1016/j.cell.2011.06.051. PMC 3160626. PMID 21854988.
- ^ “Circularization of mRNA by eukaryotic translation initiation factors”. Molecular Cell 2 (1): 135–140. (July 1998). doi:10.1016/S1097-2765(00)80122-7. PMID 9702200.
- ^ “Protein synthesis in eukaryotes: the growing biological relevance of cap-independent translation initiation”. Biological Research 38 (2–3): 121–146. (2005). doi:10.4067/S0716-97602005000200003. PMID 16238092.
- ^ Lewin's genes X (10th ed.). Sudbury, Mass.: Jones and Bartlett. (2011). ISBN 9780763766320. OCLC 456641931
- ^ “Structural and functional analysis of an mRNP complex that mediates the high stability of human beta-globin mRNA”. Molecular and Cellular Biology 21 (17): 5879–5888. (September 2001). doi:10.1128/mcb.21.17.5879-5888.2001. PMC 87307. PMID 11486027.
- ^ “The bacterial enzyme RppH triggers messenger RNA degradation by 5' pyrophosphate removal”. Nature 451 (7176): 355–358. (January 2008). Bibcode: 2008Natur.451..355D. doi:10.1038/nature06475. PMID 18202662.
- ^ “P bodies and the control of mRNA translation and degradation”. Molecular Cell 25 (5): 635–646. (March 2007). doi:10.1016/j.molcel.2007.02.011. PMID 17349952.
- ^ “AU binding proteins recruit the exosome to degrade ARE-containing mRNAs”. Cell 107 (4): 451–464. (November 2001). doi:10.1016/S0092-8674(01)00578-5. PMID 11719186.
- ^ “Multiple processing body factors and the ARE binding protein TTP activate mRNA decapping”. Molecular Cell 20 (6): 905–915. (December 2005). doi:10.1016/j.molcel.2005.10.031. PMID 16364915.
- ^ “A conserved AU sequence from the 3' untranslated region of GM-CSF mRNA mediates selective mRNA degradation”. Cell 46 (5): 659–667. (August 1986). doi:10.1016/0092-8674(86)90341-7. PMID 3488815.
- ^ “AU-rich elements: characterization and importance in mRNA degradation”. Trends in Biochemical Sciences 20 (11): 465–470. (November 1995). doi:10.1016/S0968-0004(00)89102-1. PMID 8578590.
- ^ “Quality control of eukaryotic mRNA: safeguarding cells from abnormal mRNA function”. Genes & Development 21 (15): 1833–1856. (August 2007). doi:10.1101/gad.1566807. PMID 17671086.
- ^ “The evolution of RNAi as a defence against viruses and transposable elements”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 364 (1513): 99–115. (January 2009). doi:10.1098/rstb.2008.0168. PMC 2592633. PMID 18926973.
- ^ Robert E. Farrell, Jr. RNA Methodologies, 5th Edition. Academic Press, 2017
- ^ “Principles of microRNA-target recognition”. PLOS Biology 3 (3): e85. (March 2005). doi:10.1371/journal.pbio.0030085. PMC 1043860. PMID 15723116.
- ^ Tasuku Honjo, Michael Reth, Andreas Radbruch, Frederick Alt. Molecular Biology of B Cells, 2nd Edition. Academic Press, 2014 (including "updated research on microRNAs")
- ^ “Deadenylation is a widespread effect of miRNA regulation”. RNA 15 (1): 21–32. (January 2009). doi:10.1261/rna.1399509. PMC 2612776. PMID 19029310.
- ^ a b “Tools for translation: non-viral materials for therapeutic mRNA delivery”. Nature Reviews Materials 2 (10): 17056. (12 September 2017). Bibcode: 2017NatRM...217056H. doi:10.1038/natrevmats.2017.56.
- ^ a b “RNA-Based Therapeutics and Vaccines”. Genetic Engineering News. (2015年9月15日)
- ^ “Advances in the delivery of RNA therapeutics: from concept to clinical reality”. Genome Medicine 9 (1): 60. (June 2017). doi:10.1186/s13073-017-0450-0. PMC 5485616. PMID 28655327.
- ^ “Developing mRNA-vaccine technologies”. RNA Biology 9 (11): 1319–30. (November 2012). doi:10.4161/rna.22269. PMC 3597572. PMID 23064118.
- ^ “The mRNA revolution: How COVID-19 hit fast-forward on an experimental technology” (英語). New Atlas (2021年4月23日). 2021年4月26日閲覧。
- ^ “mRNA-based therapeutics–developing a new class of drugs.” (英語), Nature Reviews Drug Discovery 13 (10): pp. 759–780, (2014), doi:10.1093/nar/gku757, PMC 4176373, PMID 25150148
- ^ “The clinical progress of mRNA vaccines and immunotherapies”. Nature Biotechnology 40 (6): 840–854. (June 2022). doi:10.1038/s41587-022-01294-2. PMID 35534554.
- ^ “La cinétique de la biosynthèse de la β-galactosidase chez E. coli considérée comme fonction de la croissance” (フランス語). Biochimica et Biophysica Acta 9 (6): 648–660. (1952). doi:10.1016/0006-3002(52)90227-8. PMID 13032175.
- ^ “Nucleic Acid Precursors and Protein Synthesis”. Proceedings of the National Academy of Sciences of the United States of America 40 (5): 263–270. (May 1954). Bibcode: 1954PNAS...40..263P. doi:10.1073/pnas.40.5.263. PMC 534118. PMID 16589470.
- ^ “Nucleic acid economy in bacteria infected with bacteriophage T2. I. Purine and pyrimidine composition”. The Journal of General Physiology 36 (6): 777–789. (July 1953). doi:10.1085/jgp.36.6.777. PMC 2147416. PMID 13069681.
- ^ a b c “Who discovered messenger RNA?”. Current Biology 25 (13): R526–R532. (29 June 2015). doi:10.1016/j.cub.2015.05.032. PMID 26126273.
- ^ F. Jacob, J. Monod "Genetic regulatory mechanisms in the synthesis of proteins". Journal of Molecular Biology, 3 (1961), pp. 318-356.
推薦文献
- “Human milk miRNAs primarily originate from the mammary gland resulting in unique miRNA profiles of fractionated milk”. Scientific Reports 6 (1): 20680. (February 2016). Bibcode: 2016NatSR...620680A. doi:10.1038/srep20680. PMC 4745068. PMID 26854194.
- “Epigenetic mechanisms linking early nutrition to long term health”. Best Practice & Research. Clinical Endocrinology & Metabolism 26 (5): 667–676. (October 2012). doi:10.1016/j.beem.2012.03.009. PMID 22980048.
- “Milk miRNAs: simple nutrients or systemic functional regulators?”. Nutrition & Metabolism 13 (1): 42. (21 June 2016). doi:10.1186/s12986-016-0101-2. PMC 4915038. PMID 27330539.
- “Early life nutrition, epigenetics and programming of later life disease”. Nutrients 6 (6): 2165–2178. (June 2014). doi:10.3390/nu6062165. PMC 4073141. PMID 24892374.
- “Immune-related microRNAs are abundant in breast milk exosomes”. International Journal of Biological Sciences 8 (1): 118–123. (2012). doi:10.7150/ijbs.8.118. PMC 3248653. PMID 22211110.
