DNA複製

DNA複製(ディーエヌエイふくせい、英: DNA replication)は、細胞分裂における核分裂の前に、DNAが複製されてその数が2倍となる過程である。生物学ではしばしば複製 (replication) と略される。セントラルドグマの一員とされる。複製される一本鎖DNAを親鎖 (parent strand)、DNA複製によって新しく合成された一本鎖DNAを娘鎖 (daughter strand) という。また、DNA複製により生じた染色体の個々を姉妹染色分体 (sister chromatid) という。
複製の機構の概説[編集]

DNA複製は複製開始 initiation 、伸長 elongation 、終結 termination の3段階で進む[1]。なお、二重らせんをとる二本鎖DNAをdsDNA ( double-stranded DNA )、そうでない一本鎖DNAをssDNA (single-stranded DNA) と表記する。
複製は、DNA上の特別な塩基配列である複製起点(replication origin、起点:origin)から開始される。複製起点周辺で部分的に二重らせんが解かれ、親鎖の途中に2本のssDNAが現れる。直ちに、さまざまな酵素の複合体がssDNAに結合し、プライマーと呼ばれる短いRNAがssDNA上に合成される。ここまでが複製開始段階である。次の伸長段階で、DNA合成酵素のDNAポリメラーゼ (DNA polymerase) を含む複合体(複製装置、replication machinery)が親ssDNAに結合する。まずDNAポリメラーゼは、プライマーの3'末端と結合している親ssDNA上の塩基の隣の塩基を識別し、それと相補的なデオキシヌクレオチド[注釈 1]をプライマーの末端に付加させる。それ以降、DNAポリメラーゼは親ssDNA上を5'から3'の方向へ移動しながら親ssDNAと相補的な塩基を娘鎖末端に付加させていく。同時に、娘鎖は親鎖と二重らせんを形成する。これと並行して、二重らせんのままの未複製部分は順次解かれていく。これが繰り返され、最終的に完全に複製した娘鎖が出来上がる。
半保存的複製[編集]
半保存的複製 (semiconservative replication) とは、一般に、DNA複製により合成された2本の二重らせんDNAが1本の娘鎖と1本の親鎖から構成されていることである。DNA複製の機構が半保存的複製であることは1958年にマシュー・メセルソンとフランクリン・シュタールにより証明された(メセルソン-スタールの実験)[2][3]。
半不連続的複製[編集]
半不連続的複製 (semidiscontinuous replication) とは、2本の親鎖のうち一方を連続的に、もう一方を半不連続的に合成するDNA複製一般の様式のことである。連続的および不連続的に合成された娘鎖をそれぞれ、リーディング鎖 (leading strand) およびラギング鎖 (lagging strand) という。DNA複製が半不連続的であることは岡崎令治により証明された[4][5]。
DNA複製が半不連続的であることは、DNAポリメラーゼがデオキシヌクレオチドの付加を、RNAとDNA両方において3'末端へしか行えないことに由来する。このことは、複製の方向を親鎖の5'から3'への方向に限定する。ほどかれた2本のssDNAは、複製前のdsDNAがssDNAにほどかれる分岐点 (複製フォーク:replication fork、伸長フォーク:growing fork) の拡大方向が複製方向と平行なものとそうでないものに分かれる。前者のssDNAおよび後者のssDNAで合成された娘鎖がそれぞれリーディング鎖およびラギング鎖である。リーディング鎖合成ではたった1つのプライマーが合成されて、複製フォークの拡大で露出した未複製の塩基を1つのDNAポリメラーゼが複製し続ける。対して、ラギング鎖合成では露出した未複製の塩基と反対の方向へとDNAポリメラーゼが進んでいくため、複製フォークが何bpか拡大するたびにプライマーが合成されなければならない。いくつものプライマーから短いDNA断片(岡崎フラグメント)の合成が繰り返され、岡崎フラグメントの連結・統合によりラギング鎖は完成する。
複製開始[編集]

複製開始には多くのタンパク質が関わり、いくつもの段階を経る。実際に娘鎖が合成される伸長段階を始めるためには、親鎖が二重らせんのないssDNAである必要がある。これは、複製に関わるタンパク質がその役割を果たすためにはssDNAとなった親鎖に結合する必要があるためである。また、親鎖と新たに合成された娘鎖が新しい塩基対を形成しなければならない。そのため、複製開始段階は二重らせんを解くことから始まり、イニシエーターによる巻き戻しが第1段階である。複製開始第2段階は娘鎖合成の足掛かりとなるプライマーの合成である。娘鎖を合成するDNAポリメラーゼは複製を開始するためには短いRNAであるプライマーが必要である。最後の段階は娘鎖伸長に関わるタンパク質が親鎖に集合することである。
レプリコン[編集]
1つの複製起点によって巻き戻しが及ぶ範囲をDNA複製の単位とし、これをレプリコン(replicon)と呼ぶ。この言葉は、フランソワ・ジャコブ、シドニー・ブレナー、Jacques Cuzin らが1963年に提唱した「レプリコン説」で定義された。レプリコン説は、もともと細菌の複製開始(レプリコンの点火)を制御する仕組みのモデルだったが、生物一般に成り立つことがわかっている。
レプリコンには、複製起点を含めたレプリケーターと呼ばれる塩基配列が存在する。レプリケーターは、比較的結合が弱い塩基対であるアデニンとチミンが多いATリッチ配列を含み[5][6][7]、イニシエーターと呼ばれるタンパク質がレプリケーター内の複製起点に結合するとATリッチ配列の巻き戻しが起こる。一般に、イニシエーターには少なくとも、複製起点への結合によるレプリコンの点火と、複製開始に必要なほかの因子をレプリケーターに引き寄せることの2つの役割を持つことが知られている。また、イニシエーターには、結合部位近くのDNAを曲げたり、その二重らせんをほどいたりするという第3の働きをして、複製開始後の伸長段階でのヘリカーゼによる巻き戻しを促進しているものもある。例えば、大腸菌のイニシエーターであるDnaAはレプリケーター (oriC) に5つある9bpの反復配列にまず結合してATPによる制御を受けるが、リン酸化前の、ATPと結合している状態のDnaAはoriCに3つある13bpの反復配列にも結合する。その結果、その13bp配列からそれぞれ20bp以上の巻き戻しが起こる[8]。
レプリコンは原核細胞の染色体に1つしかないが、真核細胞の場合は複数存在する。複製の開始位置の分散はDNA複製の早期終結に寄与していると考えられている[9]。
プライマーの導入[編集]
複製開始から伸長段階へ移行する前にDNAプライマーゼ primase により親ssDNA上に短いRNA鎖(プライマー)が合成される。プライマーとssDNAが結合したものをプライマー-鋳型接合体 (primer:template junction) という。プライマーの3'末端(プライマー末端)には三リン酸があり、DNAポリメラーゼはこのリン酸基を分解することで生じるエネルギーを用いて、プライマー末端と塩基対形成している塩基の5'側の隣の塩基(プライマーと結合している領域の手前)と相補的なデオキシヌクレオチド三リン酸を生成してプライマー末端に結合させる。以降、DNAポリメラーゼは娘鎖の3'末端の三リン酸の分解エネルギーを利用して娘鎖の伸長を進める。
伸長[編集]


伸長段階はDNAポリメラーゼによる娘鎖の合成である。前述の理由(半不連続的複製)により、娘鎖は合成様式が連続的なリーディング鎖と不連続的なラギング鎖に分かれる。リーディング鎖とラギング鎖は同時に合成されるが、これは染色体中にssDNAが存在する時間を短くするためであると考えられる[10]。DNAは紫外線や化学物質による損傷の危険性に常にさらされている。特に弛緩状態のssDNAはdsDNAと比べて切断されたときの修復がはるかに難しく、修復の際に変異を招いてしまうことが頻繁にある[10]。鋳型鎖の切断によるDNA複製が停止した場合は、相同組換えによって複製は再開されるが、水野健一らの研究によると相同組換えにより再開したDNA複製は誤りがちで、特に逆位反復配列での再開は高頻度で染色体の再編成を引き起こす[11]。
複製装置[編集]
複製装置とは、DNAヘリカーゼによってほどかれた部分的ssDNA上に形成された、そのDNAヘリカーゼも含むDNA複製に関与する因子により構成される複合体である。複製装置はプライモソームを取り込んでおり、その構成因子はDNAポリメラーゼ、DNAへリカーゼ、DNAクランプ、DNAトポイソメラーゼなどの酵素および一本鎖DNA結合タンパク質 (SSB) などのタンパク質である(後に詳述)[12]。複製装置の中でこれら構成因子は高度に協調的に機能する。細菌の大部分では、プライモソームを含む複製に関与する因子が全て複製フォークに集まり、複合体はそこに留まり続ける。このような複製装置はレプリソーム (replisome) またはDNAレプリカーゼ系 (DNA replicase system) と呼ぶ(これらの語は、もともと複製フォークに集まるタンパク質の総称)。一方、真核生物と一部の細菌では、レプリソームは形成されずに数百あるいは数千の複製装置が形成される[12]。
複製装置は、複製されるDNAに対して相対的に工場のように動かない存在であるため、複製工場 (replication factory) とも呼ばれている[12][13]。このことを他に例えるなら、複製装置は映写機で、そこに映画のフィルムのようにDNAが流れて通過し続ける。複製工場モデルにおいて、1つの複製フォークにおけるリーディング鎖とラギング鎖それぞれの2つのDNAヘリカーゼは互いに結合し、複製過程中ずっと離れない。Peter Meisterらは、出芽酵母のDNAポリメラーゼαといくつかの遺伝子座を緑色蛍光タンパク質 (GFP) でタグして複製部位を直接観察できるようにし、1つの複製起点から対称的に離れた2つの遺伝子座の距離が経時的に著しく減少することを発見した[14]。この発見は、鋳型DNAは複製されるために複製装置へと移動し、また、リーディング鎖とラギング鎖それぞれの複製装置が互いに協調していることの直接的な証拠である。その後、DNAヘリカーゼが複製中に二量体を形成していることが多くの真核生物で確認され、また、細菌の複製装置はDNA合成の際に細胞内の一か所に留まっていることが確認された[13]。
複製工場はまた、複製後に姉妹染色分体を娘細胞に分配するための引き離しに不可欠な、姉妹染色分体同士のもつれの解消を実行する。複製後に姉妹染色分体はコヒーシンによって連結されるため、もつれの解消は複製中にしかできない。複製装置が複製工場として核内で固定されている理由は、複製フォークが自由に動くことは染色体の連環の形成を誘導して有糸分裂分離を阻害するためと考えられている[14]。
巻き戻し[編集]
DNAポリメラーゼをはじめとする多くの複製因子が機能するためには、親鎖の二重らせんを二本のssDNAに分解する巻き戻しが必要である。巻き戻しは、酵素反応による、二重らせん構造を維持する水素結合の切断である。最初の巻き戻しはイニシエーターとDNAの結合により複製起点で起こり、以降はDNAヘリカーゼにより巻き戻しの範囲が拡大する。最終的に複製終結点(停止点)まで巻き戻しは進む。
巻き戻しは可逆反応であるため、別れたssDNAは再び二重らせんを構築しようとする(これを「再会合」と言う)。このため、親鎖が巻き戻されるとすぐに一本鎖DNA結合タンパク質(single-strand binding protein:SSB、らせん不安定化タンパク質:helix-destabilizing protein)が結合して再会合は防がれる[1][15]。DNAと結合したSSBは遊離SSBに対する化学親和性が非常に大きくなり、DNAと結合したSSBの隣に次のSSBがそのSSBとDNAとに結合し、これが繰り返されて複製バブル全体をSSBが覆う。例えば、T4ファージのSSBであるgp32の場合、ssDNAと結合した分子は次の分子の化学的親和性が1000倍になる[16]。また、SSB間の結合は個々のSSBのDNAへの結合を安定化させる。SSBが直接結合するDNAの部位は塩基でないので[注釈 2]、塩基間の水素結合により娘鎖を伸長させていく複製装置の邪魔をすることはない。さらに、DNAを伸びた状態にする効果もあるので、後述する娘鎖合成やプライマー合成の鋳型になりやすい[17]。こうして、巻き戻し(と後述する超らせんの解消)を経て生まれる部分的な1本鎖DNAの領域が複製バブル、二重らせんとの分岐点が複製フォークである。
複製起点に続いての水素結合の切断は、酵素であるDNAヘリカーゼが担う[1][5]。複製起点では親鎖の巻き戻しと同時に、それぞれの親1本鎖で複製装置による娘鎖の合成が始まる。DNAヘリカーゼによりさらに親鎖が巻き戻ると、これと同時にほどけた親鎖に沿って複製が進行する。実際、複製は巻き戻しと同じ速度で、どんな場合でも、ほどけている親鎖で伸長中の娘鎖と対になっていない部分や複製途中の部分はごく短い。このことは、巻き戻しが伸長段階と強力に共役していることを表す。
場合によって、1つの複製バブルにおける2つの複製フォークのうち、DNAへリカーゼが進行させるのが両方共(双方向性)か、片方だけ(一方向性)かが異なる。双方向性が確認された最初の生物は枯草菌(Bacillus subtilis)である[18]。その後、真核生物のキイロショウジョウバエ(Drosophila melanogaster)[18]やイモリ[19]でも発見された。現在では、真核生物でも原核生物でもほとんどのDNA複製は双方向性であると考えられている[9][20]。一方で、colE1と呼ばれるプラスミド(染色体外の環状DNA)などで一方向性のDNA複製が確認されている[9]。
DNAのよじれの解消[編集]

DNAへリカーゼが二重らせんをほどく際、dsDNAによじれ torsion が生じるという重大な問題が発生する。dsDNAは10 bp(10塩基対)ごとに1巻きのらせん(ターン)であるため、10 bp巻き戻すたびに他の領域までも縦軸を中心に1回転する。この回転は、両端の切れた不自然に短い直鎖状のDNAならば問題にならない。しかし、細菌や大多数のウイルスのdsDNAは環状である。そのまま巻き戻そうとすると必ずどこかが強くよじれてしまう。また、真核生物の染色体は直鎖状とはいえ巨大であり、しかも、各所で核マトリックス同士が結合してループ構造を形成している。このループ構造は環状同様に閉鎖的であるため、やはりよじれの発生は必至である。
この結果、自由に回転できないDNAは巻き戻しによるよじれのために、さらに大きならせん(DNA超らせん、あるいはDNAスーパーコイル)が生じる[21]。ちょうど、電気コードの両端を持って数回ねじると大きな輪が生まれるのに似ている(360度回すごとにコードは1回交差する)。DNAらせんは右回りであるため、複製フォークの進行方向で形成される超らせんも右回り(正の超らせん)。正の超らせんが長くなると巻き戻しに対する抵抗となり、複製フォークの進行を止めてしまう。DNA複製がスムーズに行われるためには逆向きの超らせん(負の超らせん)を導入するようDNAを巻き、正の超らせんを中和させればよい。先ほどのねじれたコードで例えると、一方の手を放すと逆方向に回転してねじれのストレスは解消する。
このようなよじれ解消機構を、1963年に正の超らせん問題を発見したケーンズ Cairns はスイベル (swivel) と名付けた。当時は仮説の存在だったが、現在ではスイベルの担い手である一群の酵素(DNAトポイソメラーゼ)が明らかになっている。DNAトポイソメラーゼは、巻き戻された一方のDNAを切断し、もう一方のDNAをその間隙に通過させたあとで再結合するという一連の反応を触媒する。この活性の効果は、DNAの構造的ストレスの指標であるリンキング数(絡まり数: Lk)により数値化できる[21]。リンキング数は、1つのdsDNAのターン数(ツイスト数、ねじれ数: Tw)と超らせんの数(ライジング数、巻数: Wr)の和 (Lk = Tw + Wr ) である(DNA超らせんの項参照)。例えば4,000 bpを持つ環状dsDNAの場合、1巻きが10 bpであるためツイスト数は400 (Lk = 400 + 0)。これを10 bpだけ巻き戻すと、ツイスト数が1減り、複製フォーク手前に正の超らせんが1巻き生じる (Lk = 399 + 1)。次の10 bpを巻き戻し、かつDNAトポイソメラーゼにより負の超らせんを1巻き生成すると、リンキング数はようやく減る (Lk = 398 + (1 – 1) = 398)。これは、正の超らせんと負の超らせんが互いに打ち消し合い、よじれから解放されたことを意味する。DNAの超らせんのない状態を弛緩型 と呼ぶ。
DNAクランプ[編集]
DNAクランプ(DNA clamp、スライディングクランプ:sliding clamp)とは、DNA複製の伸長段階に関わるタンパク質の一つである。右図のようなドーナッツ状で、中央の穴(約35オングストローム)にDNAポリメラーゼが複製したdsDNA(約20オングストローム)を通す[23]。通したDNAとの間には水分子1-2個が層を作るだけの余地がある[24]ため、DNAクランプはDNAから離れることなくDNA上を滑って動く。
DNAポリメラーゼは単独でDNAと長時間結合することができず、平均20〜100 bpほどまでしか合成できない[24]。さらに、DNAポリメラーゼが遊離してから再びDNAに戻るのに1分ほどかかる[25]。DNAと非常に安定に結合したDNAクランプはDNAポリメラーゼと強固に結合してDNAから離れないようにし、DNAポリメラーゼの活性における持続時間(連続反応性、プロセッシビティー)を維持する[1][26]。DNAポリメラーゼとDNAとの結合はたびたび切れるが、DNAポリメラーゼはDNAクランプに固定されているためすぐに合成を再開する。
強固にDNAポリメラーゼと結合するDNAクランプだが、迅速に分離させる機構もある。ラギング鎖はいくつもの岡崎フラグメント合成を必要とするし、真核生物などでは多くのレプリコンを多くのDNAポリメラーゼで複製している(#レプリコン参照)。DNAポリメラーゼの仕事は、すでに娘鎖もしくはプライマーRNAが合成された後の二重らせん領域(例えば岡崎フラグメントの末端)に到達したときに終了する。dsDNAと結合したDNAポリメラーゼは立体構造を変化させ、DNAクランプとの化学親和力を著しく下げることで、直ちにDNA から離れる[23]。
DNAクランプはDNAポリメラーゼを放出した後もDNAからしばらく離れず、複製後のDNAに働くほかのタンパク質の留め金となる。例えば、増殖細胞核抗原(PCNA: Proliferating Cell Nuclear Antigen)と呼ばれる真核生物のDNAクランプは、dsDNAをクロマチンという重要な立体構造に組み立てる酵素を新しい二重らせんに導く[27]。また、岡崎フラグメントの修復にかかわる真核生物のタンパク質もDNAクランプと結合することで正しく機能する[27]。DNAクランプと結合する全てのタンパク質には、5アミノ酸残基からなるクランプ結合配列(Gln-Leu-Ser/Asp-Leu-Phe)を持っている[28]。
DNAクランプは特にラギング鎖において多数必要となるため、複製フォークにおいて大多数のDNAクランプが集合する。末次正幸らはDNAクランプが集合した場所をクランプゾーンと名づけ、枯草菌 (Bacillus subtilis) の細胞内においてクランプゾーンが形成される様子の定量的観測に成功した[28]。複製開始前の細胞では約600個あるDNAクランプは細胞全体に拡散していたが、複製が始まると毎秒1分子が複製フォークに集まり、2~3分後に約200分子からなるクランプゾーンを形成した。それ以降は、DNAクランプの数は一定となった。毎秒1分子というDNAクランプの集合の頻度は岡崎フラグメントの形成頻度(毎秒0.5~1フラグメント)とほぼ一致するため、集合したDNAクランプはおのおの異なる岡崎フラグメントに結合すると考えられている。実際、DnaG(プライマーゼ)の枯草菌細胞内濃度を減少させるとクランプの集合頻度が1/3になり、クランプゾーン形成の所要時間が3倍に延びたことが報告されている。
DNAクランプはウイルスや細菌、微生物から人まで非常に広い範囲に存在し、どれも機能や構造が酷似している。どの生物の場合でも6回回転対称性を持ち、直径も約35オングストロームと同じである[27]。ただし、構成するサブユニットの数などは異なる。
DNAへのDNAクランプの装着および取り外しは、クランプローダー (clamp loader) が行う。大腸菌のクランプローダータンパク質はγ複合体である[10]。γ複合体は2つのτタンパク質を含み、それぞれ次に説明する脱着に関わる部位と柔軟なポリペプチドにより連結している[29]。γ複合体は指のような5本のサブユニットからなり、見た目はマジックハンドのようである。この5本の指先に大腸菌のDNAクランプであるβクランプが結合する。βクランプは閉じた環状構造をしているが、γ複合体は指に結合させている間これを開く。放せば環状構造は閉じる。これにより脱着を行うが、その時期は制御されている。装着はDNA上にプライマーが形成されれば実行する。取り外しの時期には制限する条件があり、それはβクランプがほかのタンパク質と結合していないことである[10]。細菌のDNAポリメラーゼ (pol III) はもちろんのこと、上記のヌクレオソーム集合因子やDNA修復タンパク質と結合している場合は働かない。一方、τタンパク質はDNAポリメラーゼ(のコア酵素)とDNAヘリカーゼに結合する。これはレプリソームが形成されている限り、すなわちレプリコンの複製が終わるまでずっと続く。
ニックトランスレーション[編集]

DNAリガーゼは隣り合ったデオキヌクレオチド間のジエステル結合を触媒するが、大腸菌の場合、末端に少なくとも数塩基のssDNAがはみ出し、かつ互いのその部分が塩基対を成す2本のdsDNAを通常必要とする[30]。娘鎖が完成するためには、DNA複製開始の土台として合成されたプライマーRNAをDNAに変換しなければならない。この過程をニックトランスレーションと呼ぶ[31][32]。まず、RNアーゼHがプライマーを発見し、除去する[33]。ただし、娘DNAの末端に結合した(プライマーの末端であった)リボヌクレオチド[注釈 1]は除けない。なぜなら、RNアーゼHの機能はリボヌクレオチド間の結合切断に過ぎないからである。そこで、末端のリボヌクレオチドは 5'エキソヌクレアーゼが受け持つ[33]。この酵素はDNAおよびRNAを5'末端から分解する。こうして、娘鎖に紛れたRNAの除去は完了する。
次に、プライマーの消失により生まれた隙間(ギャップ)をDNAが埋める。こちらも2つの酵素による2段階であるが、まずDNAポリメラーゼが執り行う[33][1]。ギャップ端のDNA3'を土台に、ギャップは完全に埋まる。しかし、ここまでで完全な娘鎖が出来上がったわけではない。ここで埋まるDNAとギャップ端だったDNAはつながらず、このままでは娘鎖に切れ目(ニック)が残る。DNAポリメラーゼは認識した3'末端に新しいデオキシヌクレオチドの5'末端をつなげるだけなので、補完DNAの3'末端と、最終的にぶつかるDNAの5'末端との結合を触媒しない。こうして生まれるニックはDNAリガーゼ(DNA連結酵素)で連結される[33][5][1]。上記4つの酵素によりプライマーRNAは完全にDNAと置き換わる。
終結[編集]
上記の過程(複製開始、伸長)はレプリコンの終わりまで続く。レプリコンの終わり、すなわち複製終結点に複製フォークがたどり着いたときに終結段階が始まり、複製は完了する。この段階でレプリソームはDNAから解離する。この後に2つの大きな問題が待ち構えている。カテナンによる娘鎖の絡まりと末端複製問題である。
脱カテナン化[編集]
脱カテナン化 (decatenation) は2本の娘鎖の合成を終えた後にされなければならない、極めて重要な作業の一つである。細菌の環状DNAは、DNA複製完了時に2つの娘鎖がカテナンを形成する[34]。カテナンとは、2つの環状高分子が絡まった状態であり、このままでは複製を完了したDNAを娘細胞に分配することができない。
細菌の脱カテナン化の前には、修復合成が行われる必要がある。複製フォークが複製終結点に到達したとき、複製の完了していない領域が残っているためである。修復合成はまず、この未複製の2重鎖DNAが解かれるところから始まる。一本鎖になった領域で最後のDNA合成が行われ、2本の2重鎖DNAが完成する。しかしこの段階に至っても、2つのDNAは互いにらせん状に巻きついたトーラスと呼ばれる構造を介して連結している[35]。このときの娘鎖同士の交差(つなぎ目)の数は修復合成前の親dsDNAにおける親ssDNAが交差していた数(ツイスト数の2倍)に等しい[35]。修復合成が完了した後、脱カテナン化を担うのが、II型DNAトポイソメラーゼである。大腸菌やサルモネラ菌では、DNAトポイソメラーゼ IV (topo IV) がこの役割を果たす(この反応に関わる酵素はdecatenaseと呼ばれることもある)。topo IVの変異株は染色体分離に欠損を示して致死に至る[35]。
真核生物の線状ゲノムでも、1つの複製終結点を目指して互いに近づく隣接レプリコンの間にカテナンと似た2重鎖DNA絡まりが生じる[35]。真核生物では、DNAトポイソメラーゼ II (topo II) が脱カテナン化を担う。
末端複製問題[編集]

ゲノムが直鎖状DNAである真核生物では、DNAポリメラーゼによって親鎖の3'側の最末端領域を複製できない。末端複製問題と呼ばれるこの問題は、DNAポリメラーゼが事前に用意されたプライマーの3'末端からしかでデオキシヌクレオチドの重合ができないために起こる(詳しくは#伸長)。すなわち、プライマーを置くためのスペースが3'側にないため、このままでは娘鎖は親鎖よりも短くなってしまう。
末端複製問題は次の3つの段階を経て解決する。合成が終わり、プライマーが除去された後、真核生物の新生DNAの娘鎖は5‘末端が欠けている。テロメラーゼという酵素がまず、親鎖の3’末端を鋳型鎖なしに伸長させる。次に、本来よりも長くなった3‘末端にプライマーは置かれ、DNAポリメラーゼが複製する。ここでもやはり短く複製されるが、娘鎖は本来の長さになる。
DNA複製に関係するタンパク質[編集]
ここでは、DNA複製に関係するタンパク質をいくつか簡単に取り上げる。ただし、DNA複製中に行われるDNA修復に関わるもの(DNAフォトリアーゼなど)やテロメア複製に関わるもの(テロメラーゼなど)は除く。
- DNAポリメラーゼ
- 詳細は「DNAポリメラーゼ」を参照DNAを合成する反応を行う酵素をDNAポリメラーゼと呼ぶが、1つの生物種がいくつもの種類を持つ。大腸菌の場合、DNAポリメラーゼI (pol I) とII (pol II)、III (pol III) がある。このうち、このページで登場した、DNAの合成を担うのはpol IIIである。すなわち、細菌のDNA複製の担い手はpol IIIである。真核生物の場合、DNAポリメラーゼはα、β、γ、δ、εの5種類。DNA伸長をするのはDNAポリメラーゼδである。αはプライマーゼ、βとεがDNA修復を担う。γはミトコンドリアのDNA複製を行う。さらに、ヒトにはDNA修復にかかわる酵素としてDNAポリメラーゼζ、η、θ、ι、κも発見されている。
- DNAクランプ
- 詳細は「DNAクランプ」を参照
DNAポリメラーゼと特異的かつ強力に結合し、伸長反応中にds-DNAから解離することを防ぎ、伸長段階の連続反応性を保証するタンパク質。
- DNAリガーゼ
- 詳細は「DNAリガーゼ」を参照DNA2本鎖中に、5'-末端がリン酸基 (5'-P) 、3'-末端がヒドロキシ基 (3'-OH) の状態の1本鎖切断部位(ニック)が存在するとき、この部位を認識してホスホジエステル結合により連結する酵素である。DNA複製時に、岡崎フラグメントの連結を行うほか、修復合成や組み換え反応におけるDNA鎖連結反応にも関与する。
- DNAトポイソメラーゼ
- 詳細は「DNAトポイソメラーゼ」を参照DNAのリンキング数を変えて別のトポロジー体(トポイソマー)に変換させる酵素。この変換のためにDNAを一時的に切断するが、その様式によってI型とII型の2種類に分類される。I型は二本鎖の一方の鎖だけを一時的に切断し、一方、II型は両鎖の一時的切断を引き起こす。I型は#DNAのよじれの解消で、II型は#脱カテナン化およびヌクレオソームの組み立てで活躍する。
- DNAヘリカーゼ
- 詳細は「ヘリカーゼ」を参照親鎖の二重らせんをほどくことで、複製フォークを進行させる巻き戻し酵素。
- DNAプライマーゼ
- 詳細は「DNAプライマーゼ」を参照プライマーを合成する酵素。
真正細菌のDNA複製[編集]
真正細菌のDNA複製については、主に大腸菌とそれに感染する大腸菌ファージを用いた研究により大部分が解明されている。大腸菌ファージは非常に単純なゲノムを持ったウイルスで、複製は基本的に宿主のタンパク質を利用するので研究には欠かせない。
真正細菌のレプリコン[編集]
原核細胞のゲノムは単一のレプリコンである。DNA複製は常に唯一の複製起点から、細胞周期の中ただ一度だけ実行される。この仕組みを単コピー型と呼ぶ[36][37]。ただし、真正細菌の中にはコレラ菌のように複数の環状染色体を持つものや、ボレリア菌のように複数の線状染色体をもつものも存在する。原核細胞にはゲノムDNAだけでなく、自律的に増殖する染色体外DNAたるプラスミドも存在する場合が多い。こちらは染色体同様に単コピー型制御の場合もあれば、それとは異なる多コピー型制御を受ける場合もある[36][37]。多コピー型の制御下では一回の細胞周期中にプラスミド複製が繰り返され、細胞中に複数のコピー体が存在することになる。
大腸菌と枯草菌の場合、DNA複製を終わらせるter配列に独特で面白い性質がみられる。両方向にほぼ同じ速さで進む2つの複製フォークは複製起点oriC から半周した位置に出会う。この遭遇点から約100kbにわたり、2か所の終了領域がある[36][38]。大腸菌において一方はterE, D, Aが、他方はterC, A が集まっている領域。枯草菌ではterI とterII およびこのほかの2, 3の部位である[38]。各領域は終了させる複製フォークの方向が特異的に決まっている。独特なのは、複製フォークが対応するter配列まで行くのに、他方に対応している終了領域を通り過ぎることである[36]。この配置は複製フォークの待ち伏せを起こす。すなわち、何らかの理由で一方の複製フォークが遅れ、両フォークが本来の遭遇点で出会えなくても、早く進んできたほうがter領域で止まって到着を待つのだろう[39]。
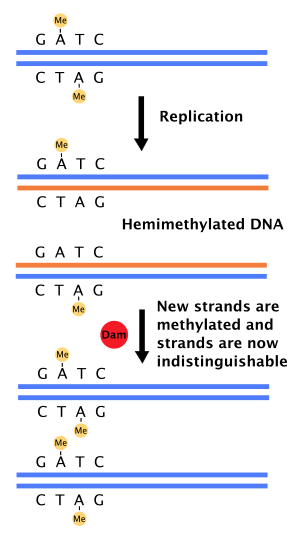
細胞周期にDNA複製がたった一回しか行われないためには複製起点に点火済みか否かを示す目印が必要となる。細菌の複製起点にはいくつかメチル化された配列が存在し、これらメチル化状態はDNA複製の前後で異なる。この違いが目印である。例えば大腸菌のoriC には11個のGATC-CTAGがあり、これはアデニンのN6位をメチル化するDamメチラーゼ (Dam methylase, Dam methyltransferase, DNA adenine methylase) の標的配列である。複製前は標的配列の両鎖ともメチル化されている。複製により、メチル化のない娘鎖が会合。結果、dsDNAの一方だけメチル化されたヘミメチル化DNA (hemimethylated DNA) となる[40]。ヘミメチル化は複製開始を阻害すると考えられている[40]。なぜなら、全くメチル化されていない複製起点は効率よく機能するため、複製開始に両鎖のメチル化が必要という考えが否定されているためである。ゲノム中のほかの典型的なGATC配列はどこにあろうと複製後1.5分以内にメチル化されるのに対し、複製起点のそれは約13分かかる[40]。このため、複製起点の標的配列は何らかの形で保護されていると思われる。
damメチラーゼをoriCから隔離する機構がseqA遺伝子の研究で明らかにされつつある[41]。
真正細菌の複製開始[編集]
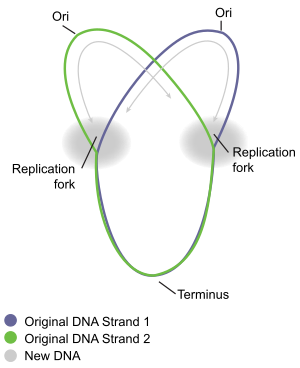

大腸菌の環状DNAは唯一の複製起点oriC から2つの方向にそれぞれ複製される。このいわゆる双方向性複製途中のDNAは、ギリシャ文字のθに見えることからシータ構造と呼ばれる。oriCの長さは245 bpで[42][43]、これは真正細菌一般の複製起点で共通しているようである。
oriCにおける複製開始の過程を示す。大腸菌にはTTATCCACAという共通配列が4つ存在し、このうち2つは残りの2つに対して逆を向く。これらをdnaAボックス dnaA box と呼び、遺伝子dnaA から発現する(dnaA産物:dnaA product である)DnaAが結合することからDNA複製は始まる。この状態をさらに詳しく述べると、親和性の高い5か所のdnaAボックスに5つ、次に親和性の低い部位に1つDnaAが結合し、これらがさらにオリゴマーを形成する。このオリゴマーは環状六量体である可能性が高く、親鎖はその外側に巻きつく[42]。複製開始の合図は、oriC にある3つの13bpの反復配列を融解させて開鎖複合体 open complex の形成を促す。そして、むき出しのssDNAにDnaCの補助でDnaBが結合する。DnaAの役割はDnaBをoriC に導くことであるが、これは開鎖複合体の出現というよりは、DnaAの直接の機能のようである。たとえば、R6Kと呼ばれるプラスミドにおいて、ヘアピンループの軸にoriC があり、DnaAの結合からDnaBが誘われる場合、二重らせんの融解は起こらない。
開鎖複合体の形成には少なくともほかにRNAポリメラーゼとHUタンパク質の2つが必須である。RNAポリメラーゼはoriC に隣接する領域にRNAを合成する。この短鎖は親鎖の一本に結合し、もともとの会合DNAに取って代わって塩基対を形成。こうして生じるDNAとRNAの部分的な二重らせんをRループと呼ぶ。一方、HUタンパク質は親鎖を屈曲させる[44]。Rループと屈曲の共存がoriC の融解を促進すると考えられている。
DnaAがDnaBを導くのと同様に、DnaBもまたプライマーゼであるDnaGをoriC に結合するよう促す。DnaBが来た開鎖複合体はその後、SSBが結合してプレプライミング複合体 prepriming complex という構造になる[42]。DnaGとほかのタンパク質が結合するのはこのssDNA領域が形成されたときである。DnaBとDnaGがそろい、プライモソームは完成。親dsDNAを解いて複製バブルを形成し、リーディング鎖のプライマーを合成する。この後、プライモソームは次の伸長段階を執り行う複製工場レプリソームの一部として働く。その役割は、第一にプライマーゼとして岡崎フラグメントのプライマー合成を繰り返すこと。第二に、DNAヘリカーゼとして親鎖を解き続けることである。プライモソームもレプリソームも複製バブルを拡張させつつ、そばに複製フォークを留める。
大腸菌ファージは宿主である大腸菌のタンパク質を拝借するとはいえ、プライマー合成の方法はファージの種類により大きく異なる。最初に発見されたM13ファージは宿主のRNAポリメラーゼをプライマーに利用する。しかし、ほかのファージや大腸菌自身はRNAポリメラーゼではなく、大腸菌DnaG遺伝子の産物であるDnaGを利用する。アーサー・コーンバーグ Arthur Kornberg によると、大腸菌や大半の大腸菌ファージにとってラギング鎖でのプライマー合成には少なくともほかにDNAヘリカーゼであるDnaBも必要であるようらしい。プライマー合成に必要なこれらのタンパク質群をプライモソーム(primosome)と呼ぶ。プライモソームは普通DnaGとDnaBの2つのみを指すが、プライモソームを形成するためにほかのタンパク質が必要な場合もある。
大腸菌のプライモソームは移動性を持つ。一本鎖DNA結合タンパク質に覆われていない、φX174ファージの環状DNA上を動きながらプライマー合成を繰り返すことができる。この性質は、岡崎フラグメントの合成を繰り返すラギング鎖合成に必要である。一方で、ただ一つの複製起点で済むリーディング鎖合成にはDnaBやRNAポリメラーゼの単独で十分である。
真正細菌の伸長段階[編集]
伸長段階の始まりは前段階におけるDNAクランプとDnaBのプレプライミング複合体への結合を引き金とする[42]。これらの相互作用はDnaAオリゴマー内でATP加水分解を起こす。すると、オリゴマーは分離するのでその複製起点からもう一度DNA複製が起こるのは防がれる。
真正細菌についての遺伝学的研究は大腸菌で際立って進んでいる。そこで、大腸菌において実際に伸長段階を担う複製装置であるレプリソームについて第一項で解説する。大腸菌ではDNA複製はまずリーディング複製から始まる。1000〜2000 ntが合成されてから次いで最初のラギング鎖合成へと続く。#伸長で前述したようにラギング鎖合成はリーディング鎖に比べ複雑である。その機構の精妙で興味深い特徴が大腸菌で発見された。トロンボーンモデルと名付けられたそれは第二項で紹介する。
大腸菌のレプリソーム[編集]

レプリソームとは、DNA複製伸長段階において複製フォークに形成される酵素の総称である。複数の複合体が集合し、1つの「工場」として機能していると考えられている。その詳細が最も明らかになっているのは大腸菌であるが、大腸菌における酵素の構成とそれらの協調的機能を紹介する。
レプリソームにおいて最も重要なのはやはりDNAポリメラーゼだろう。大腸菌においてこれを含み、実際にDNAを伸長させる複合体はDNA Pol IIIホロ酵素である。構成するタンパク質は2つのpol IIIコア酵素とγ複合体(クランプローダータンパク質)、さらにSSBと相互作用するχとφサブユニットである[45]。pol IIIコア酵素はDNAポリメラーゼたるpol III(αサブユニット)と3’→5’方向のDNA修復をするエキソヌクレアーゼ(εサブユニット)、θサブユニット、さらにDNAクランプであるβクランプ(βサブユニット二量体)で構成される[46]。一方、γ複合体はγ、δ、δ’および2つのτサブユニットから成る[45]。見た目から言えば、βクランプを構える5本のサブユニットと、その五本指の手のような巨大部位に伸びている細長いτサブユニットがある[47]。τタンパク質の先端はpol III(とDnaB)に結合し、一方、装着部位との連結鎖は柔軟である[47]。2つのpol IIIはそれぞれリーディング鎖とラギング鎖を担当するためγ複合体とつながっていてもある程度自由に動けなければならない。連結鎖の柔軟さはこのためにあるとされる。
各サブユニットの相互作用について説明する。まず、βクランプはDNAクランプの項で説明したようにpol IIIと結合する。レプリソームはさらにDnaB(DNAヘリカーゼ)[注釈 3]を含み、γ複合体と相互作用する。[29][46]。2本のτサブユニットはDnaBにも連結するために挟み込むためである。この連結はDnaBの移動速度を10倍に促進する[48]。次に、プライマーゼの相互作用はDnaBとの間で起こる。この場合、ほかの構成タンパク質と異なり、複製フォークへの結合は強固ではない。もともとプライマーゼの役割はSSBに覆われたssDNAに結合してプライマーを合成することであるが、このときにヘリカーゼとも結合する。その理由は、この結合が本来の仕事を1000倍に促進するためである[48]。仕事が済めばDNAからすぐに離れる。
真核生物の場合、大腸菌のように2つのDNAポリメラーゼによる複合体は形成しない。γ複合体にあたるクランプローダータンパク質(複製因子C : replication factor C:RFC)[49]は存在するが、リーディング鎖とラギング鎖の各DNAポリメラーゼは別々に働く。
トロンボーンモデル[編集]
トロンボーンモデルとは、大腸菌で発見されたラギング鎖合成の特徴的な様式を指す[29][50]。すなわち、ラギング鎖合成では親鎖の一部がループ構造を形成し、複製過程でこのループが演奏中のトロンボーンのように伸びたり縮んだりする。
進行方向が反対であるにもかかわらず、2つの親鎖は同じ速度で複製されるのは不思議なことである。絶えず複製を続けるリーディング鎖はともかく、ラギング鎖のpol IIIは複製作業を分散している。中断してはDNAから離れ、はるか遠くのプライマー-鋳型接合体に移動し、作業を再開しなければならない。これがリーディング鎖と同じペースというのは、解離から再結合までのタイムラグが一瞬でなければならないはず。不可思議なpol IIIのジャンプのカギはラギング鎖が成すループ構造とその根元を掴むレプリソームである。
前述したようにレプリソームはDNAへリカーゼを持つため、常に複製フォークに存在する。リーディング鎖、ラギング鎖担当のDNAポリメラーゼも含む。すなわち、レプリソームはラギング鎖において2カ所を掴む。1つはDNAへリカーゼを介した複製フォーク。ループの根元の1本はそこから分かれたばかりのssDNAである。もう1カ所はpol IIIにより複製中の部分である。ループ構造はこれら離れた2箇所の距離をなくす[51][52][50]。
2本の根元ssDNAは1本の親鎖であるが、流れる向きは異なり、どちらもループへと向かう。pol IIIの通過DNA領域とDnaBのそれを送り込むため、ループは大きくなっていく。このとき、γ複合体は開いたDNAクランプを準備している。また、岡崎フラグメントが伸長され始めてからしばらくすると、DNAヘリカーゼにプライマーゼが結合する。プライマーゼはループの中、すなわちpol IIIの複製方向と逆の位置に行き、プライマーRNAを置く。プライマーゼは離れ、やがてpol IIIは直前に伸長した岡崎フラグメントに到達する。DNAクランプの項で述べたように、pol IIIは既製の岡崎フラグメントに出会うと、親鎖(にはまったDNAクランプ)から離れる。レプリソームが根元の一つを放すことにより、ループは縮む。pol IIIは鋳型鎖から解離した後もレプリソームの一部として複製フォークに留まるので、次のプライマー-接合体へと素早く移動[51][52]。そこにγ複合体は用意していたDNAクランプをはめ、pol IIIはこれに結合する。ラギング鎖合成ではこれが繰り返される。
接合[編集]

接合においてDNA複製が利用される。接合とは細菌の生殖様式の一つで、一つの個体(供与菌、F+)が別の個体(受容菌、F-)に自身の(染色体やプラスミドの)DNAを移動させることである。その典型例は大腸菌のエピソーム[注釈 4]であるF因子(Fプラスミド)の仲介で起こる。すなわち、このF因子を、持つ供与菌が持たない受容菌に移す。
接合ではF因子(とそれと地続きの染色体)の巻き戻りが起こり、一方のssDNAは受容菌へと移動。もう片方は供与菌に残る。DNA複製はここで登場し、供与菌と受容菌両方のssDNAを正常なdsDNAに変換する。
具体的な過程を示す。接合には伝達領域と呼ばれるF因子の大きな(約33 kb)領域が必要で、伝達領域の一端にある伝達起点oriTから始まる。リラクセーズのTraIはoriTを認識し、nicと呼ばれる部位を切断する[53][54]。生じた5’末端に共有結合し、約200bpにわたりdsDNAを巻き戻す[53][54]。巻き戻しはTraIが5’末端から環に沿って移動しながら行う。遊離した5’末端は受容菌へと移動し(このとき供与菌と受容菌は接触し、つながっている)、次々と巻き戻されるssDNAを先導する。F因子がプラスミド型の場合、巻き戻しは環全体にわたり、(一本鎖の)F因子丸々1個が受容菌に伝達される。一方、染色体に組み込まれている場合、DNA伝達は伝達領域とは逆方向へ進む[53][54]。プラスミド型と同様に5’末端から受容菌へと入っていくが、これは細菌間の接触が壊れるまで続く。細菌の染色体全体が伝達するのにおよそ100分かかるが、通常はその前に接合は中断する[53][54]。
接合によって互いのF因子または染色体は一本鎖になるが、それはDNA複製によって二重らせんに戻る。DNA複製が巻き戻しと同時に起こっているなら、ウイルスのDNA複製で紹介するローリングサークル型複製に似ている。しかし、伝達は複製と全く独立した過程であり、これはローリングサークル型複製とは言えない。
真核生物のDNA複製[編集]
真核生物のDNA複製機構は基礎を真正細菌と同じにしながら、それよりもはるかに複雑となっている。その大きな特徴の一つはまず、レプリコンがゲノム中に多数点在することである。理由はいくつかあり、第一にゲノムサイズが著しく巨大である。第二に、例えば大腸菌のゲノムは1本の染色体で十全だが、人間の場合23対も存在する。最後に、ほとんどの原核生物は染色体が環状であるのに対し、ほとんどの真核生物は線状であること。これは#末端複製問題で説明したような問題を引き起こす。
真核生物のレプリコン[編集]
真核生物のレプリコンは比較的短く、酵母やショウジョウバエで約40kb、動物細胞では約100kbである[55][56]。ただし、この大きさは同一ゲノム内でも10倍以上のばらつきがある[55][56]。真核生物の複製速度は約2000bp/分であり、約50,000bp/分の細菌と比べるとずっと遅い[55][56]。また、真核生物には複製停止点がなく[55][56]、隣の複製フォークとの衝突によりその複製部位でのDNA複製は完了する[11]。
真核細胞では、DNA複製は細胞周期におけるS期(S phase)にのみ起きる。S期は最初のレプリコン点火から始まり、典型的な哺乳類の体細胞ではS期が6時間以上続く[55][38]。
複製プログラム[編集]
複製プログラムとは、真核生物のDNA複製においてレプリコンが点火される順番である。全てのレプリコンが一度に点火されないことには例外があり、ショウジョウバエの初期胚の核分裂では多数のレプリコンが同時に点火され、S期は短縮されている[56]。
染色体には「初期に複製する領域」と「後期に複製する領域」とがある[55][57]。また、ブロモデオキシウリジンで複製フォークを標識し、抗体で染色して観察すると、染色が集中した「フォーカス」が染色体あたり100〜300観察される[55][57]。このフォーカスはおよそ300以上の複製フォークを含む。これらのことから、ある時期にレプリコンの一群が一斉に点火されてそのDNA複製は局所的に制御されると考えられている。
複製プログラムの実態は複製開始因子が複製起点に作用する順番であり、クロマチン構造や核における三次元的配置といったエピジェネティクスな制御により各領域への複製開始因子の接近のしやすさを調節することで複製プログラムは制御されている[58]。出芽酵母において複製開始因子のいくつかは複製起点の数よりも少なく、上述のように全てのレプリコンの同時点火は起こらず、また、複製開始因子の複製起点への結合がDNA複製の律速段階である。Saccharomyces cerevisiaeを用いた研究から、S. cerevisiaeのヒストン脱アセチル化酵素のRpd3は初期および後期の複製起点の両方の複製開始を抑制し、Sirtuinファミリー(NAD+依存性ヒストン脱アセチル化酵素)のSir2は初期の複製起点の複製開始を促進することが明らかとなった[59]。
限られた複製因子を取り合うために、染色体外のリボソームRNAをコードしている多コピーのリボソームDNA (rDNA) 領域と単一コピー領域のDNA複製は競合しており、Sir2がDNA複製される活性なrDNAの複製起点の分布を低く抑えることにより正常なDNA複製が行われる[59]。S. cerevisiaeの複製開始点の約30%が、ヒトの場合は50%がrDNA領域といった多コピーの繰り返し配列に存在する。S. cerevisiaeのrDNAには150~200コピーの繰り返し配列があり、各繰り返し配列に複製起点があるが、1回のS期においてDNA複製される活性な複製起点はそのうち20%に過ぎない。rDNAの活性な複製起点は転写が活性な遺伝子の下流に存在する。また、rDNAにおける活性な複製起点の分布はSir2により決定されている。rDNAにおいて隣接する3~4個の活性な複製起点の集団が形成されており、各集団はSir2によるヒストン脱アセチル化により不活性化された領域で隔てられている。
Sir2がrDNA領域の複製における負の制御効果を持つのに対し、Rpd3は正の制御効果を持つ。吉田和真は、複製プログラムの制御においてヒストン脱アセチル化酵素の各複製起点への作用よりもむしろrDNA領域の複製開始点の活性の操作の重要性が大きいとする説を提唱した[59]。
複製プログラムは、細胞の系統や分化および発生の過程に応じて柔軟に変化する[60]。複製プログラムの制御の生理的意義については、DNA複製を転写や修復といった染色体の他の機能と協調させることが遺伝情報の継承において重要であると考えられている[61]。
休眠複製起点[編集]
真核生物の染色体には、正常なDNA複製で点火される数以上の余剰な複製起点(休眠複製起点)が存在し、複製フォークの進行が阻害されてフォークが停止した場合に、複製を完了させるためにフォークが到達できなかった領域にある休眠複製起点で点火される。休眠複製起点は、通常の複製起点と同様にG1期にライセンシングを受ける。マウスにおける実験から、外来のストレスがない状態であっても複製フォークの停止は多数引き起こされ、ライセンシングを受けた休眠複製起点の数が低く抑えられた個体において停止複製フォークが蓄積することが明らかとなった[62]。停止複製フォークの蓄積は複製後染色体の不分離の原因となり、がんを引き起こす。休眠複製起点の抑制は、マウスにおいてMCM六量体のドメインのひとつであるMCM4をコードするMcm4変異体(345番目のフェニルアラニンがイソロイシンに変換された点突然変異)で観察することができる。Mcm4変異体において、MCM六量体に2つの補因子CDC45とGINSが結合したCMG複合体の量が減少し、野生型と比べて分子量が低いものと同じものの両方が観察された。変異型CMG複合体のヘリカーゼ活性は野生型と同等であり、Mcm4変異は正常なMCM六量体の形成を減少させることでライセンシングされた休眠複製起点の数を減らすと考えられている。
休眠複製基点は停止複製フォークのレスキューの主要な手段であるが、染色体上に均一に分布しておらず、休眠複製基点がほとんどない脆弱部位が存在する[63][64]。脆弱部位では、停止複製フォークのレスキューにおいて相同組換えなどによる複製フォークの再始動が重要となる。脆弱部位をはじめとする染色体のさまざまな部位ではストレスによって不完全なDNA複製が起こり、このことは染色体の再編成 (gross chromosomal rearrangements:GCR)、遺伝子増幅 (copy number variation:CNV)、遺伝子欠失の原因となる[63][65]。
複製焦点[編集]
脊椎動物などの真核細胞において、複数の、複製が行われる配列(複製部位)は特定の場所に集まることが知られており、その場所を複製焦点と呼ぶ[66]。複製部位は、免疫染色による娘鎖または複製酵素の、もしくはGFPタグによる複製因子のモニタリングにより観測できるが、それらの実験によって、複製が行われるS期において大きさや位置が様々な複製焦点が、複製フォークの総数よりもはるかに小さな数で存在することが明らかとなった。
出芽酵母の複製焦点を追跡したArmelle Lengronneらの研究によると、複製起点はG1およびS期の細胞において常に移動しており、またG1期からS期への移行時にそのダイナミクスが減少する[67]。このため、複製起点は、ラミンタンパク質といった、クロマチンの立体構造を形成するタンパク質に結合するなりして核内の特定の位置に固定されているわけではなく(出芽酵母はラミンタンパク質を持たない)、またS期に入ると複製起点は自己集合して複製焦点を形成することが示唆された。
複製焦点の形成は、複製起点の点火が空間的にも時間的にも調節されていることにより引き起こされる。Drew M. Pardollらは、哺乳動物の細胞において隣接する複製起点は同時に点火されることを発見した[68]。複製部位が空間的に並列することにより複製フォークの密集化(クラスター化)がもたらされる。クラスター化は、隣接複製起点の一方が点火前に何らかの原因で巻き戻されて点火されなくなる、もしくは複製フォークの進行が阻害されて停止したとき(原因として、転写装置などのDNAと強く結合しているタンパク質との衝突、伸長反応の基質であるdNTPの枯渇、DNA損傷による妨害などがある)に、複製に失敗した領域をもう一方からの複製フォークがすぐに複製に再挑戦するようにするためと考えられている(停止複製フォークのレスキュー)。停止複製フォークのレスキューの機構には、正常なDNA複製には点火されない休眠複製起点の利用もある。
真核生物の複製開始[編集]


真核生物におけるDNA複製のモデル生物は酵母である。複製開始が行われる領域は自立複製配列 (autonomously replicating sequence:ARS) であり、そこには複製開始点複製エレメント (origin replication element:ORE) が存在する。この11塩基対[70]にタンパク質が結合し、複製開始点認識複合体 (origin recognition complex:ORC) は形成される。ORCに相当するイニシエーター-DNA複合体は、調べられた限りすべての真核生物に共通する[70]。OREのすぐ隣はDNA開裂領域 (DNA unwinding element:DUE) である。約80塩基対のこの配列は、容易に分解するようAとTに富む[70]。DUEは酵母における複製開始点であり、複製開始と伸長に関わるMCMタンパク質複合体が結合する。
真核生物の染色体上には複製起点が多数存在するが、全て細胞周期一回あたり一度しか複製が開始しないように調節されており、これを複製のライセンシングと呼ぶ。複製のライセンシングが破綻すると、ゲノムの一部が一度の細胞周期に2度複製される、また逆に複製されないなどの問題が生じる。
ライセシングの過程はG1期(S期の前)からS期にかけて起こる。ARSのレプリケーターにイニシエーターである複製起点認識複合体(Origin Recognition Complex:ORC。オークと読む)が結合することが複製開始の引き金である。ここで注目すべき、原核生物にはないライセシングの特徴は、レプリケーターとイニシエーターの結合が複製起点の点火と別である点である。
ライセシングの前にまず複製開始と伸長の機構を詳述する。複製起点点火前、G1期におけるARSとORCとの結合は複製前複合体 (prereplicative complexes:pre-RC) の形成に続く。すなわち、pre-RCはORC複合体を前身とし、4種類のタンパク質から構築される。まず、ORCの結合から2種類のヘリカーゼ装着タンパク質(Cdc6とCdt1)が引き寄せられる[71][72]。ORCと装着タンパク質が協力して複製フォークヘリカーゼを呼び、pre-RCは完成。このヘリカーゼはMcm2から7の6つのタンパク質による複合体[72]だが、これはdsDNAを囲むだけであり、巻き戻しやDNAポリメラーゼの導入には直接結びつかない[71]。しかし、G1期に生じたpre-RCは次のS期で複製の出発点となる。
伸長段階はS期に入って2種類のキナーゼがpre-RCを活性化してから開始される。サイクリン依存性キナーゼ (cyclin-dependent kinase, Cdk) とDbf4依存キナーゼ (Dbf4-dependent kinase, Ddk) はS期に入ると活性化し、pre-RCやほかの複製タンパク質をリン酸化する。するとさらに多くのタンパク質が複製起点に集まり、伸長段階へ移行する。これには3種類のDNAポリメラーゼとその補助因子が含まれ、ポリメラーゼ類は決まった順序で結合する。最初がDNA Pol δとPol εで、次にDNA Pol α/プライマーゼである[71]。実際に伸長が始まるのはDNAポリメラーゼαが結合してからで、その前にδとεが来ることで複製に関わる全てのDNAポリメラーゼを伸長前に確実にそろえることができる。集合したタンパク質のうち、DNAポリメラーゼやその招集に関わった因子の多く、Mcm複合体は複製装置として複製フォークに留まる。Cdc6やCdt1といったその他の因子は伸長段階が始まるころには解離したり破壊されたりする。
複製開始を概観してきたが、ライセシングの正体は以下に述べる調節機構である。これまで述べたように、真核生物の複製はその前にpre-RCの形成とCdkの活性化を必要とする。Cdkは既存のpre-RCの活性化のほかに実は新たなpre-RCの形成を阻害する働きも持つ[71]。すなわち、ORC複合体にほかの成分が結合することを防ぐ。Cdkの活性化レベルはG1期に低く、それ以外の細胞周期上の時期には高い<watson241>。したがって、pre-RCが形成される機会はG1期にしかない。同様に、pre-RCの活性化が起き得るのも直後のS期しかない。Mcm複合体の制御もライセシングの一端であると予想されている。Mcm複合体はDNA複製が進行すると共にゲノムDNAから順次剥がれてゆき、次のM期の終わりになるまでARSに結合しない。この説を支持する証拠の一つとして、Geminin(S期にMCM複合体が複製起点に結合しないように制御しているタンパク質の1つ)の発現を抑制するとゲノムDNAの一部の複製が重複する事が報告されている[要出典]。
また、ARSには早期に複製が開始されるものとS期の後半に複製が開始されるものとにわかれる。出芽酵母をモデルとした研究からは細胞周期のチェックポイントをつかさどるタンパク質群は、DNA障害などの異常を検知すると、後半に複製が開始されるARSからの複製開始反応をとめることで、DNA修復が終了するまで複製反応が起こるまでの時間稼ぎをおこなうことが知られている。
真核生物の伸長段階[編集]

真核生物の場合、伸長段階にかかわる酵素のいくつかは巨大な複合体(複製工場)を形成するものの、すべての酵素が複製フォークに集まるわけではないらしい。真正細菌のようにリーディング鎖とラギング鎖のDNAポリメラーゼはつながっていない。真核生物のDNAポリメラーゼは真正細菌と比べて種類が多く、また娘DNAの合成に直接かかわるDNAポリメラーゼの種類は複数存在することが確認されている。DNAポリメラーゼαはDNAプライマーゼのサブユニットを含んでおりプライマーの合成を行う。DNAポリメラーゼδはリーディング鎖の、DNAポリメラーゼεはラギング鎖の複製を行う。
ヘリカーゼがほどいたssDNAは、一本鎖DNA結合タンパク質である複製タンパク質Aが安定化させている[72]。まずDNAポリメラーゼαがプライマーを合成し、それにデオキシヌクレオチドを20 bp付加した後、クランプローダータンパク質である複製因子C (replication factor C:RFC) がDNAポリメラーゼαをDNAから移動させて、替わりにDNAクランプの増殖細胞核抗原 (PCNA) を引き寄せる。PCNAは、デオキシヌクレオチドの付加反応の連続反応性がより大きいDNAポリメラーゼδを誘導し、そこから先はδが本格的に複製を進める。PCNAがDNAからDNAポリメラーゼαを除去してDNAポリメラーゼδをDNAに結合させることをポリメラーゼ交代という[72]。
真核生物のラギング鎖における岡崎フラグメントは約200 bpの間隔で合成されることが知られており、伸長反応の開始にPCNAは其の間隔でDNAに付加されると考えられている[73]。DNAポリメラーゼδが隣接岡崎フラグメントまで伸長反応を完了させると、岡崎フラグメントの除去(岡崎フラグメントのプロセシング)され、PCNAはElg1複合体によりDNAから解離する。DNAに結合したPCNAはSUMO化(SUMOylation)されることが知られており、未修飾およびSUMO化されたPCNAの両方を標的とする。特にSUMO化されたPCNAに好んで結合し、よりよく標的とする。また、人工的に合成したDNAを用いたin vitroの系においてElg1複合体はPCNA解離活性を示さなかったが[74]、in vivoにおけるDNAの存在形態であるクロマチンを導入したin vitroにおいてPCNA解離活性が現れたため、Elg1複合体の活性はヌクレオチドにおいて発揮されることがわかっている[73]。ただし、出芽酵母の細胞においてElg1複合体非存在下でもPCNAは最終的にクロマチンから除去されるため、代替としてPCNAを除去する機構が存在することが示唆されている。
真核生物は、Elg1複合体、Rad24複合体、Ctf18複合体の3種類の複製因子様複合体 (RFC-like complex) を持っている[73]。RFCは大きなサブユニットであるRfc1と小さなRfc2~5から構成されるが、複製因子様複合体もRfc2~5を含み、Rfc1の代わりにそれぞれElg1、Rad24、またはCtf18を持つ。Rad24複合体は、PCNA様複合体9-1-1をDNA損傷部位に誘導する働きがあるとされている。Ctf18複合体はin vitroにおいてPCNAをDNAに誘導およびDNAから除去する活性があるが、それらの活性はin vivoにおける主な機能ではないことが知られており、正しい機能は明らかとなっていない[75]。
#ニックトランスレーションにおけるプライマー除去は真正細菌と異なる過程を経る。プライマー除去には5’→3’のエキソヌクレアーゼが必要だが、真正細菌と異なり真核生物でそれを担うのはDNAポリメラーゼではない。中心的な役割を果たすのはフラップエンドヌクレアーゼであるFEN1(Flap structure-specific endonuclease 1、以前はMF1と呼ばれていた)である[49]。これは岡崎フラグメントの3‘末端でDNAポリメラーゼδ複合体に結合し、その隣接プライマーを分解する。ただし、分解活性はプライマー5’最末端部のリボヌクレオチドにある三リン酸基により阻害される。これを真核生物がどのように乗り越えるかはまだはっきりと判明していない。
実際のプライマー除去機構には様々な仮説が考えられている。その一つは、プライマーの大部分はFEN1ではなくRNアーゼHによって除去されるというものである[49]。RNアーゼHはRNA間のホスホジエステル結合を切断できるが、RNA-DNAのそれはできないという特徴を持つ。そのため、少なくともDNAと隣接する最後のプライマーRNAは残ってしまうはずである。ここで、ホスホジエステル結合切断から生じた5‘末端は三リン酸ではなく一リン酸基なので除去作業はFEN1が引き継ぐ。しかし、RNアーゼを持たない細胞でもラギング鎖複製が行われることが確認された[49]。もう一つの仮説では、ヘリカーゼがプライマーと親鎖間の塩基対を切断し、はがれた部分(フラップ flap )をDNAポリメラーゼδが隣の岡崎フラグメントから伸長して補う[76]。フラップはFEN1が切断する。
細胞小器官のDNA複製[編集]
ここでは、細胞小器官(オルガネラ)のDNA複製 (organelle DNA replication:ODR) について記述する。
置き換え型複製[編集]

動物のミトコンドリア や植物の葉緑体などの細胞小器官(オルガネラ)にある小さな環状DNAでは、置き換え型複製と呼ばれる特殊なDNA複製が観察される[77][78]。まずRNAポリメラーゼが、二本のDNAの一方(H鎖、heavy strand)の複製起点に相補的なRNAを合成する。複製されるH鎖の領域と娘鎖は新しい二重らせんを形成するため、もともとH鎖と二重らせんを形成していたもう一方のDNA鎖(L鎖、light strand)は一本鎖の状態になる。この、3本のDNAが現れた領域をエンドヌクレアーゼが認識してRNAを切断し、プライマーとする[78]。ここから娘鎖の伸長は始まり、H鎖の塩基対の相手がL鎖からRNAへと置き換わっていく。この領域をDループ(置換ループ displacement loop。tRNAのDループとは関係ない)と呼ぶ。
L鎖は独自にプライマーを付加され、遅れて複製される。哺乳類のミトコンドリアDNAの場合、Dループの拡大がH鎖の3分の2まで進んだ時に、L鎖の複製は始まる[78]。L鎖の複製起点が一本鎖となり、露出するためである。H鎖の複製が完了するとL鎖が完全に外れて追い出される。この時点ではL鎖の複製は3分の1までだが、遅れながらも完了する[78]。
レトログレードシグナル[編集]
細胞分裂におけるミトコンドリアおよび葉緑体の分裂機構に研究において、単細胞紅藻シゾンCyanidioschyzon merolaeがモデル生物としてよく用いられている。C. merolaeにおける研究から、ミトコンドリアと葉緑体のDNA複製は核のそれより先んじて行われ、オルガネラにおけるDNA複製の開始はG1期/S期に特異的なサイクリン依存性キナーゼCDKA (cyclin-dependent kinase A) を活性化し、これが核の複製を開始させることが明らかとなっている[79]。この知見はオルガネラの複製開始をCDKAの活性化につなげるシグナル伝達の存在を示唆する。このような、オルガネラから核へのシグナル伝達をレトログレードシグナルという。
C. merolaeにおいて、葉緑体から発信されてCDKAの活性化を引き起こすレトログレードシグナルのシグナル分子は、テトラピロールの一種であるMg-プロトポルフィリンIX (Mg-protoporphyrin IX:Mg-ProtoIX) である[80]。また、Mg-プロトポルフィリンIXのレトログレードシグナルはタバコの培養細胞BY-2においても確認されているため、このシグナルは高等植物一般である可能性がある。葉緑体のDNA複製が行われると、葉緑体で合成されているMg-プロトポルフィリンIXが葉緑体外に放出されて細胞質に蓄積し、サイクリン1 (cyclin 1) の細胞内濃度を増大させる。サイクリン1がCDKAと結合すると、CDKAを活性化させる。
S期以外の細胞周期においてサイクリン1はポリユビキチン化され、ポリユビキチン化したタンパク質を標的とするプロテオソームによりサイクリン1が分解されている。サイクリン1のポリユビキチン化を行う酵素(ユビキチンリガーゼ)はSCF複合体であるとされている。SCF複合体はSkp1、Cullin 1、Fボックスタンパク質から構成され、標的の認識はFボックスタンパク質が行う。C. merolaeにおける典型的なFボックスタンパク質はFbx1~4の4種類があるが、サイクリン1を認識するFボックスタンパク質はFbx3である。Mg-プロトポルフィリンIXはFbx3との結合能を有しており、Mg-プロトポルフィリンIXがFbx3と結合することによりサイクリン1の結合を競合阻害すると考えられている[80]。
古細菌のDNA複製[編集]
古細菌のDNA複製については全貌が明らかになっていない。研究ではもっぱらスルフォロブス属のSulfolobus solfataricus P2などを用いる。知見の多くは、真正細菌か真核生物の複製に関わるDNA配列やタンパク質と相同な古細菌のそれから推測されている。類似遺伝子の探索では複製起点を発見することができなかったが、古細菌ゲノムの領域ごとにヌクレオチドの出現頻度を統計する方法により、ピュロコックス属であるPyrococcus abyssi の複製起点が断定された[12]。古細菌のDNA複製は真核生物寄りの複製機構を基本に、真正細菌的な要素が一部混合するようである。
古細菌の伸長段階で働くタンパク質の多くは、真核生物の当該タンパク質のアミノ酸配列にも遺伝子にもよく類似している。特に、RFCやPCNは相同タンパク質が存在する[12]。また、メタン生成古細菌の3種を除いて、現在までに配列決定されたすべての古細菌ゲノムは、少なくとも、Orc1とCdc6の両方に相同性を有する遺伝子を1つ含んでいる[81]。古細菌のDNAポリメラーゼは、デオキシヌクレオチドを合成するサブユニットが真核生物のDNAポリメラーゼδのそれと類似性を示す[12]。一方、DNA複製中に行われる校正修復を担うタンパク質は、大腸菌のDNAポリメラーゼIIIのεサブユニットと相同であるとされる[12]。
ウイルスのDNA複製[編集]
DNAウイルスの多くは、宿主のDNA複製にかかわるタンパク質を使って複製する。ヘルペスウイルス科、アデノウイルス科、パポバウイルス科、パルボウイルス科などのDNAウイルスは核内でDNAを複製するが、天然痘ウイルスを代表とするポックスウイルス科では細胞質で複製をする
鎖置換[編集]
ウイルスの中には線状ゲノムを末端から複製するという珍しい例が存在する。代表的なのはアデノウイルスとφ29ファージにおける鎖置換である[82]。両3'末端からそれぞれ一本の娘鎖が合成されるが、これは同時期ではない。すなわち、一度の複製フォーク出発に1つのDNAポリメラーゼしか伴わず、別時期のリーディング鎖合成が2回行われる。ほかの生物ならラギング鎖が合成されるだろう5'→3'の親鎖は複製が進み、遊離してもssDNAのまま放置。複製が反対側の末端に到達すると、完全に塩基対が置き換えられた親ssDNAは遊離する。このssDNAも独自に複製されるが、そのためには短くとも3'末端に塩基対が作られ、複製起点が二重らせんであることが必要である。
鎖置換を複製機構とするいくつかのウイルスは、それぞれの5'末端に末端タンパク質が共有結合している[82]。例えば、アデノウイルスではセリンがホスホジエステル結合でつながっている。末端タンパク質には、プライマーとなるヌクレオチドのシチジンを持つことと、DNAポリメラーゼと会合するという2つの役割がある[82]。このことから次のモデルが考えられている。末端タンパク質とDNAポリメラーゼが複合体を形成し、これがDNA末端に結合するというものである。次いでシチジンから娘が伸長されるのだろう。この共有結合は複製後も取り残されると考えられており、実際アデノウイルスの5’末端に前回使用されたままのセリンが観察される。これは次の複製開始まで放置され、複製のときに新しい末端タンパク質と置き換わる。
末端タンパク質はDNA末端から9〜18 bpの間に陣取る[82]。隣の17〜48 bpの領域は、複製開始に必要な宿主由来の核因子I (nuclear factor I:NF I) の結合に不可欠である[82]。したがって、複製開始複合体はDNA末端から9〜18 bpの間で形成される[82]。
ローリングサークル型複製[編集]

一部の環状DNAはローリングサークル型複製と呼ばれる特殊な機構で複製される[83]。一般的に、1本鎖環状DNAをゲノムとするファージが行う[84]。
大腸菌ファージφX174の場合を紹介する。複製が開始されたとき、DNAは二重らせんとなる。このときの二本鎖DNA状態を複製型 (replicative form:RF I) と呼ぶ。複製型のうち、もともとのゲノムを(+)鎖、新しく合成された方を(-)鎖に呼び分ける[83][84]。まず、エンドヌクレアーゼのAタンパク質が (+)鎖の複製起点にニック(切れ目)を入れる。この後、Aタンパク質はニックの5'末端に残る。このように、dsDNAにニックを入れ、生じた5‘末端に結合する酵素をリラクセーズ(弛緩酵素、relaxase)と呼ぶ[84]。さて、ニックの3’末端は(+)鎖伸長のためのプライマーとなり、(-)鎖を鋳型として新たな娘ssDNAが合成されていく。それに追い出されるように、対岸の5’末端側は伸長に連れてどんどん(-)鎖から離れる(この(+)鎖のssDNAをテールと呼ぶ)。やがて娘鎖の伸長は一周して複製起点に到達する。このとき、娘鎖は親鎖と同じ長さ、すなわち(+)鎖全体がテールとなるが、テール末端のAタンパク質は再び複製起点を認識して(+)鎖を娘鎖から切り離す。実はAタンパク質は5'末端と同時に3'末端にも連結しており、複製フォークが複製起点を過ぎるころ、すなわちちょうど一周した時にはAタンパク質も複製起点近くに存在する。(-)鎖からも離れ、遊離した(+)鎖は環状となり、ゲノムDNAは複製される。娘鎖と(-)鎖の二本鎖はその後も複製型DNAとして使い回され、同じ方法で複製は続いて(+)鎖のコピーが多数生成される。ローリングサークル型複製の名前は、娘鎖の伸長の際に二本鎖部分が反時計回りに回転し、(+)鎖が引き出されているように見えることから名づけられた。この様子は、まるでトイレットペーパーのロールが床に転がってほどけるようである。ギリシャ文字のσにも似ており、ローリングサークル型複製はσ型複製とも呼ばれる。
次にλファージの場合を紹介する。λファージはローリングサークル型複製を二本鎖DNAの複製に利用する。DNA複製の初期段階では、θ型の複製(前項#複製の機構で解説されている通常の方式に則った、環状DNAの複製)により環状DNAのコピーがいくつか生じる。しかし、ここで作られる環状DNAをλファージは頭部に取り込むことができない。そこで、これらを鋳型にしてローリングサークル型複製を行い、直鎖DNAが作られる[83]。このときのローリングサークル型複製は半不連続的である。鋳型の環状DNAから直接複製されたDNAはリーディング鎖として連続的に伸長し、鋳型の数倍の長さにまでなる[83]。そのリーディング鎖を鋳型に、ラギング鎖としてさらにDNA断片が合成されていく。こうして新生された直鎖dsDNAをコンカテマー (concatemer) と呼ぶ。コンカテマーは1ゲノム分に切り出され、二本鎖の娘鎖がファージ頭部に導入される。
人工的なDNA(断片)複製方法[編集]
- PCR法など
突然変異[編集]
複製は極めて高い精度で行われるが、それでも程度の確率で合成ミスが生じる。その結果、DNAの塩基置換が起こり、突然変異が起こる。このような複製ミスによる突然変異のほかに、紫外線や化学物質によってDNAが損傷し、突然変異が生じることがある。
注釈[編集]
- ^ a b DNAはデオキシヌクレオチドが、RNAはリボヌクレオチドが重合した高分子である。デオキシヌクレオチドの一部に塩基という部位があり、アデニン、チミン、グアニン、シトシン の4種類が存在する。各種には「相補的な」と形容される特別な関係にある塩基を一つ持つ。相補的な塩基の一組は特異的に結合し、ほかの塩基とは結合しない。dsDNAは相補的な塩基配列の組み合わせで成り立つ。
- ^ SSBは主に次の2つの様式でDNAと結合する。1) リボース‐リン酸主鎖への静電相互作用 2) DNA塩基との積み重ね相互作用。塩基とはほとんど水素結合を作らない。
- ^ 大腸菌のヘリカーゼはDnaBだけではなく、最新データ(2007年現在)では11種類あるとされる。これは、巻き戻しはDNA複製の時だけでなく、転写、組み換え、DNA修復といった様々な過程に必要であるためである。
- ^ 独立して存在することも、染色体に組み込まれることもできるプラスミド。いずれの状態でも大腸菌のそれは接合において個体間を移動する。供与菌のF因子がプラスミド型の場合、それを移し、F-はF+へと変わる。組み込まれている場合、染色体の一部または全てが伝達される。
出典[編集]
- ^ a b c d e f 『レーニンジャーの新生化学〔下〕』、著者:David L. Nelson, Michel M. Cox、監修:山科郁男、2007、p1362
- ^ 『ウィーバー分子生物学第4版』、p704
- ^ 『分子細胞生物学第5版』、東京化学同人、著者:Harvey Lodish, Paul Matsudaira, Monty Krieger, Arnold Berk, Chris A. Kaiser、訳者:石浦章一・須藤和夫・丸山工作・石川統・野田春彦、2005、p114
- ^ 『ウィーバー分子生物学第4版』、p707
- ^ a b c d 『分子細胞生物学第5版』、p115
- ^ 『レーニンジャーの新生化学〔下〕』、p1363
- ^ 『遺伝子第8版』、p329
- ^ 『ワトソン遺伝子の分子生物学第6版』、著者:James D. Watson, Tania A. Baker, Stephen P. Bell, Alexander Gann, Michael Levine, Richard Losick、監訳者:中村桂子、発行:学校法人東京電機大学出版局、2010、p231
- ^ a b c 『ウィーバー分子生物学第4版』、p712
- ^ a b c d 『ワトソン遺伝子の分子生物学第6版』、p225
- ^ a b “Recombination-restarted replication makes inverted chromosome fusions at inverted repeats”. 2014年7月5日閲覧。. Mizuno Kenichi, Miyabe Izumi, Stephanie A. Schalbetter, Antony M. Carr, Johanne M. Murray. Nature, 493, 246-249 (2013)
- ^ a b c d e f g 『ゲノム 第3版-新しい生命情報システムへのアプローチ』、著者:T. A. ブラウン、監訳者:村松正實(まさみ)、小南凌(りょう)、発行:株式会社メディカル・サイエンス・インターナショナル、2007年、p495
- ^ a b 『ワトソン遺伝子の分子生物学第6版』、p237
- ^ a b “In and out of the Replication Factory”. 2014年7月2日閲覧。. Peter Meister, Angela Taddei, Susan M. Gasser (2006). Cell Volume 125, Issue 7, 30 June 2006, pp.1233–1235
- ^ 『遺伝子第8版』、著者:Benjamin Lewin、訳者:菊池韶彦(あきひこ)、東京化学同人、2006、p361
- ^ 『ウィーバー 分子生物学』、p721
- ^ 『ワトソン遺伝子の分子生物学第6版』、p216
- ^ a b 『ウィーバー分子生物学第4版』、p710
- ^ 『ウィーバー分子生物学第4版』、p711
- ^ 『分子細胞生物学第5版』、p118
- ^ a b 『ストライヤー生化学(第6版)』、東京化学同人、著者:Lubert Stryer, Jeremy M. Berg, John L. Tymoczko、監訳者:入村達郎・岡山博人・清水孝雄、2008、pp775
- ^ 。PDB: 1W60; Kontopidis G, Wu SY, Zheleva DI, Taylor P, McInnes C, Lane DP, Fischer PM, Walkinshaw MD (February 2005). “Structural and biochemical studies of human proliferating cell nuclear antigen complexes provide a rationale for cyclin association and inhibitor design”. Proc. Natl. Acad. Sci. U.S.A. 102 (6): 1871–6. doi:10.1073/pnas.0406540102. PMC 548533. PMID 15681588.
- ^ a b 『ワトソン遺伝子の分子生物学第6版』、p221
- ^ a b 『ワトソン遺伝子の分子生物学第6版』、p220
- ^ 『ウィーバー 分子生物学』、p759
- ^ 『ウィーバー分子生物学第4版』、p761
- ^ a b c 『ワトソン遺伝子の分子生物学第6版』、p222
- ^ a b “The Replicase Sliding Clamp Dynamically Accumulates behind Progressing Replication Forks in Bacillus subtilis Cells”. 2014年7月2日閲覧。. Masayuki Su'etsugu, Jeff Errington, Molecular Cell, Volume 41, Issue 6, March 18 2011, pp.720–732
- ^ a b c 『ワトソン遺伝子の分子生物学第6版』、p226
- ^ 『ストライヤー生化学(第6版)』、p783
- ^ 『レーニンジャーの新生化学〔下〕』、p1358
- ^ 『遺伝子第8版』、p358
- ^ a b c d 『ワトソン遺伝子の分子生物学第6版』、p211
- ^ 『レーニンジャーの新生化学〔下〕』、p1370。ただし、カテナン発生のメカニズムについては「まだよくわかっていない機構」と表記。
- ^ a b c d 『ウィーバー分子生物学第4版』、p773
- ^ a b c d 『エッセンシャル遺伝子第1版』、著者:Benjamin Lewin、訳者:菊池韶彦・榊佳之・水野猛・伊庭英夫・紅順子、発行:東京化学同人、2007、p248
- ^ a b 『遺伝子第8版』、p327
- ^ a b c 『遺伝子第8版』、p330
- ^ 『エッセンシャル遺伝子第1版』、p250
- ^ a b c 『エッセンシャル遺伝子第1版』、p251
- ^ 『遺伝子第8版』、p376
- ^ a b c d 『ストライヤー生化学(第6版)』、p788
- ^ 『ゲノム 第3版-新しい生命情報システムへのアプローチ』、p483
- ^ 『ゲノム 第3版-新しい生命情報システムへのアプローチ』、p484
- ^ a b 『ストライヤー生化学(第6版)』、p726
- ^ a b 『ストライヤー生化学(第6版)』、p786
- ^ a b 『ウィーバー分子生物学第4版』、p226
- ^ a b 『ワトソン遺伝子の分子生物学第6版』、p228
- ^ a b c d 『ゲノム 第3版-新しい生命情報システムへのアプローチ』、p493
- ^ a b 『ストライヤー生化学(第6版)』、p787
- ^ a b 『ウィーバー分子生物学第4版』、p766
- ^ a b 『細胞の分子生物学第5版』、p276
- ^ a b c d 『エッセンシャル遺伝子第1版』、p263
- ^ a b c d 『遺伝子第8版』、p339
- ^ a b c d e f g 『エッセンシャル遺伝子第1版』、p252
- ^ a b c d e 『遺伝子第8版』、p331
- ^ a b 『遺伝子第8版』、p332
- ^ “Temporal and spatial regulation of eukaryotic DNA replication: From regulated initiation to genome-scale timing program”. 2014年7月3日閲覧。. Renard-Guillet C, Kanoh Y, Shirahige K, Masai H. Semin Cell Dev Biol. 2014 Jun, pp110-120
- ^ a b c “The histone deacetylases sir2 and rpd3 act on ribosomal DNA to control the replication program in budding yeast”. 2014年7月3日閲覧。. Kazumasa Yoshida, Julien Bacal, Damien Desmarais, Ismaël Padioleau, Olga Tsaponina, Andrei Chabes, Véronique Pantesco, Emeric Dubois, Hugues Parrinello, Magdalena Skrzypczak, Krzysztof Ginalski, Armelle Lengronne, Philippe Pasero. Molecular Cell, 54, pp.691-697 (2014)
- ^ “DNA replication timing”. 2014年7月3日閲覧。.Rhind, N. & Gilbert, D. M.Cold Spring Harb. Perspect., 5, a010132 (2013)
- ^ “The causes of replication stress and their consequences on genome stability and cell fate”. 2014年7月3日閲覧。. Magdalou I, Lopez BS, Pasero P, Lambert SA. Semin Cell Dev Biol. 2014 Jun, pp154-164
- ^ “Stalled fork rescue via dormant replication origins in unchallenged S phase promotes proper chromosome segregation and tumor suppression”. 2014年7月2日閲覧。. Tsuyoshi Kawabata,Spencer W. Luebben, Satoru Yamaguchi,Ivar Ilves, Ilze Matise, Tavanna Buske, Michael R. Botchan, and Naoko Shima, Mol Cell. Mar 4, 2011; 41(5), pp.543–553.
- ^ a b “Replication Stress and Mechanisms of CNV Formation”. 2014年7月3日閲覧。. M. F. Arlt, T. E. Wilson, T. W. Glover. Curr. Opin. Genet. Dev., 22, pp.204-210 (2012)
- ^ “Cell-type-specific replication initiation programs set fragility of the FRA3B fragile site”. 2014年7月3日閲覧。. A. Letessier, G. A. Millot, S. Koundrioukoff, A. M. Lachagès, N. Vogt, R. S. Hansen, B. Malfoy, O. Brison, M. Debatisse. Nature, 470, pp120-123 (2011)
- ^ “Mechanisms for recurrent and complex human genomic rearrangements”. 2014年7月3日閲覧。. P. Liu, C. M. Carvalho, P. J. Hastings, J. R. Lupski. Curr Opin Genet Dev. 22(3), pp211-20 (2012)
- ^ “A fixed site of DNA replication in eucaryotic cells”. 2014年7月2日閲覧。. Drew M. Pardoll/Bert Vogelstein/Donald S. Coffey, Cell 19, 1980. pp527-536.
- ^ Lengronne, A., Pasero, P. Bensimon, A., and Schwob, E. (2001). Nucleic Acids Res. 29, pp1433-1442.
- ^ Jackson, D.A., and Pombo, A. (1998). J. Cell Biol. 140, 1285-1295
- ^ Borlado LR, Méndez J (February 2008). “CDC6: from DNA replication to cell cycle checkpoints and oncogenesis”. Carcinogenesis 29 (2): 237–43. doi:10.1093/carcin/bgm268. PMID 18048387.
- ^ a b c 『イラストレイテッド ハーパー・生化学 原書28版』、著者:Robert K. Murray, David A. Bender, Kathleen M. Botham, Peter J. Kennelly, Victor W. Rodwell. P. Anthony Weil、監訳者:上代淑人・清水孝雄、発行:丸善株式会社、2011、p380
- ^ a b c d 『ワトソン遺伝子の分子生物学第6版』、p241
- ^ a b c d 『ストライヤー生化学(第6版)』、p789
- ^ a b c “The Elg1 Replication Factor C-like Complex Functions in PCNA Unloading during DNA Replication”. 2014年7月2日閲覧。. Molecular Cell, Volume 50, Issue 2, July 13-16 2013, pp.273–280
- ^ “Replication Protein A-Directed Unloading of PCNA by the Ctf18 Cohesion Establishment Complex”. 2014年7月2日閲覧。. Molecular Cell, Biol, 25(13). Jul 2005, pp5445–5455.
- ^ “Quantitative Proteomic Analysis of Chromatin Reveals that Ctf18 Acts in the DNA Replication Checkpoint”. 2014年7月2日閲覧。. Mol Cell Proteomics, 10(7). Jul 2011, M110.005561.
- ^ 『ゲノム 第3版-新しい生命情報システムへのアプローチ』、p494
- ^ 『ゲノム 第3版-新しい生命情報システムへのアプローチ』、p481
- ^ a b c d 『遺伝子第8版』、p333
- ^ “Tetrapyrrole signal as a cell-cycle coordinator from organelle to nuclear DNA replication in plant cells.”. 2014年7月3日閲覧。.Yuki Kobayashi, Yu Kanesaki, Ayumi Tanaka, Haruko Kuroiwa, Tsuneyoshi Kuroiwa, Kan Tanak. PNAS Vol.106 No.3 (2008)
- ^ a b “A tetrapyrrole-regulated ubiquitin ligase controls algal nuclear DNA replication”. 2014年7月3日閲覧。.Yuki Kobayashi, Sousuke Imamura, Mitsumasa Hanaoka, Kan Tanaka. Nature Cell Biology 13, pp483–487 (2011)
- ^ Elizabeth R. Barry; Stephen D. Bell (Dec 2006). “DNA Replication in the Archaea”. Microbiol Mol Biol Rev. 70 (4): 876–887.
- ^ a b c d e f 『遺伝子第8版』、p335
- ^ a b c d 『ウィーバー分子生物学第4版』、化学同人、著者:Robert F. Weaver、監訳者:杉山弘、2008、p713
- ^ a b c 『遺伝子第8版』、p336



