SCF複合体
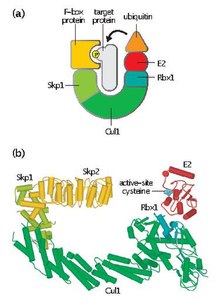
SCF複合体(SCFふくごうたい、英: SCF complex [Skp, Cullin, F-box containing complex])は複数のタンパク質からなるE3ユビキチンリガーゼ複合体で、タンパク質を26Sプロテアソームによる分解の標的とするためのユビキチン化を触媒する[2]。後期促進複合体とともに、SCF複合体は細胞周期に関与するタンパク質のユビキチン化に重要な役割を果たす。また、SCF複合体は他のさまざまなタンパク質も分解の標的とする[3]。
構成要素[編集]
SCF複合体はさまざまなF-boxタンパク質と3つのコアサブユニットが含まれる。
- F-boxタンパク質(FBP)– FBPは、まずSCF複合体非依存的に標的タンパク質に結合し、SCF複合体の基質特異性に寄与する。各FBP(Skp2など)はそれぞれいくつかの異なる基質を認識するが、認識はリン酸化やグリコシル化などの翻訳後修飾に依存している。その後、FBPはF-boxモチーフを利用してSCF複合体のSkp1に結合し、標的タンパク質を機能的なE2ユビキチン結合酵素の近傍へと運ぶ。また、FBPは細胞周期の各段階におけるSCFの活性の調節にも必要不可欠である。SCF複合体のタンパク質レベルは細胞周期を通じて一定であるが、FBPのタンパク質基質に対する親和性はサイクリン-CDKを介した標的タンパク質のリン酸化によって調節されている[4]。
- Skp1– Skp1はF-boxタンパク質の認識と結合に必要不可欠なアダプタータンパク質である。
- Cullin(CUL1)– CullinはSCF複合体の主要な構造的足場を形成し、Skp1とRbx1を連結させる。CullinとFBPのさまざまな組み合わせによって、さまざまな基質を標的とする約100種類のE3ユビキチンリガーゼが形成される[5]。
- Rbx1– Rbx1は亜鉛を結合する小さなRINGフィンガードメインを含んでおり、E2ユビキチン結合酵素を結合する。この結合によって、E2酵素から標的タンパク質のリジン残基へユビキチンの転移が行われる。
発見[編集]
SCF複合体の発見を導く最初の手がかりは、出芽酵母Saccharomyces cerevisiaeの遺伝的スクリーニングから得られた。cdc4、cdc34、cdc53などの細胞周期の進行に異常がみられる温度感受性変異体は、DNA複製が起こっていない状態でG1期に細胞周期が停止し、複数の出芽がみられる[6][7]。この表現型はS期サイクリン-CDK複合体の阻害因子であるSic1を分解できないことが原因であった[6]。これらの発見から、G1/S期の移行にタンパク質分解が重要であることが示された。
続いて、生化学的研究によりCdc34がE2酵素であり、Cdc34はSkp1、Cdc4といくつかのタンパク質が含まれるE3ユビキチンリガーゼ複合体と物理的に相互作用することが明らかにされた[6]。Skp1の既知の結合パートナー、具体的にはSkp2、サイクリンF、Cdc4は約40残基からなる共通したモチーフを持つことが判明し、F-boxモチーフと名付けられた。これらの発見に続いて提唱されたF-box仮説[8]では、F-boxタンパク質が分解標的となる基質をリクルートし、Skp1がF-boxタンパク質とコアユビキチン化複合体を関連付けているとされた。
その後の線虫Caenorhabditis elegansでの遺伝学的研究により、他のSCF複合体の構成要素が解明された[8]。
細胞周期の調節[編集]
真核生物の細胞周期[9]は、調節タンパク質の合成、分解、結合による相互作用、翻訳後修飾によって調節されている。こうした調節タンパク質の中で、2つのユビキチンリガーゼが細胞周期チェックポイントの通過に重要である。後期促進複合体(APC)は中期から後期への移行を制御し、一方SCF複合体はG1/S期とG2/M期の移行を制御する。具体的には、SCFは終期の終わりからG1/S期の移行にかけての中心小体の分裂を制御することが示されている[2]。
SCFの活性は翻訳後修飾によって大きく制御されている。例えば、ユビキチンを介したFBPの自己触媒分解はSCFの活性を低下させる機構の1つである。
SCF複合体の基質となる細胞周期関連タンパク質としてよく特徴づけられているものには次のようなものがある。
- サイクリンファミリータンパク質: サイクリンD、サイクリンE[10]
- 転写調節因子: Myc、E2f1、p130[10]
- サイクリン依存性キナーゼ阻害因子(CKI): p27Kip1、p21、Wee1[10]
- 中心小体タンパク質: Cep250、Ninein[2]
ヒトには約70種類のFBPが存在し、そのいくつかはSCF複合体の構成要素として細胞周期の制御に関与している[11]。
Skp2はp27Kip1やp21などのCKIに結合するFBPである。p27Kip1がサイクリンA/E-CDK2によってリン酸化されており、Cks1に結合しているときにのみ、Skp2はp27Kip1を結合する。Skp2の結合によって、p27Kip1はG1期の終盤とS期の序盤にユビキチン化されて分解の標的となる[4]。SCF-Skp2は、p130もリン酸化依存的に分解標的とする。
βTrCP(β-transducin repeat-containing protein)は、有糸分裂初期にWee1と、APC-Cdh1の阻害因子であるEmi1を分解の標的とするFBPである[10]。βTrCPは、これらがPlk1またはサイクリンB-CDK1によってリン酸化された後に基質として認識を行う。
Fbw7は酵母Cdc4のヒトホモログであり、サイクリンE、Myc、Notch、c-Junを分解の標的とするFBPである[4]。Fbw7は細胞周期を通じて安定であり[12]、核局在化配列が存在するため核に局在している[13]。SCF-Fbw7はSwi5とSic1(p27Kip1、9つのリン酸化部位のうち少なくとも6つがリン酸化されている場合)を分解の標的とする[14]。Sic1は通常サイクリンB-CDK1を阻害することでS期への移行を防いでおり、Sic1が分解の標的となることでS期への移行が促進される。Fbw7はいくつかの散発性の癌腫においてハプロ不全型のがん抑制因子であることが示唆されており、すなわち野生型の表現型の破壊には1つのアレルの変異で十分である[15]。
Fbxo4は、ヒトの癌腫においてがん抑制因子であることが示唆されている他のFBPである。SCF-Fbxo4はサイクリンD1を分解標的とすることで細胞周期の制御に関与している[4]。
サイクリンFは筋萎縮性側索硬化症(ALS)と前頭側頭型認知症(FTD)と関係しているFBPである[16][17]。サイクリンFのリン酸化を阻害する変異はSCF-サイクリンFの活性を変化させ、ALSとFTDでの神経細胞の変性と関係した下流の過程に影響を与えている可能性が高い[17]。通常、サイクリンFはE2f1を分解の標的とする。
がん[編集]
SCF複合体はヒトの一部のがんでアップレギュレーションされており、また生化学的に明確な活性部位を持つため、魅力的な抗がん剤標的となっている[18]。上述したFBPの多くはがんへの関与が示唆されているが、細胞毒性が薬剤開発の障害となっている[19]。
Skp2を標的としたアンチセンスオリゴヌクレオチドとsiRNAが創薬パイプラインにあり、予備的研究ではSkp2のダウンレギュレーションによってメラノーマ、肺がん細胞、口腔がん細胞、膠芽腫細胞の増殖を阻害できることが示されている[19]。
βTrCPを標的としたsiRNAは、乳がん細胞と子宮頸がん細胞に対し既存の化学療法への感受性を高めることが示されている[19]。
植物ホルモンシグナル伝達[編集]
植物ホルモンのオーキシンはTir1(Transport Inhibitor Response 1)タンパク質に結合する。Tir1はAFB(Auxin Signaling F-box Protein)であり、オーキシン受容体として機能する。オーキシンが結合したTir1はSCF-Tir1のAUX/IAAリプレッサーへの結合を促進する。その後のリプレッサーの分解によって、AUX/IAA遺伝子(オーキシン応答性遺伝子)が活性化される[20]。
植物ホルモンのジャスモン酸は、FBPであるCoi1タンパク質に結合する。SCF-Coi1はJAZ転写因子に結合し、分解標的とする。JAZ転写因子の分解によってジャスモン酸応答性遺伝子の転写が行われる[21]。
出典[編集]
- ^ Zheng, Ning; Schulman, Brenda A.; Song, Langzhou; Miller, Julie J.; Jeffrey, Philip D.; Wang, Ping; Chu, Claire; Koepp, Deanna M. et al. (2002-04-18). “Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex”. Nature 416 (6882): 703–709. doi:10.1038/416703a. ISSN 0028-0836. PMID 11961546.
- ^ a b c Ou, Young; Rattner, J.B. (2004), “The Centrosome in Higher Organisms: Structure, Composition, and Duplication”, International Review of Cytology (Elsevier) 238: 119–182, doi:10.1016/s0074-7696(04)38003-4, ISBN 978-0-12-364642-2, PMID 15364198
- ^ Morgan, David "Protein Degradation in Cell-Cycle Control", The Cell Cycle; Principles of Control 2007
- ^ a b c d The Molecular Basis of Cancer. Elsevier. (2008). doi:10.1016/b978-1-4160-3703-3.x5001-7. ISBN 978-1-4160-3703-3
- ^ Hegde, Ashok N. (2010), “Ubiquitin-Dependent Protein Degradation”, Comprehensive Natural Products II, Elsevier, pp. 699–752, doi:10.1016/b978-008045382-8.00697-3, ISBN 978-0-08-045382-8 2019年12月1日閲覧。
- ^ a b c Patton, E (1998-06-01). “Combinatorial control in ubiquitin-dependent proteolysis: don't Skp the F-box hypothesis”. Trends in Genetics 14 (6): 236–243. doi:10.1016/s0168-9525(98)01473-5. ISSN 0168-9525. PMID 9635407.
- ^ Schwob, E (1994-10-21). “The B-type cyclin kinase inhibitor p40SIC1 controls the G1 to S transition in S. cerevisiae”. Cell 79 (2): 233–244. doi:10.1016/0092-8674(94)90193-7. ISSN 0092-8674. PMID 7954792.
- ^ a b Willems, Andrew R.; Schwab, Michael; Tyers, Mike (November 2004). “A hitchhiker's guide to the cullin ubiquitin ligases: SCF and its kin”. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research 1695 (1–3): 133–170. doi:10.1016/j.bbamcr.2004.09.027. ISSN 0167-4889. PMID 15571813.
- ^ Vodermaier, Hartmut C. (September 2004). “APC/C and SCF: Controlling Each Other and the Cell Cycle”. Current Biology 14 (18): R787–R796. doi:10.1016/j.cub.2004.09.020. ISSN 0960-9822. PMID 15380093.
- ^ a b c d Fischer, Martin; Dang, Chi V.; DeCaprio, James A. (2018), “Control of Cell Division”, Hematology (Elsevier): pp. 176–185, doi:10.1016/b978-0-323-35762-3.00017-2, ISBN 978-0-323-35762-3
- ^ Lambrus, Bramwell G.; Moyer, Tyler C.; Holland, Andrew J. (2017-08-31). Applying the auxin-inducible degradation (AID) system for rapid protein depletion in mammalian cells. doi:10.1101/182840.
- ^ Mathias, Neal; Steussy, C. Nic; Goebl, Mark G. (1998-02-13). “An Essential Domain within Cdc34p Is Required for Binding to a Complex Containing Cdc4p and Cdc53p inSaccharomyces cerevisiae”. Journal of Biological Chemistry 273 (7): 4040–4045. doi:10.1074/jbc.273.7.4040. ISSN 0021-9258. PMID 9461595.
- ^ Blondel, M. (2000-11-15). “Nuclear-specific degradation of Far1 is controlled by the localization of the F-box protein Cdc4”. The EMBO Journal 19 (22): 6085–6097. doi:10.1093/emboj/19.22.6085. ISSN 1460-2075. PMC 305831. PMID 11080155.
- ^ Kishi, T.; Ikeda, A.; Koyama, N.; Fukada, J.; Nagao, R. (2008-09-11). “A refined two-hybrid system reveals that SCFCdc4-dependent degradation of Swi5 contributes to the regulatory mechanism of S-phase entry”. Proceedings of the National Academy of Sciences 105 (38): 14497–14502. Bibcode: 2008PNAS..10514497K. doi:10.1073/pnas.0806253105. ISSN 0027-8424. PMC 2567208. PMID 18787112.
- ^ Calhoun, Eric S.; Jones, Jessa B.; Ashfaq, Raheela; Adsay, Volkan; Baker, Suzanne J.; Valentine, Virginia; Hempen, Paula M.; Hilgers, Werner et al. (October 2003). “BRAF and FBXW7 (CDC4, FBW7, AGO, SEL10) Mutations in Distinct Subsets of Pancreatic Cancer”. The American Journal of Pathology 163 (4): 1255–1260. doi:10.1016/s0002-9440(10)63485-2. ISSN 0002-9440. PMC 1868306. PMID 14507635.
- ^ Yu, Yujiao; Nakagawa, Tadashi; Morohoshi, Akane; Nakagawa, Makiko; Ishida, Noriko; Suzuki, Naoki; Aoki, Masashi; Nakayama, Keiko (2019-09-30). “Pathogenic mutations in the ALS gene CCNF cause cytoplasmic mislocalization of Cyclin F and elevated VCP ATPase activity”. Human Molecular Genetics 28 (20): 3486–3497. doi:10.1093/hmg/ddz119. ISSN 0964-6906. PMID 31577344.
- ^ a b Lee, Albert; Rayner, Stephanie L.; De Luca, Alana; Gwee, Serene S. L.; Morsch, Marco; Sundaramoorthy, Vinod; Shahheydari, Hamideh; Ragagnin, Audrey et al. (October 2017). “Casein kinase II phosphorylation of cyclin F at serine 621 regulates the Lys48-ubiquitylation E3 ligase activity of the SCF (cyclin F) complex”. Open Biology 7 (10): 170058. doi:10.1098/rsob.170058. ISSN 2046-2441. PMC 5666078. PMID 29021214.
- ^ Skaar, Jeffrey R.; Pagan, Julia K.; Pagano, Michele (December 2014). “SCF ubiquitin ligase-targeted therapies”. Nature Reviews Drug Discovery 13 (12): 889–903. doi:10.1038/nrd4432. ISSN 1474-1776. PMC 4410837. PMID 25394868.
- ^ a b c Sun, L. Jia and Y. (2011-02-28). “SCF E3 Ubiquitin Ligases as Anticancer Targets”. Current Cancer Drug Targets 11 (3): 347–356. doi:10.2174/156800911794519734. PMC 3323109. PMID 21247385.
- ^ Dharmasiri, Nihal; Dharmasiri, Sunethra; Estelle, Mark (May 2005). “The F-box protein TIR1 is an auxin receptor”. Nature 435 (7041): 441–445. Bibcode: 2005Natur.435..441D. doi:10.1038/nature03543. ISSN 0028-0836. PMID 15917797.
- ^ Devoto, Alessandra; Nieto-Rostro, Manuela; Xie, Daoxin; Ellis, Christine; Harmston, Rebecca; Patrick, Elaine; Davis, Jackie; Sherratt, Leigh et al. (November 2002). “COI1 links jasmonate signalling and fertility to the SCF ubiquitin-ligase complex inArabidopsis”. The Plant Journal 32 (4): 457–466. doi:10.1046/j.1365-313x.2002.01432.x. ISSN 0960-7412. PMID 12445118.
