T4ファージ
| T4ファージ | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 Bacteriophage T4 Structural Model[1]
| ||||||||||||||||||||||||
| 分類 | ||||||||||||||||||||||||
| ||||||||||||||||||||||||
| シノニム | ||||||||||||||||||||||||
|
Enterobacteria phage T4 [2] |
腸内細菌ウイルスT4 (T4ファージ) は大腸菌に感染するバクテリオファージの種である。二本鎖DNAウイルスに分類され、ミオウイルス科に属する。T4ファージの感染は必ず溶菌を起こし、溶原化しない。過去には他のファージ種や系統であるT2ファージやT6ファージと合わせて、T偶数ファージとも呼ばれていた。
バクテリオファージの語源は「細菌を食べるもの」を意味し、ファージは偏性細胞内寄生体で宿主の細胞内で増殖し、溶菌とともに細胞外へ放たれる。160種ほどの遺伝子を持つT4ファージは、ウイルスとしては最大級の大きさを持ち、モデル生物として重用されてきた。現在に至るまでT4ファージはウイルス学と分子生物学の発展に重要な役割を果たしてきている[3][4]。
T4ファージの研究における利用[編集]
1940年代から現在に至るまでT偶数ファージは最も研究されてきたモデル生物である。モデル生物には一般にできるだけ単純なゲノム構造が必要とされる。一方でT偶数ファージはウイルスとしては最大級でかつ複雑なウイルスであり、約160種の遺伝子を持つ。また、他のウイルスではありえない、ヒドロキシメチルシトシン(HMC)がシトシンの代わりに存在するという特徴を持つ。しかもこのHMCは特定のパターンで糖鎖修飾を受ける。また、T偶数ファージは遺伝子発現制御にも独自の特徴を持つ[3]。このような特徴によりT偶数ファージは、薬剤耐性の伝播を担う形質導入、新しい酵素などの形質の獲得に関わる溶原変換、変異をもたらす細菌ゲノムへのランダムな挿入、細菌の疫学的型別への応用、さらには遺伝子工学におけるクローニングベクターとしての利用といった重要な意義を持つ。例えば、遺伝子ライブラリやモノクローナル抗体のライブラリの構築にはファージが用いられる。さらにはファージは自然環境下で水から細菌を除去する作用も持つ[5]。
ゲノムとその構造[編集]
T4ウイルスは169kbpの二本鎖DNAをゲノムとし、289のタンパク質をコードする[6]。T4ゲノムは末端が重複しており、まずユニットとして複製された後に、各ゲノムユニットが末端間で結合してコンカテマーと呼ばれる直列多量体を形成する。パッケージングにおいてコンカテマーは元のゲノムの長さと同じ長さになるように非特異的な部位で切断され、元のゲノムと巡回置換なゲノムを生じる[7]。また、T4ゲノムは真核生物の様なイントロン配列を含む
翻訳[編集]
シャイン・ダルガノ配列、GAGGはT4ファージの初期遺伝子で支配的だが、GAGG配列は感染初期のmRNA分解を開始するT4エンドヌクレアーゼRegBの標的でもある[8]。
ウイルス粒子の構造[編集]

T4ファージはウイルスとしては大きく、ほとんどのウイルスの長さが25nmから200nmの長さであるのに対し、幅がおよそ90nm、長さが200nmである。DNAのゲノムはカプシドと呼ばれる二十面体の頭部に格納される[9]。尾部は空洞であり、細菌の細胞に吸着したファージが、細胞内に核酸を送る際に核酸が尾部を通過する[10]。尾部は宿主の細胞表面に存在する受容体の認識において重要であり、吸着した細菌が宿主域の範囲であるか否かを決定する[11]。
T4ファージの尾部の末端である6MDaの基盤は、13種類のタンパク質(遺伝子産物5、5.4、6、7、8、9、10、11、12、25、27、48、53)の組み合わせからなる、全127本のポリペプチド鎖で構成される。近年原子レベルで詳細な構造が明らかにされた。尾管の近位領域はgp54が構成しているが、主な部分はgp19が構成している。ものさしタンパク質gp29は基盤-尾管複合体に存在するが、モデル化されていない[12]。
感染の過程[編集]
T4ファージはlong tail fiber (LTF) が大腸菌の細胞表面に存在するOmpCポリンタンパク質とリポ多糖 (LPS) に結合することで感染する[13][14]。認識シグナルはLTFを通じて基盤に送られ、これによりshort tail fiber (STF) が不可逆的に大腸菌の細胞表面に結合する。基盤の構造変化と筒状の構造物であるtail sheathの収縮により、尾管の末端にあるGP5が細菌の細胞外膜に穴を開ける。GP5のリゾチームドメインが活性化し、細胞表層のペプチドグリカン層を分解する。残りの膜成分も分解を受けると、頭部に格納されたDNAが尾管を通って大腸菌の細胞内へ侵入する。
増殖[編集]
ファージが細菌内に侵入して細胞を破壊するまでの溶菌サイクルは37℃でおおよそ30分程度で終了する。ビルレントファージは細菌宿主に感染すると直ちに自己増殖を開始する。子ウイルスの数が一定の量に達すると、ファージは宿主を溶解して破壊し、菌体外へ放出されて次の宿主細胞へと感染する[15]。この宿主の溶解とファージの放出を溶菌サイクルと呼ぶ。つまり、溶菌サイクルは感染した細胞とその細胞膜の破壊を伴うウイルスの増殖過程であると言える。そのため、ウイルスは増殖と宿主細胞への感染のために以下の5つの過程を踏まえる必要がある。
- 吸着と侵入
- 宿主の遺伝子発現の拘束
- 酵素の合成
- DNAの複製
- 新しいウイルス粒子の構成
新しいウイルスの合成が完了すると宿主の細胞は破けて新生ウイルスを環境中に放ち、宿主の細胞は崩壊に至る。菌体が崩壊した際に放出される子孫ウイルスの数をburst sizeと呼び、T4ファージの場合は感染した1個の菌体当たりで100-150個である。
吸着と侵入[編集]
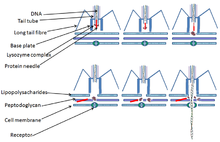
他のファージと同様に、T偶数ファージはランダムに宿主の菌体表面に吸着するわけではない。ファージは菌体の表面にある、特定の構造を持つタンパク質である、受容体に選択的に結合する。受容体の種類はファージの種ごとに異なり、タイコ酸、細胞壁を構成するタンパク質やリポ多糖、鞭毛、線毛など多岐にわたる構造が受容体としてファージとの結合に寄与しうる。ファージが細菌に感染してその生活環を完成させるためには、感染の最初の過程である菌体表面への吸着を果たす必要がある。吸着はファージ毎に特異的な宿主細菌に対してのみ生じるものであり、2つの段階を経て行われる。第1の過程は可逆的な結合であり、ファージのLTFが宿主細菌の持つ受容体に結合する。第2の過程である不可逆的過程においては、ファージの基盤がファージと細菌の結合を担う。
不可逆的な吸着を果たしたT4ファージは尾部の外側を覆う鞘を収縮させ、その内部に存在する管状構造を細菌の細胞壁と細胞膜に向けて注入する。細胞壁にはペプチドグリカンの層が存在するが、先端のgp5がこれを分解する。近年の研究により、注入された内筒は菌体の内膜を貫通せず、内膜と融合することが明らかにされた。このようにしてできた通路をファージのゲノムDNAが通過し、菌体内へと侵入する[16][17][18]。
複製とパッケージング[編集]
T4ファージのゲノムは宿主のRolling Circle Replicationによって合成される。ファージが生菌の菌体内でDNA複製にかける時間は、ファージ感染大腸菌におけるDNA伸長速度として測定される[19]。37°CでDNAの対数増加期における伸長速度は毎秒749bpである。DNA複製時における1塩基あたりの変異率は、1.7 × 10−8であり[20]、T4ファージのDNA複製は非常に正確である。これは300コピーのT4ファージゲノムが、一つしかエラーを生じないことを意味する。また、T4ファージは独自のDNA修復機構を持っている。ファージの頭部は足場タンパク質の周囲に空の状態で組み立てられ、足場タンパク質はその後分解される。DNAは小さい孔を通過して前駆体頭部 (prohead) に格納されるが、DNAの格納に関わるのがDNAと最初相互作用するgp17であり、この分子はDNAの折りたたみモーター及びヌクレアーゼとしても機能する。T4ファージの頭部にDNAを折りたたむ速度は1秒あたり2000塩基長であり、大きさを同等にした場合、その馬力は平均的な乗用車用エンジンに相当する[21]。また溶菌サイクルにおいて、ファージのカプシドへ細菌DNAの一部を取り込む形質導入が生じる[22]。
放出[編集]
増殖過程の最終段階で、T4ファージは宿主となる菌体からウイルス粒子を放出する。ウイルス粒子の放出は細菌の細胞膜が破壊された後に起きる。ウイルスタンパク質がペプチドグリカンや細胞膜を破壊する溶菌を起こす。放出されたバクテリオファージは他の菌体に感染し、増殖サイクルを繰り返す。
出典[編集]
- ^ Padilla-Sanchez, Victor (2021). “Structural Model of Bacteriophage T4”. WikiJournal of Science 4 (1): 5. doi:10.15347/WJS/2021.005.
- ^ “ICTV Taxonomy history: Escherichia virus T4” (英語). International Committee on Taxonomy of Viruses (ICTV). 2018年12月26日閲覧。 “Caudovirales > Myoviridae > Tevenvirinae > T4virus > Escherichia virus T4”
- ^ a b Norkin, Leonard C. (2010). Virology, Molecular Biology and Pathogenesis. Washington: American Society for Microbiology. pp. 725. ISBN 978-1-55581-453-3
- ^ Prescott, Harley, and Klein (2008). Microbiology (seventh ed.). McGraw Hill. pp. 1078. ISBN 978-007-126727-4
- ^ “Bacteriophages”. microrao. 1900年1月1日閲覧。
- ^ Miller, ES; Kutter, E; Mosig, G; Arisaka, F; Kunisawa, T; Rüger, W (March 2003). “Bacteriophage T4 genome.”. Microbiology and Molecular Biology Reviews 67 (1): 86–156, table of contents. doi:10.1128/MMBR.67.1.86-156.2003. PMC 150520. PMID 12626685.
- ^ Brock Biology of Microorganisms (11th ed.). Prentice Hall. (2006). ISBN 978-0-13-144329-7
- ^ Malys N (2012). “Shine-Dalgarno sequence of bacteriophage T4: GAGG prevails in early genes”. Molecular Biology Reports 39 (1): 33–9. doi:10.1007/s11033-011-0707-4. PMID 21533668.
- ^ Prescott, Harley, and Klein (2008). Microbiology (seventh ed.). McGraw-Hill. ISBN 978-007-126727-4
- ^ Petr G Leiman, Fumio Arisaka, Mark J van Raaij, Victor A Kostyuchenko, Anastasia A Aksyuk, Shuji Kanamaru & Michael G Rossmann. Morphogenesis of the T4 tail and tail fibers, Virology Journal, volume 7, Article number: 355 (2010) https://virologyj.biomedcentral.com/articles/10.1186/1743-422X-7-355
- ^ Ackermann, H.-W.; Krisch, H. M. (6 April 2014). “A catalogue of T4-type bacteriophages”. Archives of Virology 142 (12): 2329–2345. doi:10.1007/s007050050246. PMID 9672598.
- ^ Taylor, Nicholas M. I.; Prokhorov, Nikolai S.; Guerrero-Ferreira, Ricardo C.; Shneider, Mikhail M.; Browning, Christopher; Goldie, Kenneth N.; Stahlberg, Henning; Leiman, Petr G. (2016). “Structure of the T4 baseplate and its function in triggering sheath contraction”. Nature 533 (7603): 346–352. Bibcode: 2016Natur.533..346T. doi:10.1038/nature17971. PMID 27193680.
- ^ Yu, F.; Mizushima, S. (1982). “Roles of lipopolysaccharide and outer membrane protein OmpC of Escherichia coli K-12 in the receptor function for bacteriophage T4”. Journal of Bacteriology 151 (2): 718–722. doi:10.1128/JB.151.2.718-722.1982. PMC 220313. PMID 7047495.
- ^ Furukawa, H.; Mizushima, S. (1982). “Roles of cell surface components of Escherichia coli K-12 in bacteriophage T4 infection: Interaction of tail core with phospholipids”. Journal of Bacteriology 150 (2): 916–924. doi:10.1128/JB.150.2.916-924.1982. PMC 216445. PMID 7040345.
- ^ Sherwood, Linda (2011). Prescott's Microbiology (eighth ed.). McGraw-Hill
- ^ Xu, Jingwei; Xiang, Ye (2017-07-01). Tsai, Billy. ed. “Membrane Penetration by Bacterial Viruses” (英語). Journal of Virology 91 (13): e00162–17, e00162–17. doi:10.1128/JVI.00162-17. ISSN 0022-538X. PMC 5469264. PMID 28404851.
- ^ Norkin, Leonard C. (2010). Virology, Molecular Biology and Pathogenesis. Washington: American Society for Microbiology. p. 31. ISBN 978-1-55581-453-3
- ^ Prescott, Harley, and Klein (2008). Microbiology (seventh ed.). McGraw Hill. p. 427. ISBN 978-007-126727-4
- ^ “DNA elongation rates and growing point distributions of wild-type phage T4 and a DNA-delay amber mutant”. J Mol Biol 106 (4): 963–81. (1976). doi:10.1016/0022-2836(76)90346-6. PMID 789903.
- ^ Drake JW (1970) The Molecular Basis of Mutation. Holden-Day, San Francisco ISBN 0816224501, 978-0816224500
- ^ Rao, Venigalla B; Black, Lindsay W (1 January 2010). “Structure and assembly of bacteriophage T4 head”. Virology Journal 7 (1): 356. doi:10.1186/1743-422X-7-356. PMC 3012670. PMID 21129201.
- ^ Leonard C., Norkin (2010). Virology, Molecular Biology and Pathogenesis. Washington: American Society for Microbiology
関連文献[編集]
- Leiman P.G.; Kanamaru S; Mesyanzhinov V.V.; Arisaka F.; Rossmann M.G. (2003). “Structure and morphogenesis of bacteriophage T4.”. Cellular and Molecular Life Sciences 60 (11): 2356–2370. doi:10.1007/s00018-003-3072-1. PMID 14625682.
- Karam, J., Petrov, V., Nolan, J., Chin, D., Shatley, C., Krisch, H., and Letarov, A. The T4-like phages genome project. https://web.archive.org/web/20070523215704/http://phage.bioc.tulane.edu/. (The T4-like phage full genomic sequence depository)
- Mosig, G., and F. Eiserling. 2006. T4 and related phages: structure and development, R. Calendar and S. T. Abedon (eds.), The Bacteriophages. Oxford University Press, Oxford. (Review of phage T4 biology) ISBN 0-19-514850-9
- Filee J. Tetart F.; Suttle C.A.; Krisch H.M. (2005). “Marine T4-type bacteriophages, a ubiquitous component of the dark matter of the biosphere”. Proc. Natl. Acad. Sci. USA 102 (35): 12471–6. Bibcode: 2005PNAS..10212471F. doi:10.1073/pnas.0503404102. PMC 1194919. PMID 16116082. (Indication of prevalence and T4-like phages in the wild)
- Chibani-Chennoufi S.; Canchaya C.; Bruttin A.; Brussow H. (2004). “Comparative genomics of the T4-Like Escherichia coli phage JS98: implications for the evolution of T4 phages”. J. Bacteriol. 186 (24): 8276–86. doi:10.1128/JB.186.24.8276-8286.2004. PMC 532421. PMID 15576776. (Characterization of a T4-like phage)
- “The diversity and evolution of the T4-type bacteriophages”. Res. Microbiol. 154 (4): 259–67. (May 2003). doi:10.1016/S0923-2508(03)00069-X. PMID 12798230.
- Miller, E.S.; Kutter E.; Mosig G.; Arisaka F.; Kunisawa T.; Ruger W. (2003). “Bacteriophage T4 genome”. Microbiol. Mol. Biol. Rev. 67 (1): 86–156. doi:10.1128/MMBR.67.1.86-156.2003. PMC 150520. PMID 12626685. (Review of phage T4, from the perspective of its genome)
- Desplats C.; Dez C.; Tetart F.; Eleaume H.; Krisch H.M. (2002). “Snapshot of the genome of the pseudo-T-even bacteriophage RB49”. J. Bacteriol. 184 (10): 2789–2804. doi:10.1128/JB.184.10.2789-2804.2002. PMC 135041. PMID 11976309. (Overview of the RB49 genome, a T4-like phage)
- “A bipartite bacteriophage T4 SOC and HOC randomized peptide display library: detection and analysis of phage T4 terminase (gp17) and late sigma factor (gp55) interaction”. J Mol Biol 319 (2): 289–304. (2002). doi:10.1016/S0022-2836(02)00298-X. PMID 12051907. (T4 phage application in biotechnology for studying protein interaction)
- Tétart F.; Desplats C.; Kutateladze M.; Monod C.; Ackermann H.-W.; Krisch H.M. (2001). “Phylogeny of the major head and tail genes of the wide-ranging T4-type bacteriophages”. J. Bacteriol. 183 (1): 358–366. doi:10.1128/JB.183.1.358-366.2001. PMC 94885. PMID 11114936. (Indication of the prevalence of T4-type sequences in the wild)
- Abedon S.T. (2000). “The murky origin of Snow White and her T-even dwarfs”. Genetics 155 (2): 481–6. PMC 1461100. PMID 10835374. (Historical description of the isolation of the T4-like phages T2, T4, and T6)
- “A catalogue of T4-type bacteriophages”. Arch. Virol. 142 (12): 2329–45. (1997). doi:10.1007/s007050050246. PMID 9672598. オリジナルの1 November 2001時点におけるアーカイブ。. (Nearly complete list of then-known T4-like phages)
- “The genome of the pseudo T-even bacteriophages, a diverse group that resembles T4”. J. Mol. Biol. 267 (2): 237–49. (March 1997). doi:10.1006/jmbi.1996.0867. PMID 9096222. (Overview of various T4-like phages from the perspective of their genomes)
- Kutter E.; Gachechiladze K.; Poglazov A.; Marusich E.; Shneider M.; Aronsson P.; Napuli A.; Porter D. et al. (1995). “Evolution of T4-related phages”. Virus Genes 11 (2–3): 285–297. doi:10.1007/BF01728666. PMID 8828153. (Comparison of the genomes of various T4-like phages)
- Karam, J. D. et al. 1994. Molecular Biology of Bacteriophage T4. ASM Press, Washington, DC. (The second T4 bible, go here, as well as Mosig and Eiserling, 2006, to begin to learn about the biology T4 phage) ISBN 1-55581-064-0
- Eddy, S. R. 1992. Introns in the T-Even Bacteriophages. PhD thesis. University of Colorado at Boulder. (Chapter 3 provides overview of various T4-like phages as well as the isolation of then-new T4-like phages)
- Surdis, T.J "et al" Bacteriophage attachment methods specific to T4, analysis, Overview.
- Mathews, C. K., E. M. Kutter, G. Mosig, and P. B. Berget. 1983. Bacteriophage T4. American Society for Microbiology, Washington, DC. (The first T4 bible; not all information here is duplicated in Karam et al., 1994; see especially the introductory chapter by Doermann for a historical overview of the T4-like phages) ISBN 0-914826-56-5
- Russell, R. L. 1967. Speciation Among the T-Even Bacteriophages. PhD thesis. California Institute of Technology. (Isolation of the RB series of T4-like phages)
- “Non-canonical RNA arrangement in T4-even phages: accommodated ribosome binding site at the gene 26-25 intercistronic junction”. Mol Microbiol 73 (6): 1115–1127. (2009). doi:10.1111/j.1365-2958.2009.06840.x. PMID 19708923. (rare type of translational regulation characterized in T4)
- Kay D.; Fildes P. (1962). “Hydroxymethylcytosine-containing and tryptophan-dependent bacteriophages isolated from city effluents”. J. Gen. Microbiol. 27: 143–6. doi:10.1099/00221287-27-1-143. PMID 14454648. (T4-like phage isolation, including that of phage Ox2)
