「フォールディング」の版間の差分
タグ: サイズの大幅な増減 ビジュアルエディター |
|||
| 1行目: | 1行目: | ||
[[Image:Protein folding.png |
[[Image:Protein folding.png|thumb|right|360px|フォールディング前とフォールディング後のタンパク質]] |
||
[[File:Protein structure.png|right|360px|thumb|タンパク質フォールディングの結果]] |
|||
'''フォールディング''' (folding) は、[[タンパク質]]が特定の立体構造に折りたたまれる現象をいう。 |
|||
タンパク質'''フォールディング''' ({{Lang-en|Protein folding}}) とは、タンパク質鎖がその本来の三次元構造、通常は生物学的に機能する[[コンホメーション]](立体構造)を、迅速かつ再現性のある方法で獲得する[[物理変化|物理的なプロセス]]である。これは、[[ポリペプチド]]が[[ランダムコイル]]からその特徴的で機能的な[[タンパク質構造|三次元構造]]に折りたたまれる物理的な過程である<ref name="Alberts">{{cite book | last1 = Alberts | first1 = Bruce | first2 = Alexander | last2 = Johnson | first3 = Julian | last3 = Lewis | first4 = Martin | last4 = Raff | first5 = Keith | last5 = Roberts | first6 = Peter | last6 = Walters | name-list-format = vanc | title = Molecular Biology of the Cell; Fourth Edition | publisher = Garland Science| year = 2002 | location = New York and London | chapter = The Shape and Structure of Proteins | chapterurl = https://www.ncbi.nlm.nih.gov/books/NBK26830/ | isbn = 978-0-8153-3218-3 | author-link1 = Bruce Alberts}}</ref>。それぞれのタンパク質は、[[mRNA]]の配列から[[アミノ酸]]の直鎖に[[翻訳 (生物学)|翻訳]]されるとき、折りたたまれていないポリペプチドまたはランダムコイルとして存在する。そのポリペプチドは、安定した (長続きする) 立体構造を欠いている (第1図の左側)。そのポリペプチド鎖が[[リボソーム]]で合成されていく過程で、直鎖が三次元構造に折りたたまれる。フォールディングは、ポリペプチド鎖の翻訳中でも始まる。アミノ酸は互いに相互作用して、明確に定義された三次元構造、つまり{{仮リンク|天然状態|en|Native state|label=}}として知られている折りたたまれたタンパク質 (図の右側) を生成する。結果として生じる三次元構造は、アミノ酸配列または[[一次構造]] ({{仮リンク|アンフィンセンのドグマ|en|Anfinsen's dogma|label=}}) によって決定される<ref name="Anfinsen">{{cite journal | vauthors = Anfinsen CB | title = The formation and stabilization of protein structure | journal = The Biochemical Journal | volume = 128 | issue = 4 | pages = 737–49 | date = July 1972 | pmid = 4565129 | pmc = 1173893 | doi = 10.1042/bj1280737 }}</ref>。 |
|||
タンパク質が機能を発揮するために正しい三次元構造が不可欠であるが、機能性タンパク質の一部は[[天然変性タンパク質|折りたたまれていない状態]]のままになっていることがあり<ref>{{cite book | first1 = Jeremy M. | last1 = Berg | first2 = John L. | last2 = Tymoczko | first3 = Lubert | last3 = Stryer | author-link3 = Lubert Stryer | name-list-format = vanc | title = Biochemistry | publisher = W. H. Freeman | location = San Francisco | year = 2002 | isbn = 978-0-7167-4684-3 | chapter = 3. Protein Structure and Function | chapterurl = https://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Search&db=books&doptcmdl=GenBookHL&term=stryer%5Bbook%5D+AND+215168%5Buid%5D&rid=stryer.chapter.280}}</ref>、ゆえに{{仮リンク|タンパク質動力学|en|Protein dynamics|label=}}が重要となる。本来の構造に折りたたまれないと、一般に不活性なタンパク質が生成されるが、場合によっては、誤って折りたたまれたタンパク質の機能が改変されたり、毒性のある機能性を持つこともある。いくつかの[[神経変性疾患]]やその他の[[疾患]]は、誤って折りたたまれたタンパク質によって形成された[[アミロイド]]{{仮リンク|原線維|en|Fibril|label=}}の蓄積に起因すると考えられている<ref name="Selkoe:03">{{cite journal | vauthors = Selkoe DJ | title = Folding proteins in fatal ways | journal = Nature | volume = 426 | issue = 6968 | pages = 900–4 | date = December 2003 | pmid = 14685251 | doi = 10.1038/nature02264 | bibcode = 2003Natur.426..900S | s2cid = 6451881 }}</ref>。多くの[[アレルギー]]は、一部のタンパク質が正しく折りたたまれていないことが原因で、[[免疫系]]が特定のタンパク質構造に対する[[抗体]]を産生しないために引き起こされる<ref>{{cite book | last1 = Alberts | first1 = Bruce | first2 = Dennis | last2 = Bray | first3 = Karen | last3 = Hopkin | first4 = Alexander | last4 = Johnson | first5 = Julian | last5 = Lewis | first6 = Martin | last6 = Raff | first7 = Keith | last7 = Roberts | first8 = Peter | last8 = Walter | name-list-format = vanc | title = Essential cell biology | date = 2010 | publisher = Garland Science | location = New York, NY | isbn = 978-0-8153-4454-4 | pages = 120–70 | edition = Third | chapter = Protein Structure and Function}}</ref>。 |
|||
==概要== |
|||
細胞内で[[翻訳 (生物学)|翻訳]]されたタンパク質は、すぐにそのアミノ酸配列に応じて固有の立体構造に折りたたまれ、機能を有するようになる。この状態を天然状態(native state)という。この天然状態の構造を熱や変性剤で崩して機能を失わせたものを、変性状態(denatured state)という。フォールディングとは、翻訳時から天然状態への変化、または変性状態から天然状態への変化のことをいう。 |
|||
タンパク質の[[変性]]は、折りたたまれた状態から[[ランダムコイル|折りたたまれていない状態]]に移行するプロセスである。これは、[[調理]]、[[火傷]]、[[プロテオパチー]]、その他の状況で起こる。 |
|||
タンパク質はアミノ酸の複数結合した直鎖状の分子で、可能な立体構造は無数に存在するが、細胞内ではそれぞれ特定の立体構造をとる。このため、かつては細胞内にタンパク質を天然構造に折り畳む「鋳型」があると考えられていた。しかし1950年代から60年代にかけて[[クリスチャン・アンフィンセン]]が、リボヌクレアーゼが試験管内において変性状態から天然状態に変化することを示し、「タンパク質は自発的に、熱力学的に最も安定な立体構造をとる」とした。これを[[:en:Anfinsen's dogma|アンフィンセンのドグマ]]と言い、この折りたたみ過程がフォールディングである。 |
|||
フォールディング・プロセスの所要時間は、目的のタンパク質によって劇的に異なる。[[In vitro|細胞外]]で調べたとき、最も遅く折りたたまれるタンパク質は、主に[[プロリン]][[異性化]]のために折りたたまれるのに数分から数時間を要し、プロセスが完了するまでにチェックポイントのようないくつかの中間状態を通過する必要がある<ref>{{cite journal | vauthors = Kim PS, Baldwin RL | title = Intermediates in the folding reactions of small proteins | journal = Annual Review of Biochemistry | volume = 59 | pages = 631–60 | year = 1990 | pmid = 2197986 | doi = 10.1146/annurev.bi.59.070190.003215 }}</ref>。一方、長さが100アミノ酸までの非常に小さなシングル[[タンパク質ドメイン|ドメイン]]タンパク質は、通常、1回のステップで折りたたむことができる<ref>{{cite journal | vauthors = Jackson SE | title = How do small single-domain proteins fold? | journal = Folding & Design | volume = 3 | issue = 4 | pages = R81-91 | year = 1998 | pmid = 9710577 | doi = 10.1016/S1359-0278(98)00033-9 | doi-access = free }}</ref>。時間スケールはミリ秒が一般的で、非常に速い既知のタンパク質のフォールディング反応は数マイクロ秒以内に完了する<ref>{{cite journal | vauthors = Kubelka J, Hofrichter J, Eaton WA | title = The protein folding 'speed limit' | journal = Current Opinion in Structural Biology | volume = 14 | issue = 1 | pages = 76–88 | date = February 2004 | pmid = 15102453 | doi = 10.1016/j.sbi.2004.01.013 | url = https://zenodo.org/record/1259347 }} |
|||
なお、一部には自発的にフォールディングせず[[シャペロン]]の補助を必要とするタンパク質や、天然状態では構造が確定していない[[天然変性タンパク質]]なども存在する。 |
|||
</ref>。 |
|||
==フォールディング |
== タンパク質のフォールディング過程 == |
||
n残基のタンパク質はおよそ10<sup>n</sup>通りの構造が可能であり、タンパク質が最安定構造を網羅的に探すことは現実的に不可能である(レヴィンタールのパラドクス)。このため、フォールディングには特定の経路が存在する。 |
|||
=== 一次構造 === |
|||
かつては、タンパク質のフォールディングはまず核(足場)となる構造が形成され、その後その周辺の構造が段階的に完成していくというモデルが考えられていた。しかし近年の実験技術の向上により、現在では100残基以下の小型タンパク質に関しては、変性状態と天然状態の二状態いずれかしか観測されず、中間的な構造はほとんど存在しないことが分かっている。このため、小型タンパク質のフォールディングは協同的(cooperative)であるとされる。これは、進化の過程でタンパク質が高速にフォールディングできるアミノ酸配列を獲得したことによると考えられる。実際、ランダムにアミノ酸を並べたポリペプチドでは多くの[[準安定]]な中間状態が存在し、フォールディングは遅い。 |
|||
タンパク質の[[一次構造]]である直線的なアミノ酸配列は、その本来のコンホメーションを決定する<ref name="Anfinsen1">{{cite journal | vauthors = Anfinsen CB | title = Principles that govern the folding of protein chains | journal = Science | volume = 181 | issue = 4096 | pages = 223–30 | date = July 1973 | pmid = 4124164 | doi = 10.1126/science.181.4096.223 | bibcode = 1973Sci...181..223A }}</ref>。特定のアミノ酸残基と、ポリペプチド鎖内におけるそれらの位置は、タンパク質のどの部分が密接に折り重なり、その三次元構造を形成するかを決定する要因となる。アミノ酸組成は配列ほど重要ではない<ref name="Voet_2016">{{cite book | title = Principles of Biochemistry | first1 = Donald | last1 = Voet | first2 = Judith G. | last2 = Voet | first3 = Charlotte W. | last3 = Pratt | name-list-format = vanc | publisher = Wiley | year = 2016 | edition = Fifth | isbn = 978-1-118-91840-1 }}</ref>。しかし、フォールディングの本質的な事実は、各タンパク質のアミノ酸配列が、本来の構造とその状態に到達するための経路の両方を指定する情報を含んでいることである。これは、ほぼ同じアミノ酸配列が常に同じように折りたためるということではない<ref>{{cite journal | vauthors = Alexander PA, He Y, Chen Y, Orban J, Bryan PN | title = The design and characterization of two proteins with 88% sequence identity but different structure and function | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 104 | issue = 29 | pages = 11963–8 | date = July 2007 | pmid = 17609385 | pmc = 1906725 | doi = 10.1073/pnas.0700922104 | bibcode = 2007PNAS..10411963A }}</ref>。類似したタンパク質でも、環境要因によってコンホメーションは異なり、どこで見つかったかによって異なる方法で折りたたまれる。 |
|||
=== 二次構造 === |
|||
100残基以上の大型タンパク質においては、フォールディングの過程は[[ジスルフィド結合]]の形成などを含むため複雑で、中間体が見られるものも多い。 |
|||
[[File:Alpha helix.png|thumb|332x332px|[[αヘリックス]]の螺旋形成]] |
|||
[[File:BetaPleatedSheetProtein.png|left|thumb|150x150px|主鎖内に水素結合を有する反平行[[Βシート|βプリーツシート]]]] |
|||
[[二次構造]]の形成は、タンパク質がその本来の構造を取るための、フォールディング・プロセスの最初のステップである。二次構造の特徴は[[αヘリックス]]や[[βシート]]として知られている構造で、[[ライナス・ポーリング]]によって最初に述べられたように、{{仮リンク|分子内|en|Intramolecular force|label=}}[[水素結合]]によって安定化されているために急速に折りたたまれる。分子内水素結合<!-- intramolecular hydrogen bonds -->の形成は、タンパク質の安定性に別の重要な貢献をしている<ref name="Rose">{{cite journal | vauthors = Rose GD, Fleming PJ, Banavar JR, Maritan A | title = A backbone-based theory of protein folding | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 103 | issue = 45 | pages = 16623–33 | date = November 2006 | pmid = 17075053 | pmc = 1636505 | doi = 10.1073/pnas.0606843103 | bibcode = 2006PNAS..10316623R | citeseerx = 10.1.1.630.5487 }}</ref>。αヘリックスは、{{仮リンク|主鎖|en|Backbone chain|label=}}の水素結合により螺旋状に形成される (右図参照)<ref name="Voet_2016" />。βプリーツシートは、水素結合を形成するために主鎖が折り曲がって形成される構造である (左図参照)。水素結合は、[[ペプチド結合]]のアミド水素とカルボニル酸素の間にある。逆平行βプリーツシートと平行βプリーツシートが存在し、平行βシートによって形成される傾斜水素結合と比較して、逆平行βプリーツシートの方が理想的な180度の角度で水素結合を形成するため水素結合の安定性が高くなっている<ref name="Voet_2016" />。 |
|||
=== 三次構造 === |
|||
なお生体内ではタンパク質の[[翻訳 (生物学)|翻訳]]とフォールディングが並行して起こるため、フォールディングの過程は試験管内と必ずしも同じではない。通常はフォールディングは翻訳よりも速いため、先に翻訳された[[N末端]]側から順次フォールディングが行われる<ref>{{cite journal |author=Fedorov AN |title=Cotranslational Protein Folding |journal=J. Biol. Chem. |volume=272 |issue=52 |pages=32715-32718 |year=1997 |doi=10.1074/jbc.272.52.32715}}</ref>。 |
|||
αヘリックスおよびβプリーツシートは、本質的に[[両親媒性分子|両親媒性]]であるか、または親水性部分と疎水性部分を含むことができる。二次構造のこの性質はタンパク質の三次構造形成を助け、親水性側がタンパク質を取り囲む[[水溶液|水溶性]]環境に面し、疎水性側がタンパク質の疎水性コアに面するようにフォールディングが起こる<ref name="Fersht_1999" />。二次構造は階層的に三次構造の形成によって取って代わられる。タンパク質の[[三次構造]]が形成され、疎水性相互作用によって安定化されると、2つの[[システイン]]残基間に形成された[[ジスルフィド結合]]の形で[[共有結合]]が存在することもある。タンパク質の三次構造には、単一のポリペプチド鎖が含まれるが、折りたたまれたポリペプチド鎖の追加の相互作用により四次構造が形成される<ref>{{cite web | url = http://www.nature.com/scitable/topicpage/protein-structure-14122136 | title = Protein Structure | publisher = Nature Education | accessdate = 2016-11-26 | work = Scitable }}</ref>。 |
|||
=== 四次構造 === |
|||
==実験的研究== |
|||
三次構造は、いくつかのタンパク質の[[四次構造]]の形成で取って代わられ、これには通常、すでに折りたたまれてたサブユニットの「集合」または「会合」を含む。言い換えれば、複数のポリペプチド鎖が相互作用して、完全に機能する四次タンパク質を形成する可能性がある<ref name="Voet_2016" />。 |
|||
タンパク質のフォールディングは通常1秒以内に起きるため、反応途中の溶液を一部取得して構造を解析するといったことは出来ず、分光学などの手法による研究が進められている。 |
|||
; 円二色性スペクトル |
|||
: タンパク質内で[[αヘリックス]]が形成されることで、溶液の[[旋光性]]が変化する。これを[[円二色性スペクトル]]測定により定量する。 |
|||
; トリプトファン蛍光 |
|||
: [[トリプトファン]]残基は芳香環を持つため紫外光を吸収して蛍光を発する。このときの蛍光は、タンパク質内部などの疎水性な環境では長波長にシフトする。そこでトリプトファンの蛍光波長を測定すれば、トリプトファン残基周辺の疎水性を測定でき、フォールディング進行の指標となる。なおトリプトファン残基は通常タンパク質に1〜2個しか無いので、複数の蛍光が重なってしまうといった問題は起こりにくい。 |
|||
; 重水素パルスラベル法 |
|||
: フォールディング途中のタンパク質を重水(D<sub>2</sub>O)中にごく短時間晒し、水素を重水素に置換する。このときタンパク質中の多くの水素はごく弱い酸性を持っているため、タンパク質の表面にある水素は重水素置換されるが、水素結合を形成している水素やタンパク質の内側にある水素は置換されない。この操作の後、重水素の位置をNMRで特定することで、その時点でのタンパク質の構造を推定できる。 |
|||
=== タンパク質フォールディングの推進力 === |
|||
==理論的研究== |
|||
[[File:225 Peptide Bond-01.jpg|thumb|263x263px|タンパク質の構造をまとめた全形態]] |
|||
タンパク質の構造はX線結晶構造解析やNMRを用いれば実験的に決定できるが、フォールディング過程を実験的に決めるのは困難である。このため理論的計算の研究も多くなされている。 |
|||
フォールディングは、主に[[疎水性相互作用]]、分子内[[水素結合]]の形成、[[ファンデルワールス力]]によって導かれる{{仮リンク|自発的過程|en|Spontaneous process|label=}}であり、[[配座エントロピー]]によって対抗を受ける<ref>{{cite book | first1 = Charlotte | last1 = Pratt | first2 = Kathleen | last2 = Cornely | name-list-format = vanc | chapter-url = http://www.wiley.com/college/pratt/0471393878/instructor/review/thermodynamics/7_relationship.html | chapter = Thermodynamics | title = Essential Biochemistry | publisher = Wiley | accessdate = 2016-11-26 | date = 2004 | isbn = 978-0-471-39387-0 | url-access = registration | url = https://archive.org/details/essentialbiochem00char }}</ref>。フォールディングのプロセスは、多くの場合、[[翻訳 (生物学)|共翻訳]]的(部分的な翻訳)に開始されるため、タンパク質の[[C末端]]部分がまだリボソームによって[[タンパク質生合成|合成]]されている間に、先に翻訳されたタンパク質の[[N末端]]が折りたたみ始まる<ref>{{cite journal|author=Fedorov AN|year=1997|title=Cotranslational Protein Folding|journal=J. Biol. Chem.|volume=272|issue=52|pages=32715-32718|doi=10.1074/jbc.272.52.32715}}</ref>。ただし、タンパク質分子は、生合成中または[[タンパク質生合成|生合成]]後に自発的に折りたたまれることがある<ref>{{Cite journal|date=2011-02-01|title=Folding at the birth of the nascent chain: coordinating translation with co-translational folding|url=|journal=Current Opinion in Structural Biology |volume=21 |issue=1 |pages=25–31 |doi=10.1016/j.sbi.2010.10.008 |pmid=21111607 |issn=0959-440X |last1=Zhang |first1=Gong |last2=Ignatova |first2=Zoya}}</ref>。これらの[[高分子]]は「[[自己集合|自分自身で折りたためる]]」と考えられるが、このプロセスは[[溶媒]] ([[水]]または[[脂質二重層]])<ref>{{cite journal | vauthors = van den Berg B, Wain R, Dobson CM, Ellis RJ | title = Macromolecular crowding perturbs protein refolding kinetics: implications for folding inside the cell | journal = The EMBO Journal | volume = 19 | issue = 15 | pages = 3870–5 | date = August 2000 | pmid = 10921869 | pmc = 306593 | doi = 10.1093/emboj/19.15.3870 }}</ref>、[[塩]]の濃度、[[水素イオン指数|pH]]、[[温度]]、補助因子および分子[[シャペロン]]の存在にも依存している。 |
|||
タンパク質は、可能な曲げ角度またはコンホメーションが制限されているため、フォールディング能力に制限がある。タンパク質フォールディングのこれらの許容角度は、[[ラマチャンドランプロット]]として知られている二次元プロットで表され、許容回転のpsiとphi角度で示される<ref>{{cite web | url = http://www.proteinstructures.com/Structure/Structure/Ramachandran-plot.html | title = Torsion Angles and the Ramachnadran Plot in Protein Structures | first = Salam | last = Al-Karadaghi | name-list-format = vanc | work = www.proteinstructures.com | accessdate = 2016-11-26}}</ref>。 |
|||
10〜80残基の小型タンパク質は既に計算機上でのフォールディングの再現に成功している<ref>{{cite journal |author=Kresten LL |title=How Fast-Folding Proteins Fold |journal=SCIENCE |volume=334 |pages=517-520 |year=2011 |doi=10.1126/science.1208351}}</ref>。しかし、大型のタンパク質で全原子の全相互作用の計算を実行することは困難である。このため、各アミノ酸残基のα炭素だけに着目したり、天然構造に存在する相互作用だけに基づいたポテンシャル(郷ポテンシャル)を用いるなどの近似計算が行われている。 |
|||
==== 疎水性効果 ==== |
|||
[[Image:Protein folding schematic.png|thumb|181x181px|{{仮リンク|疎水性凝集|en|Hydrophobic collapse|label=}}。コンパクトなフォールディング (右側)では、疎水性アミノ酸 (黒い球体として表示) が中央に向かって凝集し、水性環境から遮蔽された状態になる。|left]]タンパク質のフォールディングが自発的な反応であるためには、細胞内で熱力学的に有利でなければならない。タンパク質のフォールディングは自発的な反応であることが知られているため、負の[[ギブス自由エネルギー]]値をとる必要がある。タンパク質のフォールディングにおけるギブス自由エネルギーは[[エンタルピー]]と[[エントロピー]]に直接関係している<ref name="Voet_2016" />。負のデルタGが発生し、タンパク質のフォールディングが熱力学的に有利になるためには、エンタルピー、エントロピー、またはその両方が有利でなければならない。[[File:Molecular Dynamics Simulation of the Hydrophobic Solvation of Argon.webm|thumb|水分子が疎水性溶質の近くでより整然となるにつれて、エントロピーは減少する。|262x262px]] |
|||
水にさらされる疎水性側鎖の数を最小限にすることは、フォールディングプロセスの背後にある重要な推進力である<ref name="Pace">{{cite journal | vauthors = Pace CN, Shirley BA, McNutt M, Gajiwala K | title = Forces contributing to the conformational stability of proteins | journal = FASEB Journal | volume = 10 | issue = 1 | pages = 75–83 | date = January 1996 | pmid = 8566551 | doi = 10.1096/fasebj.10.1.8566551 }}</ref>。疎水効果とは、タンパク質の疎水性鎖がタンパク質の中心部 (親水性環境から離れる) に凝集する現象である<ref name="Voet_2016" />。水性環境下では、水分子はタンパク質の疎水性領域や側鎖の周囲に凝集し、秩序だった水分子の水殻<!-- water shells -->を形成する傾向がある<ref>{{cite journal | vauthors = Cui D, Ou S, Patel S | title = Protein-spanning water networks and implications for prediction of protein-protein interactions mediated through hydrophobic effects | journal = Proteins | volume = 82 | issue = 12 | pages = 3312–26 | date = December 2014 | pmid = 25204743 | doi = 10.1002/prot.24683 }}</ref>。疎水性領域を中心とした水分子の秩序は系内の秩序を増大させ、エントロピーの負の変化に寄与する (系内のエントロピーは減少する)。水分子はこれらの水分子ケージに固定されており、{{仮リンク|疎水性凝集|en|Hydrophobic collapse|label=}}、または疎水性基の内側へのフォールディングを促進する。疎水性凝集は、秩序だった水分子を解放する水ケージ<!-- water cages -->の破壊を介してシステムにエントロピーを導入する<ref name="Voet_2016" />。球状に折りたたまれたタンパク質のコア内で相互作用する多数の疎水性基は、膨大に蓄積されたファンデルワールス力 (特に[[ロンドン分散力]]) のため、折りたたみ後のタンパク質の安定性に大きく貢献する<ref name="Voet_2016" />。[[疎水効果|疎水性効果]]は、大きな疎水性領域を含む両親媒性分子を含む水性媒体が存在する場合にのみ、熱力学の推進力として存在する<ref>{{cite journal | vauthors = Tanford C | title = The hydrophobic effect and the organization of living matter | journal = Science | volume = 200 | issue = 4345 | pages = 1012–8 | date = June 1978 | pmid = 653353 | doi = 10.1126/science.653353 | bibcode = 1978Sci...200.1012T }}</ref>。水素結合の強さは環境に依存するため、疎水性コアに包まれた水素結合は、水性環境にさらされた水素結合よりも天然状態の安定性に寄与する<ref name="Deechongkit">{{cite journal | vauthors = Deechongkit S, Nguyen H, Powers ET, Dawson PE, Gruebele M, Kelly JW | title = Context-dependent contributions of backbone hydrogen bonding to beta-sheet folding energetics | journal = Nature | volume = 430 | issue = 6995 | pages = 101–5 | date = July 2004 | pmid = 15229605 | doi = 10.1038/nature02611 | bibcode = 2004Natur.430..101D | s2cid = 4315026 }}</ref>。 |
|||
球状の折りたたみを持つタンパク質では、疎水性アミノ酸はランダムに分布したり、一緒にクラスター化されるのではなく、一次配列に沿って散在する傾向がある<ref>{{cite journal |last1=Irbäck |first1=Anders |last2=Sandelin |first2=Erik |title=On Hydrophobicity Correlations in Protein Chains |journal=Biophysical Journal |date=November 2000 |volume=79 |issue=5 |pages=2252–2258 |doi=10.1016/S0006-3495(00)76472-1|pmid=11053106 |pmc=1301114 |arxiv=cond-mat/0010390 |bibcode=2000BpJ....79.2252I }}</ref><ref>{{cite journal |last1=Irbäck |first1=A. |last2=Peterson |first2=C. |last3=Potthast |first3=F. |title=Evidence for nonrandom hydrophobicity structures in protein chains. |journal=Proceedings of the National Academy of Sciences |date=3 September 1996 |volume=93 |issue=18 |pages=9533–9538 |doi=10.1073/pnas.93.18.9533|pmid=8790365 |pmc=38463 |arxiv=chem-ph/9512004 |bibcode=1996PNAS...93.9533I }}</ref>。しかし、[[天然変性タンパク質|天然変成]]傾向のある新生([[de novo|''de novo'']])タンパク質は<ref>{{cite journal |last1=Wilson |first1=Benjamin A. |last2=Foy |first2=Scott G. |last3=Neme |first3=Rafik |last4=Masel |first4=Joanna |title=Young genes are highly disordered as predicted by the preadaptation hypothesis of de novo gene birth |journal=Nature Ecology & Evolution |date=24 April 2017 |volume=1 |issue=6 |pages=0146–146 |doi=10.1038/s41559-017-0146|pmc=5476217 |pmid=28642936 }}</ref><ref>{{cite journal |last1=Willis |first1=Sara |last2=Masel |first2=Joanna |title=Gene Birth Contributes to Structural Disorder Encoded by Overlapping Genes |journal=Genetics |date=September 2018 |volume=210 |issue=1 |pages=303–313 |doi=10.1534/genetics.118.301249 |pmid=30026186 |pmc=6116962}}</ref>、一次配列に沿って疎水性アミノ酸がクラスター化するという逆のパターンを示す<ref>{{cite journal |last1=Foy |first1=Scott G. |last2=Wilson |first2=Benjamin A. |last3=Bertram |first3=Jason |last4=Cordes |first4=Matthew H. J. |last5=Masel |first5=Joanna |title=A Shift in Aggregation Avoidance Strategy Marks a Long-Term Direction to Protein Evolution |journal=Genetics |date=April 2019 |volume=211 |issue=4 |pages=1345–1355 |doi=10.1534/genetics.118.301719 |pmid=30692195 |pmc=6456324}}</ref>。 |
|||
==脚注== |
|||
==== シャペロン ==== |
|||
[[File:PDB 1gme EBI.jpg|thumb|小型真核生物の[[熱ショックタンパク質]]の例]] |
|||
[[分子シャペロン]] ({{lang-en-short|molecular chaperone}}) は、生体内 (''[[in vivo]]'') で他のタンパク質を正しく折りたたむのに役立つタンパク質の一種である。シャペロンは、すべての細胞内区画に存在し、ポリペプチド鎖と相互作用して、タンパク質の本来の三次元コンホメーションを形成できるようにする。ただし、シャペロン自体は、それらが補助しているタンパク質の最終構造には含まれていない<ref name="Dobson_2003" />。シャペロンは、新生ポリペプチドがリボソームによって合成されている場合でも、フォールディングを助けることができる<ref name="Hartl_1996" />。分子シャペロンは、結合することによって機能し、フォールディング経路でタンパク質の不安定な構造を安定化させるが、シャペロンには、それらが補助しているタンパク質の正しい本来の構造を知るために必要な情報は含まれておらず、むしろ、シャペロンは、誤った折りたたみ構造を防ぐことによって機能する<ref name="Hartl_1996">{{cite journal | vauthors = Hartl FU | title = Molecular chaperones in cellular protein folding | journal = Nature | volume = 381 | issue = 6583 | pages = 571–9 | date = June 1996 | pmid = 8637592 | doi = 10.1038/381571a0 | bibcode = 1996Natur.381..571H | s2cid = 4347271 }}</ref>。このように、シャペロンは実際には、本来の構造に向かうフォールディング経路に関与する個々のステップの速度を増加させることはなく、その代わりに、適切な中間体の探索を遅くする可能性のあるポリペプチド鎖の不要な凝集を減らすことで機能し、ポリペプチド鎖が正しいコンホメーションをとるためのより効率的な経路を提供する<ref name="Dobson_2003" />。シャペロンは、フォールディング触媒と混同されるべきではなく、フォールディング経路の遅いステップを実際に触媒する。フォールディング触媒の例は、タンパク質ジスルフィド異性化酵素(protein disulfide isomerases)およびペプチジルプロリル異性化酵素(peptidyl-prolyl isomerases)があり、それぞれジスルフィド結合の形成またはシスおよびトランス立体異性体間の相互変換に関与している可能性がある<ref name="Hartl_1996" />。シャペロンは、生体内でのタンパク質フォールディングのプロセスにおいて重要であることが示されている。なぜなら、シャペロンは、タンパク質が「生物学的に適切な」状態になるために十分効率的に適切な配列とコンホメーションをとるのに必要な手助けタンパク質に提供するからである<ref name="Hartl_2011">{{cite journal | vauthors = Hartl FU, Bracher A, Hayer-Hartl M | title = Molecular chaperones in protein folding and proteostasis | journal = Nature | volume = 475 | issue = 7356 | pages = 324–32 | date = July 2011 | pmid = 21776078 | doi = 10.1038/nature10317 | s2cid = 4337671 }}</ref>。これは、[[In vitro|''in vitro'']]で行われたタンパク質フォールディング実験で実証されたように、ポリペプチド鎖は理論的にはシャペロンの助けなしにその本来の構造に折りたたむことができることを意味するものであるが<ref name="Hartl_2011" />、このプロセスはあまりにも非効率的であるか、または遅すぎて生物学的システムには存在しないことが判明している。したがって、シャペロンは生体内でのタンパク質のフォールディングに必要である。シャペロンは、本来構造の形成を助ける役割に加えて、タンパク質の輸送、分解、さらには外部変性因子にさらされた[[変性|変性タンパク質]]が正しい本来の構造にリフォールディング(再折りたたみ)する機会を与えるなど、様々な役割に関与していることが明らかになっている<ref>{{cite journal | vauthors = Kim YE, Hipp MS, Bracher A, Hayer-Hartl M, Hartl FU | title = Molecular chaperone functions in protein folding and proteostasis | journal = Annual Review of Biochemistry | volume = 82 | pages = 323–55 | year = 2013 | pmid = 23746257 | doi = 10.1146/annurev-biochem-060208-092442 }}</ref>。 |
|||
完全に変性したタンパク質は、三次構造と二次構造の両方を欠いており、いわゆる[[ランダムコイル]]として存在している。特定の条件下では、一部のタンパク質は再折りたたみ(リフォールド)する可能性があるが、多くの場合、変性は不可逆的である<ref name="Shortle">{{cite journal | vauthors = Shortle D | title = The denatured state (the other half of the folding equation) and its role in protein stability | journal = FASEB Journal | volume = 10 | issue = 1 | pages = 27–34 | date = January 1996 | pmid = 8566543 | doi = 10.1096/fasebj.10.1.8566543 }}</ref>。細胞は、熱変性の影響からタンパク質を守るために、[[熱ショックタンパク質]] (シャペロンの一種) として知られる[[酵素]]を用いて、他のタンパク質のフォールディングや折りたたまれた状態の維持を助けている。熱ショックタンパク質は、[[細菌]]からヒトにいたるまで、調査したすべての種で発見されており、非常に早い段階で進化し、重要な機能を持っていることが示唆されている。一部のタンパク質は、シャペロンの助けを借りて他のタンパク質との相互作用によりフォールディングが中断されないように個々のタンパク質を分離するか、誤って折りたたまれたタンパク質を展開して本来の正しい構造にリフォールディングをしない限り、細胞内では全く折りたたまれないことがある<ref name="Lee_2005">{{cite journal | vauthors = Lee S, Tsai FT | title = Molecular chaperones in protein quality control | journal = Journal of Biochemistry and Molecular Biology | volume = 38 | issue = 3 | pages = 259–65 | year = 2005 | pmid = 15943899 | doi = 10.5483/BMBRep.2005.38.3.259 | doi-access = free }}</ref>。この機能は、[[不溶性]]のアモルファス凝集体への[[沈殿]]のリスクを防ぐために非常に重要である。タンパク質の変性や天然状態の破壊に関与する外部要因には、温度、外部磁場 (電場、磁場)<ref name="ojeda">{{cite journal | vauthors = Ojeda-May P, Garcia ME | title = Electric field-driven disruption of a native beta-sheet protein conformation and generation of a helix-structure | journal = Biophysical Journal | volume = 99 | issue = 2 | pages = 595–9 | date = July 2010 | pmid = 20643079 | pmc = 2905109 | doi = 10.1016/j.bpj.2010.04.040 | bibcode = 2010BpJ....99..595O }}</ref>、分子クラウディング<ref name="berg">{{cite journal | vauthors = van den Berg B, Ellis RJ, Dobson CM | title = Effects of macromolecular crowding on protein folding and aggregation | journal = The EMBO Journal | volume = 18 | issue = 24 | pages = 6927–33 | date = December 1999 | pmid = 10601015 | pmc = 1171756 | doi = 10.1093/emboj/18.24.6927 }}</ref>、さらにはタンパク質のフォールディングに大きな影響を与える可能性のある空間の制限 (すなわち閉じ込め) が含まれる<ref>{{cite journal | vauthors = Ellis RJ | title = Molecular chaperones: assisting assembly in addition to folding | journal = Trends in Biochemical Sciences | volume = 31 | issue = 7 | pages = 395–401 | date = July 2006 | pmid = 16716593 | doi = 10.1016/j.tibs.2006.05.001 }}</ref>。高濃度の[[溶質]]、極端な[[水素イオン指数|pH]]、機械的な力、化学的変性剤の存在も同様にタンパク質の変性に寄与する。これらの個々の要因はストレスとしてまとめて分類される。シャペロンは、細胞ストレス時には高濃度で存在し、変性タンパク質や誤って折りたたまれたタンパク質だけでなく、新生タンパク質の適切なフォールディングを助けることが示されている<ref name="Dobson_2003" />。 |
|||
一部の条件下では、タンパク質は生化学的に機能する形に折りたたまれない。細胞が通常生存する温度範囲よりも高い温度や低い温度では、[[耐熱性|熱的に不安定]]なタンパク質は折りたたまれなかったり、変性する (これが煮沸すると[[卵白]]が不透明になる理由である)。しかし、タンパク質の熱安定性が一定であるとは限らない。例えば、[[超好熱始原菌|超好熱性細菌]]は122 °Cの高温で生育することが確認されているが<ref>{{cite journal | vauthors = Takai K, Nakamura K, Toki T, Tsunogai U, Miyazaki M, Miyazaki J, Hirayama H, Nakagawa S, Nunoura T, Horikoshi K | title = Cell proliferation at 122 degrees C and isotopically heavy CH4 production by a hyperthermophilic methanogen under high-pressure cultivation | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 105 | issue = 31 | pages = 10949–54 | date = August 2008 | pmid = 18664583 | pmc = 2490668 | doi = 10.1073/pnas.0712334105 | bibcode = 2008PNAS..10510949T }}</ref>、これにはもちろん、重要なタンパク質やタンパク質集合体の完全な補体が、その温度以上で安定している必要がある。 |
|||
[[大腸菌]]は[[バクテリオファージT4]]の宿主であり、ファージにコードされたgp31タンパク質は、大腸菌の[[シャペロン|シャペロンタンパク質]] {{仮リンク|GroES|en|GroES|label=}}と機能的に相同であるように見え、感染時にバクテリオファージT4[[ウイルス]]粒子の組み立てにおいてそれを置き換えることができる<ref name="Marusich1998">Marusich EI, Kurochkina LP, Mesyanzhinov VV. Chaperones in bacteriophage T4 assembly. Biochemistry (Mosc). 1998;63(4):399-406</ref>。GroESと同様に、gp31は{{仮リンク|GroEL|en|GroEL|label=}}シャペロニンとの安定な複合体を形成する。これはバクテリオファージT4メジャー・キャプシド・タンパク質gp23の''in vivo''でのフォールディングおよび組み立てに絶対的に必要である<ref name="Marusich1998" />。 |
|||
== タンパク質の誤ったフォールディングと神経変性疾患 == |
|||
{{main|プロテオパチー}} |
|||
タンパク質は、通常の天然状態を得られない場合、ミスフォールド(誤った折りたたみ)をしていると考えられる。これは、アミノ酸配列の突然変異または外部要因による通常のフォールディングプロセスの混乱が原因である可能性がある<ref name="Chaudhuri_2006" />。ミスフォールドされたタンパク質は、通常、クロスβ構造として知られる超分子配列で組織化された[[βシート]]を含んでいる。これらのβシートが豊富に含んだ集合体は、非常に安定し極めて不溶性であり、一般にタンパク質分解に対して耐性がある<ref name="Soto_2006">{{cite journal | vauthors = Soto C, Estrada L, Castilla J | title = Amyloids, prions and the inherent infectious nature of misfolded protein aggregates | journal = Trends in Biochemical Sciences | volume = 31 | issue = 3 | pages = 150–5 | date = March 2006 | pmid = 16473510 | doi = 10.1016/j.tibs.2006.01.002 }}</ref>。これらの{{仮リンク|フィブリル|en|Fibril|label=}}状集合体の構造的安定性は、βストランド間の主鎖水素結合によって形成されたタンパク質モノマー間の広範な相互作用によってもたらされる<ref name="Soto_2006" />。タンパク質のミスフォールディングは、他のタンパク質の凝集体または[[オリゴマー]]へのさらなるミスフォールディングや蓄積を引き起こす可能性がある。細胞内で凝集したタンパク質のレベルが上昇すると、変性疾患や細胞死を引き起こす可能性のある[[アミロイド]]様構造の形成につながる<ref name="Chaudhuri_2006">{{cite journal | vauthors = Chaudhuri TK, Paul S | title = Protein-misfolding diseases and chaperone-based therapeutic approaches | journal = The FEBS Journal | volume = 273 | issue = 7 | pages = 1331–49 | date = April 2006 | pmid = 16689923 | doi = 10.1111/j.1742-4658.2006.05181.x }}</ref>。アミロイドは、分子間水素結合を含む線維状構造であり、極めて不溶性で、転換されたタンパク質の集合体から作られる<ref name="Chaudhuri_2006" />。そのため、プロテアソーム経路では、凝集する前にミスフォールドしたタンパク質を分解するのに十分な効率が得られない場合がある。ミスフォールドされたタンパク質は、互いに相互作用して構造化された凝集体を形成し、分子間相互作用を通じて毒性を獲得する可能性がある<ref name="Chaudhuri_2006" />。 |
|||
凝集タンパク質は、[[クロイツフェルト・ヤコブ病]]、[[牛海綿状脳症]] (狂牛病) などの[[プリオン]]関連疾患、[[アルツハイマー病]]および{{仮リンク|家族性アミロイド|en|Familial amyloid polyneuropathy|label=}}心筋症または多神経症などの[[アミロイド]]関連疾患<ref name="pmid12560553">{{cite journal | vauthors = Hammarström P, Wiseman RL, Powers ET, Kelly JW | title = Prevention of transthyretin amyloid disease by changing protein misfolding energetics | journal = Science | volume = 299 | issue = 5607 | pages = 713–6 | date = January 2003 | pmid = 12560553 | doi = 10.1126/science.1079589 | bibcode = 2003Sci...299..713H | s2cid = 30829998 }}</ref>、ならびに[[ハンチントン病]]および[[パーキンソン病]]などの細胞内凝集性疾患と関連している<ref name="Selkoe:03" /><ref name="ChitiDobson">{{cite journal | vauthors = Chiti F, Dobson CM | title = Protein misfolding, functional amyloid, and human disease | journal = Annual Review of Biochemistry | volume = 75 | pages = 333–66 | year = 2006 | pmid = 16756495 | doi = 10.1146/annurev.biochem.75.101304.123901 }}</ref>。これらの加齢性変性疾患は、不溶性の細胞外凝集体および(または)クロスβ[[アミロイド]]{{仮リンク|原繊維|en|Fibril|label=}}を含む細胞内封入体へのミスフォールドタンパク質の凝集に関連している。これは凝集体が原因なのか、それとも単にタンパク質の恒常性の喪失、合成、フォールディング、凝集、タンパク質代謝回転のバランスを反映しているだけなのかは完全には明らかではない。最近、[[欧州医薬品庁]]は、トランスサイレチンアミロイド疾患の治療のための{{仮リンク|タファミディス|en|Tafamidis|label=}}(Tafamidis)またはビンダケル (Vyndaqel; 四量体トランスサイレチンの動態安定化剤) の使用を承認した。このことは、ヒトのアミロイド疾患において、アミロイド原繊維形成プロセス (原繊維自体ではなく) が、有糸分裂後の組織の変性を引き起こすことを示唆している<ref name="pmid16359163">{{cite journal | vauthors = Johnson SM, Wiseman RL, Sekijima Y, Green NS, Adamski-Werner SL, Kelly JW | title = Native state kinetic stabilization as a strategy to ameliorate protein misfolding diseases: a focus on the transthyretin amyloidoses | journal = Accounts of Chemical Research | volume = 38 | issue = 12 | pages = 911–21 | date = December 2005 | pmid = 16359163 | doi = 10.1021/ar020073i }}</ref>。フォールディングや機能ではなく、ミスフォールディングや過度の分解は、[[アンチトリプシン]]関連[[肺気腫]]、[[嚢胞性線維症]]、[[リソソーム蓄積症]]などの多くの[[プロテオパチー]]疾患を引き起こし、機能の喪失が障害の根源となっている。後者の疾患を修正するためにタンパク質補充療法が歴史的に使用されてきたが、新たなアプローチは、{{仮リンク|薬理シャペロン|en|Pharmacological chaperone|label=}}を使用して変異タンパク質を折りたたんで機能させる状態にすることが挙げられる。 |
|||
== タンパク質のフォールディングを研究するための実験技術 == |
|||
タンパク質のフォールディングに関する推論は、突然変異研究{{Enlink|Phi value analysis|英語版}}を通じて行うことができるが、タンパク質のフォールディングを研究するための実験技術は、通常、タンパク質の{{仮リンク|段階的アンフォールディング|en|Equilibrium unfolding|label=}}やフォールディングと、標準的な非結晶学的技術を用いたコンホメーション変化の観察に依存している。 |
|||
=== X線結晶構造解析 === |
|||
[[File:X ray diffraction.png|thumb|[[X線結晶構造解析]]のステップ|321x321px]] |
|||
[[X線結晶構造解析]]は、折りたたまれたタンパク質の三次元構造を解読するための、効率的で重要な方法の一つである<ref name="Cowtan_2001">{{cite encyclopedia | url = http://people.bu.edu/mfk/restricted566/phaseproblem.pdf | title = Phase Problem in X-ray Crystallography, and Its Solution | last = Cowtan | first = Kevin | name-list-format = vanc | date = 2001 | encyclopedia = Encyclopedia of Life Sciences |publisher=Macmillan Publishers Ltd, Nature Publishing Group|accessdate=November 3, 2016}}</ref>。X線結晶構造解析を行うためには、対象となるタンパク質が結晶格子内に配置されている必要がある。タンパク質を結晶格子内に配置するためには、結晶化に適した溶媒を用意し、溶液中で過飽和状態の純粋なタンパク質を得て、溶液中で結晶を析出させる必要がある<ref>{{cite book | url=https://books.google.com/books?id=Jobr7svN0IIC&pg=PR5 |title = Principles of Protein X-Ray Crystallography | last = Drenth | first = Jan | name-list-format = vanc | date = 2007-04-05 | publisher = Springer Science & Business Media | isbn = 978-0-387-33746-3 }}</ref>。タンパク質が結晶化されると、X線ビームは結晶格子を介して集中することができ、ビームを回折したり、様々な方向にビームを外側に向けて発射したりする。これらの出射ビーム<!-- exiting beams -->は、内包されたタンパク質の特定の三次元構成に相関している。X線は、タンパク質の結晶格子内の個々の原子を取り囲む電子雲と特異的に相互作用し、識別可能な回折パターンを生成する<ref name="Fersht_1999">{{cite book |url=https://books.google.com/books?id=QdpZz_ahA5UC&pg=PR20 |title=Structure and Mechanism in Protein Science: A Guide to Enzyme Catalysis and Protein Folding | last = Fersht | first = Alan | name-list-format = vanc | date = 1999 | publisher = Macmillan | isbn = 978-0-7167-3268-6 }}</ref>。電子密度雲をX線の振幅を関連付けることによってのみ、このパターンを読み取ることができ、この方法を複雑にする位相や位相角の仮定を導くことになる<ref>{{cite journal |doi=10.1107/S0907444903017815 |title=The phase problem |journal=Acta Crystallographica Section D |volume=59 |issue=11 |pages=1881–90 |year=2003 |last1=Taylor |first1=Garry | name-list-format = vanc |pmid=14573942 |doi-access=free }}</ref>。[[フーリエ変換]]という数学的基礎によって確立された関係がなければ、「位相問題」は回折パターン予測を非常に困難にする<ref name="Fersht_1999" />。{{仮リンク|多重同型置換|en|Multiple isomorphous replacement|label=}}のような新しい方法では、重金属イオンの存在を利用してX線をより予測可能な方法で回折させ、関与する変数の数を減らして位相の問題を解決している<ref name="Cowtan_2001" />。 |
|||
=== 蛍光分光法 === |
|||
[[蛍光分光法]]は、タンパク質の折りたたみ状態を調べるための高感度な手法である。フェニルアラニン(Phe)、チロシン(Tyr)、トリプトファン(Trp)の3つのアミノ酸は固有の蛍光特性を持つが、TyrとTrpのみが[[量子収率]]が高く、良好な蛍光シグナルが得られるため、実験的に使用されている。TrpとTyrは共に280 nmの波長で励起されるのに対し、Trpだけは295 nmの波長で励起される。それらの芳香族性のため、TrpとTyr残基は、タンパク質の疎水性コア、2つのタンパク質ドメイン間の界面、またはオリゴマータンパク質のサブユニット間の界面に、完全または部分的に埋もれていることがよくある。この無極性環境では、これらの残基は高い量子収率を持ち、それゆえ高い蛍光強度を示す。タンパク質の3次構造や4次構造が破壊されると、これらの側鎖は溶媒の親水性環境にさらされるようになり、量子収率が低下して低い蛍光強度になる。Trp残基については、その最大蛍光発光の波長も環境に依存する。 |
|||
蛍光分光法は、変性剤の値の関数として、蛍光発光強度または最大発光波長の変動を測定することにより、タンパク質の{{仮リンク|平衡アンフォールディング|en|equilibrium unfolding|label=}}を特徴付けるために使用される<ref name="pmid=26607240">{{cite journal | vauthors = Bedouelle H | title = Principles and equations for measuring and interpreting protein stability: From monomer to tetramer | journal = Biochimie | volume = 121 | pages = 29–37 | date = February 2016 | pmid = 26607240 | doi = 10.1016/j.biochi.2015.11.013 }}</ref><ref>{{cite journal | vauthors = Monsellier E, Bedouelle H | title = Quantitative measurement of protein stability from unfolding equilibria monitored with the fluorescence maximum wavelength | journal = Protein Engineering, Design & Selection | volume = 18 | issue = 9 | pages = 445–56 | date = September 2005 | pmid = 16087653 | doi = 10.1093/protein/gzi046 | doi-access = free }}</ref>。ここで変性剤は、化学分子 (尿素、塩酸グアニジニウム)、温度、pH、圧力などである。互いに異なるが離散的なタンパク質の状態、すなわち天然状態、中間状態、アンフォールド状態の間の平衡は、変性剤の値に依存するため、それらの平衡混合物の全体的な蛍光シグナルもこの値に依存する。このようにして、全体的なタンパク質シグナルを変性剤の値に関連付けるプロファイルが得られる<ref>{{cite journal | vauthors = Park YC, Bedouelle H | title = Dimeric tyrosyl-tRNA synthetase from Bacillus stearothermophilus unfolds through a monomeric intermediate. A quantitative analysis under equilibrium conditions | journal = The Journal of Biological Chemistry | volume = 273 | issue = 29 | pages = 18052–9 | date = July 1998 | pmid = 9660761 | doi = 10.1074/jbc.273.29.18052 | doi-access = free }}</ref><ref>{{cite journal | vauthors = Ould-Abeih MB, Petit-Topin I, Zidane N, Baron B, Bedouelle H | title = Multiple folding states and disorder of ribosomal protein SA, a membrane receptor for laminin, anticarcinogens, and pathogens | journal = Biochemistry | volume = 51 | issue = 24 | pages = 4807–21 | date = June 2012 | pmid = 22640394 | doi = 10.1021/bi300335r }}</ref>。平衡アンフォールディングのプロファイルは、アンフォールディングの中間体を検出し、識別することを可能にする。Hugues Bedouelleによって一般的な方程式が開発され、そのようなプロファイルから、ホモマーまたはヘテロマーのタンパク質のアンフォールディング平衡を特徴づける熱力学的パラメータを三量体まで、および潜在的には四量体まで得ることができた<ref name="pmid=26607240" />。蛍光分光法は、{{仮リンク|ストップフロー|en|Stopped-flow|label=}}のような高速混合装置と組み合わせて、タンパク質のフォールディング動態を測定し<ref>{{cite journal | vauthors = Royer CA | title = Probing protein folding and conformational transitions with fluorescence | journal = Chemical Reviews | volume = 106 | issue = 5 | pages = 1769–84 | date = May 2006 | pmid = 16683754 | doi = 10.1021/cr0404390 }}</ref>、{{仮リンク|シェブロンプロット|en|Chevron plot|label=}}を生成し、Phi値分析{{Enlink|Phi value analysis|英語版}}を導出することができる。 |
|||
=== 円偏光二色性 === |
|||
{{Main|円偏光二色性}} |
|||
[[円偏光二色性]]は、タンパク質のフォールディングを研究するための最も一般的で基本的なツールの一つである。[[円二色性]]分光法は、[[円偏光]]の吸収を測定する。タンパク質では、αヘリックスやβシートなどの構造は不斉であるため、このような光を吸収する。この光の吸収は、タンパク質アンサンブルの折りたたみ度合いのマーカーとして機能する。この技術は、変性剤濃度または[[温度]]関数としてこの吸収の変化を測定することにより、タンパク質の{{仮リンク|平衡アンフォールディング|en|Equilibrium unfolding|label=}}を測定するために使用されてきた。変性溶解は、タンパク質のm値、または変性剤依存性と同様にアンフォールディングの[[自由エネルギー]]を測定する。[[温度]]溶解は、タンパク質の{{仮リンク|変性温度|en|Denaturation midpoint|label=}}(Tm)を測定する<ref name="pmid=26607240" />。蛍光分光法に関しては、円二色性分光法をストップフローなどの高速混合装置と組み合わせて、タンパク質のフォールディング[[反応速度論|動態]]を測定し、{{仮リンク|シェブロンプロット|en|Chevron plot|label=}}を生成することができる。 |
|||
=== タンパク質の振動円二色性 === |
|||
最近開発されたタンパク質の{{仮リンク|振動円二色性|en|Vibrational circular dichroism|label=}} (vibrational circular dichroism; VCD) 技術は現在、[[フーリエ変換]](FT)機器を用いており、非常に大きなタンパク質分子でも、溶液中のタンパク質構造を決定する強力な手段を提供する。このようなタンパク質のVCD研究は、タンパク質結晶の[[X線回折]]、重水(D<sub>2</sub>O)中のタンパク質溶液の[[FT-IR]]データ、または第一原理量子計算と組み合わせて、[[円偏光二色性]](CD)からは得られない明確な構造決定を提供することがよくある{{Citation needed|date=April 2010}}。 |
|||
=== タンパク質核磁気共鳴分光法 === |
|||
{{main|en:Protein NMR}} |
|||
タンパク質のフォールディングは、[[NMR分光法]]を使用して日常的に研究されており、例えば、その天然状態でのタンパク質の主鎖アミドプロトンの水素-重水素交換{{Enlink|Hydrogen–deuterium exchange|英語版}}を監視することで、タンパク質の残基固有の安定性と全体的な安定性の両方を提供する<ref>Beatrice M.P. Huyghues-Despointes, C. Nick Pace, S. Walter Englander, and J. Martin Scholtz. "Measuring the Conformational Stability of a Protein by Hydrogen Exchange." Methods in Molecular Biology. Kenneth P. Murphy Ed. Humana Press, Totowa, New Jersey, 2001. pp. 69–92</ref>。 |
|||
=== 二重偏光干渉法 === |
|||
{{Main|en:Dual polarisation interferometry}} |
|||
{{仮リンク|二重偏光干渉法|en|Dual-polarization interferometry|label=}}は、分子層の光学特性を測定する表面ベースの技術である。タンパク質のフォールディングを特徴づけるために使用される場合、タンパク質の単層の全体的なサイズとその密度をサブ・オングストーム分解能でリアルタイムに測定することによって[[コンホメーション]]を測定するが<ref name="CrossFreeman2008">{{cite book |doi=10.1002/9780470061565.hbb055 |chapter=Dual Polarization Interferometry: A Real-Time Optical Technique for Measuring (Bio)molecular Orientation, Structure and Function at the Solid/Liquid Interface |title=Handbook of Biosensors and Biochips |year=2008 |last1=Cross |first1=Graham H. |last2=Freeman |first2=Neville J. |last3=Swann |first3=Marcus J. | name-list-format = vanc |isbn=978-0-470-01905-4 }}</ref>、タンパク質フォールディングの速度論のリアルタイム測定は、~10 Hzよりも遅いプロセスに限られている。[[円偏光二色性]]と同様に、フォールディングのための刺激は変性剤または温度である可能性がある。 |
|||
=== 高時間分解能でのフォールディングの研究 === |
|||
タンパク質のフォールディングの研究は、高速で時間分解技術の開発によって近年、大きく進展した。実験者は、折りたたまれていないタンパク質のサンプルのフォールディングを迅速に誘発し、結果として生じる{{仮リンク|タンパク質動力学|en|Protein dynamics|label=}}を観察する。使用されている高速化技術には、[[中性子散乱]]<ref name="Callaway">{{cite journal | vauthors = Bu Z, Cook J, Callaway DJ | title = Dynamic regimes and correlated structural dynamics in native and denatured alpha-lactalbumin | journal = Journal of Molecular Biology | volume = 312 | issue = 4 | pages = 865–73 | date = September 2001 | pmid = 11575938 | doi = 10.1006/jmbi.2001.5006 }}</ref>、超高速溶液混合、光化学的手法、レーザー{{仮リンク|温度ジャンプ|en|Temperature jump|label=}}分光法などがある。これらの技術の開発に貢献した多くの科学者の中には、Jeremy Cook, Heinrich Roder, Harry Gray, Martin Gruebele, Brian Dyer, William Eaton, Sheena Radford, Chris Dobson, Alan Fersht, Bengt Nölting, Lars Konermannがいる。 |
|||
=== タンパク質分解 === |
|||
[[タンパク質分解]] (proteolysis) は、広範囲の溶液条件下 (例: {{仮リンク|高速並列タンパク質分解|en|Fast parallel proteolysis|label=}}) でアンフォールドされた画分を探索するために日常的に使用されている<ref name="Minde">{{cite journal | vauthors = Minde DP, Maurice MM, Rüdiger SG | title = Determining biophysical protein stability in lysates by a fast proteolysis assay, FASTpp | journal = PLOS ONE | volume = 7 | issue = 10 | pages = e46147 | year = 2012 | pmid = 23056252 | pmc = 3463568 | doi = 10.1371/journal.pone.0046147 | bibcode = 2012PLoSO...746147M }}</ref><ref name="Park">{{cite journal | vauthors = Park C, Marqusee S | title = Pulse proteolysis: a simple method for quantitative determination of protein stability and ligand binding | journal = Nature Methods | volume = 2 | issue = 3 | pages = 207–12 | date = March 2005 | pmid = 15782190 | doi = 10.1038/nmeth740 | s2cid = 21364478 }}</ref>。 |
|||
=== 単一分子力分光法 === |
|||
単離されたタンパク質やシャペロンを持つタンパク質のフォールディング機構を理解するために、[[光ピンセット]]やAFMなどの単一分子力技術 (Single-molecule force spectroscopy) が用いられてきた<ref name="pmid24001118">{{cite journal | vauthors = Mashaghi A, Kramer G, Lamb DC, Mayer MP, Tans SJ | title = Chaperone action at the single-molecule level | journal = Chemical Reviews | volume = 114 | issue = 1 | pages = 660–76 | date = January 2014 | pmid = 24001118 | doi = 10.1021/cr400326k }}</ref>。光ピンセットは、単一のタンパク質分子をC末端とN末端から引き伸ばし、それを展開して、その後のリフォールディングを研究するために使用されてきた<ref>{{cite journal | vauthors = Jagannathan B, Marqusee S | title = Protein folding and unfolding under force | journal = Biopolymers | volume = 99 | issue = 11 | pages = 860–9 | date = November 2013 | pmid = 23784721 | pmc = 4065244 | doi = 10.1002/bip.22321 }}</ref>。この手法により、単一分子レベルでフォールディング率を測定できる。例えば、光ピンセットは最近、血液凝固に関与するタンパク質のフォールディングとアンフォールディングの研究に応用されている。[[ヴォン・ヴィレブランド因子]] (von Willebrand factor (vWF)) は、血液凝固プロセスに不可欠な役割を持つタンパク質である。単一分子の光ピンセット測定を使用して、カルシウム結合vWFが血液中でせん断力センサーとして働くことを発見した。せん断力はvWFのA2ドメインのアンフォールディングにつながり、そのリフォールディング速度はカルシウムの存在下で劇的に向上する<ref>{{cite journal | vauthors = Jakobi AJ, Mashaghi A, Tans SJ, Huizinga EG | title = Calcium modulates force sensing by the von Willebrand factor A2 domain | journal = Nature Communications | volume = 2 | pages = 385 | date = July 2011 | pmid = 21750539 | pmc = 3144584 | doi = 10.1038/ncomms1385 | bibcode = 2011NatCo...2..385J }}</ref>。最近では、単純な src SH3 ドメインが、力を受けると複数のアンフォールディング経路にアクセスすることも明らかにされた<ref>{{cite journal | vauthors = Jagannathan B, Elms PJ, Bustamante C, Marqusee S | title = Direct observation of a force-induced switch in the anisotropic mechanical unfolding pathway of a protein | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 109 | issue = 44 | pages = 17820–5 | date = October 2012 | pmid = 22949695 | pmc = 3497811 | doi = 10.1073/pnas.1201800109 | bibcode = 2012PNAS..10917820J }}</ref>。 |
|||
=== ビオチン標識 === |
|||
ビオチン標識 (Biotin painting) は、(未)フォールディング・タンパク質の状態特異的な細胞スナップショットを可能にする。 ビオチン標識は、予測される[[天然変性タンパク質|天然変成タンパク質]]への偏りを示している<ref name="Biotinylation by proximity labelling favours unfolded proteins">{{cite journal | vauthors = Minde DP, Ramakrishna M, Lilley KS | title = Biotinylation by proximity labelling favours unfolded proteins | journal = bioRxiv | year = 2018 | doi = 10.1101/274761 | doi-access = free }}</ref>。 |
|||
== タンパク質フォールディングの計算科学的研究 == |
|||
タンパク質フォールディングの[[計算科学]]的研究には、タンパク質の安定性、速度論、および構造予測に関連する3つの主要な側面が含まれる。以下の最近のレビューは、タンパク質フォールディングに利用可能な計算手法をまとめたものである<ref>{{cite journal | vauthors = Compiani M, Capriotti E | title = Computational and theoretical methods for protein folding | journal = Biochemistry | volume = 52 | issue = 48 | pages = 8601–24 | date = December 2013 | pmid = 24187909 | doi = 10.1021/bi4001529 }}</ref>。 |
|||
=== レヴィンタールのパラドックス === |
|||
1969年、{{仮リンク|サイラス・レヴィンタール|en|Cyrus Levinthal|label=}}は、折りたたまれていないポリペプチド鎖の自由度が非常に大きいため、タンパク質分子は天文学的な数の起こりうるコンホメーションを持っていることに着目した。彼の論文の中で、3<sup>300</sup>または10<sup>143</sup>という推定がなされている<ref>{{Cite web|url=https://en.wikibooks.org/wiki/Structural_Biochemistry/Proteins/Protein_Folding#The_Levinthal_Paradox|title=Structural Biochemistry/Proteins/Protein Folding - Wikibooks, open books for an open world|website=en.wikibooks.org|accessdate=2016-11-05}}</ref>。{{仮リンク|レヴィンタールのパラドックス|en|Levinthal's paradox|label=}}は、タンパク質が起こりうるすべてのコンホメーションを順次サンプリングして折りたたまれた場合、たとえコンホメーションが高速 ([[ナノ秒]]または[[ピコ秒]]スケール) でサンプリングされたとしても、天文学的な量の時間がかかるという観察に基づく思考実験である<ref>{{cite journal | last = Levinthal | first = Cyrus | name-list-format = vanc | year = 1968 | title = Are there pathways for protein folding? | url = http://www.biochem.wisc.edu/courses/biochem704/Reading/Levinthal1968.pdf | journal = Journal de Chimie Physique et de Physico-Chimie Biologique | volume = 65 | pages = 44–45 | archiveurl = https://web.archive.org/web/20090902211239/http://www.biochem.wisc.edu/courses/biochem704/Reading/Levinthal1968.pdf | archivedate = 2009-09-02 | doi = 10.1051/jcp/1968650044 | bibcode = 1968JCP....65...44L }}</ref>。タンパク質はこれよりもはるかに速く折りたたまれるという観測に基づいて、レヴィンタールは、ランダムなコンホメーション探索は発生しないため、タンパク質は一連の準安定な[[反応中間体|中間状態]]を経て折りたたまれなければならないと提案した。 |
|||
=== タンパク質フォールディングのエネルギー地形 === |
|||
[[File:Folding funnel schematic.svg|thumb|286x286px|折りたたまれていないポリペプチド鎖が本来の構造を取るようになるエネルギー・ファンネル(漏斗)。]] |
|||
フォールディング中のタンパク質の{{仮リンク|配位空間|en|Configuration space (physics)|label=}}は、[[エネルギー地形]] (energy landscape) として可視化できる。Joseph Bryngelsonと{{仮リンク|Peter Wolynes|en|Peter Wolynes|label=}}によると、タンパク質は[[最小フラストレーション原理]]に従っており、自然に進化したタンパク質はフォールディング時のエネルギー地形を最適化し<ref name="bryngelson">{{cite journal | vauthors = Bryngelson JD, Onuchic JN, Socci ND, Wolynes PG | title = Funnels, pathways, and the energy landscape of protein folding: a synthesis | journal = Proteins | volume = 21 | issue = 3 | pages = 167–95 | date = March 1995 | pmid = 7784423 | doi = 10.1002/prot.340210302 | arxiv = chem-ph/9411008 | s2cid = 13838095 }}</ref>、自然はタンパク質の折りたたみ状態が十分に安定するようにアミノ酸配列を選択していることを意味している。さらに、折りたたまれた状態の獲得は、十分に高速なプロセスにならなければならない。自然がタンパク質のフラストレーションのレベルを減らしたとしても、タンパク質のエネルギー地形における局所的な最小値が存在することからもわかるように、ある程度のフラストレーションは今のところ残っている。 |
|||
これらの進化的に選択された配列の結果として、タンパク質は、天然状態に向かうグローバルな「ファンネル(漏斗)状のエネルギー地形」({{仮リンク|José Onuchic|en|José Onuchic|label=}}の造語<ref>{{cite journal | author1 = Leopold PE | author2 = Montal M | author3 = Onuchic JN | authorlink3 = José Onuchic | title = Protein folding funnels: a kinetic approach to the sequence-structure relationship | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 89 | issue = 18 | pages = 8721–5 | date = September 1992 | pmid = 1528885 | pmc = 49992 | doi = 10.1073/pnas.89.18.8721 | bibcode = 1992PNAS...89.8721L }}</ref>) を持っていると一般的に考えられている。この「{{仮リンク|フォールディング・ファンネル|en|Folding funnel|label=}}」地形により、タンパク質は、単一のメカニズムに限定されるのではなく、多数の経路や中間体のいずれかを介して天然状態にフォールディングできる。この理論は、モデルタンパク質の計算シミュレーション({{仮リンク|格子タンパク質|en|Lattice protein|label=}})と実験的研究の両方で支持されており<ref name="bryngelson" />、[[タンパク質構造予測|タンパク質構造の予測]]と{{仮リンク|タンパク質構造設計|en|Protein design|label=}}のための方法を改善するために使用されてきた<ref name="bryngelson" />。平準化自由エネルギー地形によるタンパク質フォールディングの説明も、熱力学第2法則と合致している<ref>{{cite journal |doi=10.1016/j.physa.2008.12.004 |title=Protein folding as an evolutionary process |journal=Physica A: Statistical Mechanics and Its Applications | volume = 388 | issue = 6 | pages = 851–62 | year = 2009 | last1 = Sharma | first1 = Vivek | last2 = Kaila | first2 = Ville R.I. | last3 = Annila | first3 = Arto | name-list-format = vanc | bibcode = 2009PhyA..388..851S }}</ref>。物理的には、エネルギー地形を、地理的な地形のように、単に最大値、[[鞍点]]、最小値、ファンネルを持った可視化可能なポテンシャル曲面や全エネルギー曲面の観点から考えることは、あるいはいくらか誤解を招く可能性がある。妥当な記述は、実際には、多様体が様々なより複雑な位相形態をとる可能性のある高次元の位相空間である<ref name="Robson_2008">{{cite book |doi=10.1016/S0079-6603(08)00405-4 |pmid=19121702 |chapter=Protein Folding Revisited |title=Molecular Biology of Protein Folding, Part B |volume=84 |pages=161–202 |series=Progress in Molecular Biology and Translational Science |year=2008 |last1=Robson |first1=Barry |last2=Vaithilingam |first2=Andy | name-list-format = vanc |isbn=978-0-12-374595-8 }}</ref>。 |
|||
折りたたまれていないポリペプチド鎖は、ファンネルの一番上に位置し、折りたたまれていないバリエーションの数が最も多く、エネルギー状態は高も高くなる。このようなエネルギー地形は、初期の可能性が多数あることを示しているが、可能なのは単一の天然状態のみである。しかし、それは可能な多くのフォールディング経路を明らかにしていない。同じ正確なタンパク質の異なる分子は、同じ天然構造に到達する限り、わずかに異なるフォールディング経路をたどり、異なる低エネルギー中間体を探すことができる場合がある<ref name="Dill_2012">{{cite journal | vauthors = Dill KA, MacCallum JL | title = The protein-folding problem, 50 years on | journal = Science | volume = 338 | issue = 6110 | pages = 1042–6 | date = November 2012 | pmid = 23180855 | doi = 10.1126/science.1219021 | bibcode = 2012Sci...338.1042D | s2cid = 5756068 }}</ref>。異なる経路は、各経路の熱力学的な有利性に応じて、異なる利用頻度を持つ可能性がある。これは、ある経路が他の経路よりも熱力学的に有利であることが分かった場合、本来の構造を追求するために、より頻繁に使用される可能性が高いことを意味する<ref name="Dill_2012" />。タンパク質が折りたたみ始め、さまざまなコンホメーションをとると、常に以前よりも熱力学的に有利な構造を求め、エネルギーファンネルを通過し続けることになる。二次構造の形成は、タンパク質内の安定性の向上を強く示しており、ポリペプチド骨格によって想定される二次構造の一つの組み合わせだけが最低のエネルギーを持ち、ゆえにタンパク質は天然状態で存在することになる<ref name="Dill_2012" />。ポリペプチドが折りたたみを開始すると形成される最初の構造の中には、αヘリックスおよびβターンがあり、αヘリックスはわずか100ナノ秒で形成され、βターンは1マイクロ秒で形成される<ref name="Dobson_2003">{{cite journal | vauthors = Dobson CM | title = Protein folding and misfolding | journal = Nature | volume = 426 | issue = 6968 | pages = 884–90 | date = December 2003 | pmid = 14685248 | doi = 10.1038/nature02261 | bibcode = 2003Natur.426..884D | s2cid = 1036192 }}</ref>。 |
|||
エネルギー・ファンネル地形には、特定のタンパク質の遷移状態が見られる[[鞍点]]が存在する<ref name="Dobson_2003" />。エネルギー・ファンネル図の遷移状態とは、タンパク質が最終的に本来の構造をとることを想定した場合、そのタンパク質のすべての分子がとらなければならないコンホメーションである。どのタンパク質も、最初に遷移状態を通過しなければ、本来の構造をとることはできない<ref name="Dobson_2003" />。遷移状態は、単なる別の中間段階ではなく、天然状態の変化形または未熟な形と呼ぶことができる<ref name="Fersht_2000">{{cite journal | vauthors = Fersht AR | title = Transition-state structure as a unifying basis in protein-folding mechanisms: contact order, chain topology, stability, and the extended nucleus mechanism | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 97 | issue = 4 | pages = 1525–9 | date = February 2000 | pmid = 10677494 | pmc = 26468 | doi = 10.1073/pnas.97.4.1525 | bibcode = 2000PNAS...97.1525F }}</ref>。遷移状態のフォールディングは律速であることが示されており、それが本来のフォールディングよりも高いエネルギー状態で存在しているとしても、本来の構造と極めて類似する。遷移状態の中には、タンパク質が折りたたむことができる核(足場)となる構造が存在しており、核の上に構造が段階的に完成してゆく「凝縮核形成」<!-- nucleation condensation -->と呼ばれるプロセスによって形成される<ref name="Fersht_2000" />。 |
|||
=== タンパク質フォールディングのモデリング === |
|||
[[File:ACBP MSM from Folding@home.tiff|right|thumb|350px|[[Folding@home]]は、ここに示すような[[隠れマルコフモデル|マルコフ状態モデル]]を使用して、タンパク質が初期のランダムなコイル状の状態 (左) から自然の三次元構造 (右) に凝集するときに取ることができる起こりうる形状やフォールディング経路をモデル化する。]] |
|||
計算による[[タンパク質構造予測]]のための[[De novo|デ・ノボ]] (''de novo'') または[[Ab initio|第一原理]] (''ap initio'') 的手法は、どちらもタンパク質フォールディングの実験的研究に関連しているが、厳密には区別されるものである。[[分子動力学法]] (MD) は、タンパク質のフォールディングと動力学を[[In silico|イン・シリコ]] (in silico) で研究するための重要なツールである<ref name="Rizzuti">{{cite journal | vauthors = Rizzuti B, Daggett V | title = Using simulations to provide the framework for experimental protein folding studies | journal = Archives of Biochemistry and Biophysics | volume = 531 | issue = 1–2 | pages = 128–35 | date = March 2013 | pmid = 23266569 | pmc = 4084838 | doi = 10.1016/j.abb.2012.12.015 }}</ref>。最初の平衡フォールディング・シミュレーションは、暗黙の溶媒モデルと[[アンブレラサンプリング法|アンブレラ・サンプリング法]]を用いて行われた<ref>{{cite journal | vauthors = Schaefer M, Bartels C, Karplus M | title = Solution conformations and thermodynamics of structured peptides: molecular dynamics simulation with an implicit solvation model | journal = Journal of Molecular Biology | volume = 284 | issue = 3 | pages = 835–48 | date = December 1998 | pmid = 9826519 | doi = 10.1006/jmbi.1998.2172 }}</ref>。計算コストが高いため、明示的な水を用いた第一原理計算によるフォールディング・シミュレーションは、ペプチドや非常に小さなタンパク質に限定される<ref>{{cite web|url=http://www.cs.ucl.ac.uk/staff/d.jones/t42morph.html|title=Fragment-based Protein Folding Simulations|first=David|last=Jones|name-list-format=vanc|publisher=University College London|accessdate=2020-10-12}}</ref><ref>{{cite web|url=http://www.biomolecular-modeling.com/Abalone/Protein-folding.html|title=Protein folding|format=by Molecular Dynamics|accessdate=2020-10-12|publisher=}}</ref>。より大きなタンパク質のMDシミュレーションは、実験的な構造の動力学、または、その高温アンフォールディングに限定される。小さなサイズのタンパク質 (約50残基以上) のフォールディングのような長時間のフォールディングプロセス (約1ミリ秒を超える) は、{{仮リンク|粗視化モデル|en|Coarse-grained modeling|label=}}を用いて解析することができる<ref>{{cite journal | vauthors = Kmiecik S, Gront D, Kolinski M, Wieteska L, Dawid AE, Kolinski A | title = Coarse-Grained Protein Models and Their Applications | journal = Chemical Reviews | volume = 116 | issue = 14 | pages = 7898–936 | date = July 2016 | pmid = 27333362 | doi = 10.1021/acs.chemrev.6b00163 | doi-access = free }}</ref><ref name="Kmiecik">{{cite journal | vauthors = Kmiecik S, Kolinski A | title = Characterization of protein-folding pathways by reduced-space modeling | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 104 | issue = 30 | pages = 12330–5 | date = July 2007 | pmid = 17636132 | pmc = 1941469 | doi = 10.1073/pnas.0702265104 | bibcode = 2007PNAS..10412330K }}</ref><ref name="teritfix">{{cite journal | vauthors = Adhikari AN, Freed KF, Sosnick TR | title = De novo prediction of protein folding pathways and structure using the principle of sequential stabilization | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 109 | issue = 43 | pages = 17442–7 | date = October 2012 | pmid = 23045636 | pmc = 3491489 | doi = 10.1073/pnas.1209000109 | bibcode = 2012PNAS..10917442A }}</ref>。 |
|||
[[スタンフォード大学]]の[[Vijay Pande]]氏のグループが作成した100ペタFLOP級の[[分散コンピューティング]]プロジェクト [[Folding@home]] は、ボランティアのパーソナルコンピュータの[[CPU]]と[[Graphics Processing Unit|GPU]]のアイドル処理時間を利用して、タンパク質のフォールディングをシミュレーションする。このプロジェクトは、タンパク質のフォールディングのミスフォールディング(誤った折りたたみ)を理解し、疾患研究のための[[創薬]][[医薬品設計|ドラッグデザイン]]を加速することを目的としている。 |
|||
D. E. Shaw Research社{{Enlink|D. E. Shaw Research|英語版}}のカスタム[[ASIC]]と相互接続を中心に設計・構築された超並列スーパーコンピュータ[[アントン (スーパーコンピュータ)|Anton]](アントン)で、長時間の連続軌道シミュレーションが実行された。Antonを使用して実行されたシミュレーションの公開された最長の結果は、355KでのNTL9の2.936ミリ秒のシミュレーションである<ref name="pmid22034434">{{cite journal | vauthors = Lindorff-Larsen K, Piana S, Dror RO, Shaw DE | title = How fast-folding proteins fold | journal = Science | volume = 334 | issue = 6055 | pages = 517–20 | date = October 2011 | pmid = 22034434 | doi = 10.1126/science.1208351 | bibcode = 2011Sci...334..517L | s2cid = 27988268 }}</ref>。 |
|||
== 注釈と出典 == |
|||
{{脚注ヘルプ}} |
{{脚注ヘルプ}} |
||
{{Reflist|33em}} |
|||
<references/> |
|||
==参考文献== |
== 参考文献 == |
||
* 藤博幸 編「タンパク質の立体構造入門」 講談社 (2010) |
* 藤博幸 編「タンパク質の立体構造入門」 講談社 (2010) |
||
* Voet & Voet "Biochemistry" 3rd edition, Wiley (2004) |
* Voet & Voet "Biochemistry" 3rd edition, Wiley (2004) |
||
== 関連項目 == |
== 関連項目 == |
||
* [[アントン (スーパーコンピュータ)]] |
|||
* [[シャペロン]] |
* [[シャペロン]] |
||
* {{仮リンク|シェブロン・プロット|en|Chevron plot|label=}} |
|||
* [[Folding@Home]] |
|||
* {{仮リンク|変性中間点|en|Denaturation midpoint|label=}} |
|||
* [[:en:Fast parallel proteolysis]] |
|||
* {{Ill2|ダウンヒル・フォールディング|en|Downhill folding|label=}} |
|||
{{タンパク質}} |
|||
* {{仮リンク|高速並列タンパク質分解|en|Fast parallel proteolysis|label=}} |
|||
{{三次構造}} |
|||
* {{Ill2|フォールディング (化学)|en|Folding (chemistry)|label=}} |
|||
* [[Folding@home|Folding@Home]] |
|||
* [[Foldit]] - コンピュータ・ゲーム |
|||
* [[力場 (化学)]] |
|||
* {{Ill2|Pt-barrel|en|Pt-barrel|label=}} |
|||
* {{Ill2|タンパク質動力学|en|Protein dynamics|label=}} |
|||
* {{Ill2|タンパク質ミスフォールディング周期的増幅|en|Protein misfolding cyclic amplification|label=}} (PMCA法) |
|||
* {{Ill2|タンパク質構造予測ソフトウェア|en|Protein structure prediction software|label=}} |
|||
* [[プロテオパチー]] |
|||
* [[Rosetta@home]] |
|||
* [[分子力学モデリング用ソフトの比較]] |
|||
* {{仮リンク|統計的ポテンシャル|en|Statistical potential|label=}} |
|||
* {{仮リンク|時間分解質量分析法|en|Time-resolved mass spectrometry|label=}} |
|||
== 外部リンク == |
|||
* [http://fold.it/portal/info/science FoldIt - Folding Protein Game] |
|||
* [https://foldingathome.org/about/the-foldinghome-consortium/ Folding@Home] |
|||
* [http://boinc.bakerlab.org/rosetta Rosetta@Home] |
|||
* [http://www.worldcommunitygrid.org/research/proteome/overview.do Human Proteome Folding Project] |
|||
* [http://www.scfbio-iitd.res.in/bhageerath/bhageerath_h.jsp BHAGEERATH-H: Protein tertiary structure prediction server] |
|||
{{Template:タンパク質}} |
|||
{{Template:三次構造}} |
|||
{{Template:生体分子構造}} |
|||
{{DEFAULTSORT:ふおおるていんく}} |
{{DEFAULTSORT:ふおおるていんく}} |
||
[[Category:タンパク質]] |
[[Category:タンパク質]] |
||
[[Category:タンパク質構造]] |
[[Category:タンパク質構造]] |
||
2020年10月18日 (日) 11:06時点における版


タンパク質フォールディング (英語: Protein folding) とは、タンパク質鎖がその本来の三次元構造、通常は生物学的に機能するコンホメーション(立体構造)を、迅速かつ再現性のある方法で獲得する物理的なプロセスである。これは、ポリペプチドがランダムコイルからその特徴的で機能的な三次元構造に折りたたまれる物理的な過程である[1]。それぞれのタンパク質は、mRNAの配列からアミノ酸の直鎖に翻訳されるとき、折りたたまれていないポリペプチドまたはランダムコイルとして存在する。そのポリペプチドは、安定した (長続きする) 立体構造を欠いている (第1図の左側)。そのポリペプチド鎖がリボソームで合成されていく過程で、直鎖が三次元構造に折りたたまれる。フォールディングは、ポリペプチド鎖の翻訳中でも始まる。アミノ酸は互いに相互作用して、明確に定義された三次元構造、つまり天然状態として知られている折りたたまれたタンパク質 (図の右側) を生成する。結果として生じる三次元構造は、アミノ酸配列または一次構造 (アンフィンセンのドグマ) によって決定される[2]。
タンパク質が機能を発揮するために正しい三次元構造が不可欠であるが、機能性タンパク質の一部は折りたたまれていない状態のままになっていることがあり[3]、ゆえにタンパク質動力学が重要となる。本来の構造に折りたたまれないと、一般に不活性なタンパク質が生成されるが、場合によっては、誤って折りたたまれたタンパク質の機能が改変されたり、毒性のある機能性を持つこともある。いくつかの神経変性疾患やその他の疾患は、誤って折りたたまれたタンパク質によって形成されたアミロイド原線維の蓄積に起因すると考えられている[4]。多くのアレルギーは、一部のタンパク質が正しく折りたたまれていないことが原因で、免疫系が特定のタンパク質構造に対する抗体を産生しないために引き起こされる[5]。
タンパク質の変性は、折りたたまれた状態から折りたたまれていない状態に移行するプロセスである。これは、調理、火傷、プロテオパチー、その他の状況で起こる。
フォールディング・プロセスの所要時間は、目的のタンパク質によって劇的に異なる。細胞外で調べたとき、最も遅く折りたたまれるタンパク質は、主にプロリン異性化のために折りたたまれるのに数分から数時間を要し、プロセスが完了するまでにチェックポイントのようないくつかの中間状態を通過する必要がある[6]。一方、長さが100アミノ酸までの非常に小さなシングルドメインタンパク質は、通常、1回のステップで折りたたむことができる[7]。時間スケールはミリ秒が一般的で、非常に速い既知のタンパク質のフォールディング反応は数マイクロ秒以内に完了する[8]。
タンパク質のフォールディング過程
一次構造
タンパク質の一次構造である直線的なアミノ酸配列は、その本来のコンホメーションを決定する[9]。特定のアミノ酸残基と、ポリペプチド鎖内におけるそれらの位置は、タンパク質のどの部分が密接に折り重なり、その三次元構造を形成するかを決定する要因となる。アミノ酸組成は配列ほど重要ではない[10]。しかし、フォールディングの本質的な事実は、各タンパク質のアミノ酸配列が、本来の構造とその状態に到達するための経路の両方を指定する情報を含んでいることである。これは、ほぼ同じアミノ酸配列が常に同じように折りたためるということではない[11]。類似したタンパク質でも、環境要因によってコンホメーションは異なり、どこで見つかったかによって異なる方法で折りたたまれる。
二次構造
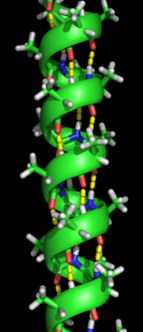

二次構造の形成は、タンパク質がその本来の構造を取るための、フォールディング・プロセスの最初のステップである。二次構造の特徴はαヘリックスやβシートとして知られている構造で、ライナス・ポーリングによって最初に述べられたように、分子内水素結合によって安定化されているために急速に折りたたまれる。分子内水素結合の形成は、タンパク質の安定性に別の重要な貢献をしている[12]。αヘリックスは、主鎖の水素結合により螺旋状に形成される (右図参照)[10]。βプリーツシートは、水素結合を形成するために主鎖が折り曲がって形成される構造である (左図参照)。水素結合は、ペプチド結合のアミド水素とカルボニル酸素の間にある。逆平行βプリーツシートと平行βプリーツシートが存在し、平行βシートによって形成される傾斜水素結合と比較して、逆平行βプリーツシートの方が理想的な180度の角度で水素結合を形成するため水素結合の安定性が高くなっている[10]。
三次構造
αヘリックスおよびβプリーツシートは、本質的に両親媒性であるか、または親水性部分と疎水性部分を含むことができる。二次構造のこの性質はタンパク質の三次構造形成を助け、親水性側がタンパク質を取り囲む水溶性環境に面し、疎水性側がタンパク質の疎水性コアに面するようにフォールディングが起こる[13]。二次構造は階層的に三次構造の形成によって取って代わられる。タンパク質の三次構造が形成され、疎水性相互作用によって安定化されると、2つのシステイン残基間に形成されたジスルフィド結合の形で共有結合が存在することもある。タンパク質の三次構造には、単一のポリペプチド鎖が含まれるが、折りたたまれたポリペプチド鎖の追加の相互作用により四次構造が形成される[14]。
四次構造
三次構造は、いくつかのタンパク質の四次構造の形成で取って代わられ、これには通常、すでに折りたたまれてたサブユニットの「集合」または「会合」を含む。言い換えれば、複数のポリペプチド鎖が相互作用して、完全に機能する四次タンパク質を形成する可能性がある[10]。
タンパク質フォールディングの推進力
フォールディングは、主に疎水性相互作用、分子内水素結合の形成、ファンデルワールス力によって導かれる自発的過程であり、配座エントロピーによって対抗を受ける[15]。フォールディングのプロセスは、多くの場合、共翻訳的(部分的な翻訳)に開始されるため、タンパク質のC末端部分がまだリボソームによって合成されている間に、先に翻訳されたタンパク質のN末端が折りたたみ始まる[16]。ただし、タンパク質分子は、生合成中または生合成後に自発的に折りたたまれることがある[17]。これらの高分子は「自分自身で折りたためる」と考えられるが、このプロセスは溶媒 (水または脂質二重層)[18]、塩の濃度、pH、温度、補助因子および分子シャペロンの存在にも依存している。
タンパク質は、可能な曲げ角度またはコンホメーションが制限されているため、フォールディング能力に制限がある。タンパク質フォールディングのこれらの許容角度は、ラマチャンドランプロットとして知られている二次元プロットで表され、許容回転のpsiとphi角度で示される[19]。
疎水性効果

タンパク質のフォールディングが自発的な反応であるためには、細胞内で熱力学的に有利でなければならない。タンパク質のフォールディングは自発的な反応であることが知られているため、負のギブス自由エネルギー値をとる必要がある。タンパク質のフォールディングにおけるギブス自由エネルギーはエンタルピーとエントロピーに直接関係している[10]。負のデルタGが発生し、タンパク質のフォールディングが熱力学的に有利になるためには、エンタルピー、エントロピー、またはその両方が有利でなければならない。
水にさらされる疎水性側鎖の数を最小限にすることは、フォールディングプロセスの背後にある重要な推進力である[20]。疎水効果とは、タンパク質の疎水性鎖がタンパク質の中心部 (親水性環境から離れる) に凝集する現象である[10]。水性環境下では、水分子はタンパク質の疎水性領域や側鎖の周囲に凝集し、秩序だった水分子の水殻を形成する傾向がある[21]。疎水性領域を中心とした水分子の秩序は系内の秩序を増大させ、エントロピーの負の変化に寄与する (系内のエントロピーは減少する)。水分子はこれらの水分子ケージに固定されており、疎水性凝集、または疎水性基の内側へのフォールディングを促進する。疎水性凝集は、秩序だった水分子を解放する水ケージの破壊を介してシステムにエントロピーを導入する[10]。球状に折りたたまれたタンパク質のコア内で相互作用する多数の疎水性基は、膨大に蓄積されたファンデルワールス力 (特にロンドン分散力) のため、折りたたみ後のタンパク質の安定性に大きく貢献する[10]。疎水性効果は、大きな疎水性領域を含む両親媒性分子を含む水性媒体が存在する場合にのみ、熱力学の推進力として存在する[22]。水素結合の強さは環境に依存するため、疎水性コアに包まれた水素結合は、水性環境にさらされた水素結合よりも天然状態の安定性に寄与する[23]。
球状の折りたたみを持つタンパク質では、疎水性アミノ酸はランダムに分布したり、一緒にクラスター化されるのではなく、一次配列に沿って散在する傾向がある[24][25]。しかし、天然変成傾向のある新生(de novo)タンパク質は[26][27]、一次配列に沿って疎水性アミノ酸がクラスター化するという逆のパターンを示す[28]。
シャペロン

分子シャペロン (英: molecular chaperone) は、生体内 (in vivo) で他のタンパク質を正しく折りたたむのに役立つタンパク質の一種である。シャペロンは、すべての細胞内区画に存在し、ポリペプチド鎖と相互作用して、タンパク質の本来の三次元コンホメーションを形成できるようにする。ただし、シャペロン自体は、それらが補助しているタンパク質の最終構造には含まれていない[29]。シャペロンは、新生ポリペプチドがリボソームによって合成されている場合でも、フォールディングを助けることができる[30]。分子シャペロンは、結合することによって機能し、フォールディング経路でタンパク質の不安定な構造を安定化させるが、シャペロンには、それらが補助しているタンパク質の正しい本来の構造を知るために必要な情報は含まれておらず、むしろ、シャペロンは、誤った折りたたみ構造を防ぐことによって機能する[30]。このように、シャペロンは実際には、本来の構造に向かうフォールディング経路に関与する個々のステップの速度を増加させることはなく、その代わりに、適切な中間体の探索を遅くする可能性のあるポリペプチド鎖の不要な凝集を減らすことで機能し、ポリペプチド鎖が正しいコンホメーションをとるためのより効率的な経路を提供する[29]。シャペロンは、フォールディング触媒と混同されるべきではなく、フォールディング経路の遅いステップを実際に触媒する。フォールディング触媒の例は、タンパク質ジスルフィド異性化酵素(protein disulfide isomerases)およびペプチジルプロリル異性化酵素(peptidyl-prolyl isomerases)があり、それぞれジスルフィド結合の形成またはシスおよびトランス立体異性体間の相互変換に関与している可能性がある[30]。シャペロンは、生体内でのタンパク質フォールディングのプロセスにおいて重要であることが示されている。なぜなら、シャペロンは、タンパク質が「生物学的に適切な」状態になるために十分効率的に適切な配列とコンホメーションをとるのに必要な手助けタンパク質に提供するからである[31]。これは、in vitroで行われたタンパク質フォールディング実験で実証されたように、ポリペプチド鎖は理論的にはシャペロンの助けなしにその本来の構造に折りたたむことができることを意味するものであるが[31]、このプロセスはあまりにも非効率的であるか、または遅すぎて生物学的システムには存在しないことが判明している。したがって、シャペロンは生体内でのタンパク質のフォールディングに必要である。シャペロンは、本来構造の形成を助ける役割に加えて、タンパク質の輸送、分解、さらには外部変性因子にさらされた変性タンパク質が正しい本来の構造にリフォールディング(再折りたたみ)する機会を与えるなど、様々な役割に関与していることが明らかになっている[32]。
完全に変性したタンパク質は、三次構造と二次構造の両方を欠いており、いわゆるランダムコイルとして存在している。特定の条件下では、一部のタンパク質は再折りたたみ(リフォールド)する可能性があるが、多くの場合、変性は不可逆的である[33]。細胞は、熱変性の影響からタンパク質を守るために、熱ショックタンパク質 (シャペロンの一種) として知られる酵素を用いて、他のタンパク質のフォールディングや折りたたまれた状態の維持を助けている。熱ショックタンパク質は、細菌からヒトにいたるまで、調査したすべての種で発見されており、非常に早い段階で進化し、重要な機能を持っていることが示唆されている。一部のタンパク質は、シャペロンの助けを借りて他のタンパク質との相互作用によりフォールディングが中断されないように個々のタンパク質を分離するか、誤って折りたたまれたタンパク質を展開して本来の正しい構造にリフォールディングをしない限り、細胞内では全く折りたたまれないことがある[34]。この機能は、不溶性のアモルファス凝集体への沈殿のリスクを防ぐために非常に重要である。タンパク質の変性や天然状態の破壊に関与する外部要因には、温度、外部磁場 (電場、磁場)[35]、分子クラウディング[36]、さらにはタンパク質のフォールディングに大きな影響を与える可能性のある空間の制限 (すなわち閉じ込め) が含まれる[37]。高濃度の溶質、極端なpH、機械的な力、化学的変性剤の存在も同様にタンパク質の変性に寄与する。これらの個々の要因はストレスとしてまとめて分類される。シャペロンは、細胞ストレス時には高濃度で存在し、変性タンパク質や誤って折りたたまれたタンパク質だけでなく、新生タンパク質の適切なフォールディングを助けることが示されている[29]。
一部の条件下では、タンパク質は生化学的に機能する形に折りたたまれない。細胞が通常生存する温度範囲よりも高い温度や低い温度では、熱的に不安定なタンパク質は折りたたまれなかったり、変性する (これが煮沸すると卵白が不透明になる理由である)。しかし、タンパク質の熱安定性が一定であるとは限らない。例えば、超好熱性細菌は122 °Cの高温で生育することが確認されているが[38]、これにはもちろん、重要なタンパク質やタンパク質集合体の完全な補体が、その温度以上で安定している必要がある。
大腸菌はバクテリオファージT4の宿主であり、ファージにコードされたgp31タンパク質は、大腸菌のシャペロンタンパク質 GroESと機能的に相同であるように見え、感染時にバクテリオファージT4ウイルス粒子の組み立てにおいてそれを置き換えることができる[39]。GroESと同様に、gp31はGroELシャペロニンとの安定な複合体を形成する。これはバクテリオファージT4メジャー・キャプシド・タンパク質gp23のin vivoでのフォールディングおよび組み立てに絶対的に必要である[39]。
タンパク質の誤ったフォールディングと神経変性疾患
タンパク質は、通常の天然状態を得られない場合、ミスフォールド(誤った折りたたみ)をしていると考えられる。これは、アミノ酸配列の突然変異または外部要因による通常のフォールディングプロセスの混乱が原因である可能性がある[40]。ミスフォールドされたタンパク質は、通常、クロスβ構造として知られる超分子配列で組織化されたβシートを含んでいる。これらのβシートが豊富に含んだ集合体は、非常に安定し極めて不溶性であり、一般にタンパク質分解に対して耐性がある[41]。これらのフィブリル状集合体の構造的安定性は、βストランド間の主鎖水素結合によって形成されたタンパク質モノマー間の広範な相互作用によってもたらされる[41]。タンパク質のミスフォールディングは、他のタンパク質の凝集体またはオリゴマーへのさらなるミスフォールディングや蓄積を引き起こす可能性がある。細胞内で凝集したタンパク質のレベルが上昇すると、変性疾患や細胞死を引き起こす可能性のあるアミロイド様構造の形成につながる[40]。アミロイドは、分子間水素結合を含む線維状構造であり、極めて不溶性で、転換されたタンパク質の集合体から作られる[40]。そのため、プロテアソーム経路では、凝集する前にミスフォールドしたタンパク質を分解するのに十分な効率が得られない場合がある。ミスフォールドされたタンパク質は、互いに相互作用して構造化された凝集体を形成し、分子間相互作用を通じて毒性を獲得する可能性がある[40]。
凝集タンパク質は、クロイツフェルト・ヤコブ病、牛海綿状脳症 (狂牛病) などのプリオン関連疾患、アルツハイマー病および家族性アミロイド心筋症または多神経症などのアミロイド関連疾患[42]、ならびにハンチントン病およびパーキンソン病などの細胞内凝集性疾患と関連している[4][43]。これらの加齢性変性疾患は、不溶性の細胞外凝集体および(または)クロスβアミロイド原繊維を含む細胞内封入体へのミスフォールドタンパク質の凝集に関連している。これは凝集体が原因なのか、それとも単にタンパク質の恒常性の喪失、合成、フォールディング、凝集、タンパク質代謝回転のバランスを反映しているだけなのかは完全には明らかではない。最近、欧州医薬品庁は、トランスサイレチンアミロイド疾患の治療のためのタファミディス(Tafamidis)またはビンダケル (Vyndaqel; 四量体トランスサイレチンの動態安定化剤) の使用を承認した。このことは、ヒトのアミロイド疾患において、アミロイド原繊維形成プロセス (原繊維自体ではなく) が、有糸分裂後の組織の変性を引き起こすことを示唆している[44]。フォールディングや機能ではなく、ミスフォールディングや過度の分解は、アンチトリプシン関連肺気腫、嚢胞性線維症、リソソーム蓄積症などの多くのプロテオパチー疾患を引き起こし、機能の喪失が障害の根源となっている。後者の疾患を修正するためにタンパク質補充療法が歴史的に使用されてきたが、新たなアプローチは、薬理シャペロンを使用して変異タンパク質を折りたたんで機能させる状態にすることが挙げられる。
タンパク質のフォールディングを研究するための実験技術
タンパク質のフォールディングに関する推論は、突然変異研究 (英語版) を通じて行うことができるが、タンパク質のフォールディングを研究するための実験技術は、通常、タンパク質の段階的アンフォールディングやフォールディングと、標準的な非結晶学的技術を用いたコンホメーション変化の観察に依存している。
X線結晶構造解析

X線結晶構造解析は、折りたたまれたタンパク質の三次元構造を解読するための、効率的で重要な方法の一つである[45]。X線結晶構造解析を行うためには、対象となるタンパク質が結晶格子内に配置されている必要がある。タンパク質を結晶格子内に配置するためには、結晶化に適した溶媒を用意し、溶液中で過飽和状態の純粋なタンパク質を得て、溶液中で結晶を析出させる必要がある[46]。タンパク質が結晶化されると、X線ビームは結晶格子を介して集中することができ、ビームを回折したり、様々な方向にビームを外側に向けて発射したりする。これらの出射ビームは、内包されたタンパク質の特定の三次元構成に相関している。X線は、タンパク質の結晶格子内の個々の原子を取り囲む電子雲と特異的に相互作用し、識別可能な回折パターンを生成する[13]。電子密度雲をX線の振幅を関連付けることによってのみ、このパターンを読み取ることができ、この方法を複雑にする位相や位相角の仮定を導くことになる[47]。フーリエ変換という数学的基礎によって確立された関係がなければ、「位相問題」は回折パターン予測を非常に困難にする[13]。多重同型置換のような新しい方法では、重金属イオンの存在を利用してX線をより予測可能な方法で回折させ、関与する変数の数を減らして位相の問題を解決している[45]。
蛍光分光法
蛍光分光法は、タンパク質の折りたたみ状態を調べるための高感度な手法である。フェニルアラニン(Phe)、チロシン(Tyr)、トリプトファン(Trp)の3つのアミノ酸は固有の蛍光特性を持つが、TyrとTrpのみが量子収率が高く、良好な蛍光シグナルが得られるため、実験的に使用されている。TrpとTyrは共に280 nmの波長で励起されるのに対し、Trpだけは295 nmの波長で励起される。それらの芳香族性のため、TrpとTyr残基は、タンパク質の疎水性コア、2つのタンパク質ドメイン間の界面、またはオリゴマータンパク質のサブユニット間の界面に、完全または部分的に埋もれていることがよくある。この無極性環境では、これらの残基は高い量子収率を持ち、それゆえ高い蛍光強度を示す。タンパク質の3次構造や4次構造が破壊されると、これらの側鎖は溶媒の親水性環境にさらされるようになり、量子収率が低下して低い蛍光強度になる。Trp残基については、その最大蛍光発光の波長も環境に依存する。
蛍光分光法は、変性剤の値の関数として、蛍光発光強度または最大発光波長の変動を測定することにより、タンパク質の平衡アンフォールディングを特徴付けるために使用される[48][49]。ここで変性剤は、化学分子 (尿素、塩酸グアニジニウム)、温度、pH、圧力などである。互いに異なるが離散的なタンパク質の状態、すなわち天然状態、中間状態、アンフォールド状態の間の平衡は、変性剤の値に依存するため、それらの平衡混合物の全体的な蛍光シグナルもこの値に依存する。このようにして、全体的なタンパク質シグナルを変性剤の値に関連付けるプロファイルが得られる[50][51]。平衡アンフォールディングのプロファイルは、アンフォールディングの中間体を検出し、識別することを可能にする。Hugues Bedouelleによって一般的な方程式が開発され、そのようなプロファイルから、ホモマーまたはヘテロマーのタンパク質のアンフォールディング平衡を特徴づける熱力学的パラメータを三量体まで、および潜在的には四量体まで得ることができた[48]。蛍光分光法は、ストップフローのような高速混合装置と組み合わせて、タンパク質のフォールディング動態を測定し[52]、シェブロンプロットを生成し、Phi値分析 (英語版) を導出することができる。
円偏光二色性
円偏光二色性は、タンパク質のフォールディングを研究するための最も一般的で基本的なツールの一つである。円二色性分光法は、円偏光の吸収を測定する。タンパク質では、αヘリックスやβシートなどの構造は不斉であるため、このような光を吸収する。この光の吸収は、タンパク質アンサンブルの折りたたみ度合いのマーカーとして機能する。この技術は、変性剤濃度または温度関数としてこの吸収の変化を測定することにより、タンパク質の平衡アンフォールディングを測定するために使用されてきた。変性溶解は、タンパク質のm値、または変性剤依存性と同様にアンフォールディングの自由エネルギーを測定する。温度溶解は、タンパク質の変性温度(Tm)を測定する[48]。蛍光分光法に関しては、円二色性分光法をストップフローなどの高速混合装置と組み合わせて、タンパク質のフォールディング動態を測定し、シェブロンプロットを生成することができる。
タンパク質の振動円二色性
最近開発されたタンパク質の振動円二色性 (vibrational circular dichroism; VCD) 技術は現在、フーリエ変換(FT)機器を用いており、非常に大きなタンパク質分子でも、溶液中のタンパク質構造を決定する強力な手段を提供する。このようなタンパク質のVCD研究は、タンパク質結晶のX線回折、重水(D2O)中のタンパク質溶液のFT-IRデータ、または第一原理量子計算と組み合わせて、円偏光二色性(CD)からは得られない明確な構造決定を提供することがよくある[要出典]。
タンパク質核磁気共鳴分光法
タンパク質のフォールディングは、NMR分光法を使用して日常的に研究されており、例えば、その天然状態でのタンパク質の主鎖アミドプロトンの水素-重水素交換 (英語版) を監視することで、タンパク質の残基固有の安定性と全体的な安定性の両方を提供する[53]。
二重偏光干渉法
二重偏光干渉法は、分子層の光学特性を測定する表面ベースの技術である。タンパク質のフォールディングを特徴づけるために使用される場合、タンパク質の単層の全体的なサイズとその密度をサブ・オングストーム分解能でリアルタイムに測定することによってコンホメーションを測定するが[54]、タンパク質フォールディングの速度論のリアルタイム測定は、~10 Hzよりも遅いプロセスに限られている。円偏光二色性と同様に、フォールディングのための刺激は変性剤または温度である可能性がある。
高時間分解能でのフォールディングの研究
タンパク質のフォールディングの研究は、高速で時間分解技術の開発によって近年、大きく進展した。実験者は、折りたたまれていないタンパク質のサンプルのフォールディングを迅速に誘発し、結果として生じるタンパク質動力学を観察する。使用されている高速化技術には、中性子散乱[55]、超高速溶液混合、光化学的手法、レーザー温度ジャンプ分光法などがある。これらの技術の開発に貢献した多くの科学者の中には、Jeremy Cook, Heinrich Roder, Harry Gray, Martin Gruebele, Brian Dyer, William Eaton, Sheena Radford, Chris Dobson, Alan Fersht, Bengt Nölting, Lars Konermannがいる。
タンパク質分解
タンパク質分解 (proteolysis) は、広範囲の溶液条件下 (例: 高速並列タンパク質分解) でアンフォールドされた画分を探索するために日常的に使用されている[56][57]。
単一分子力分光法
単離されたタンパク質やシャペロンを持つタンパク質のフォールディング機構を理解するために、光ピンセットやAFMなどの単一分子力技術 (Single-molecule force spectroscopy) が用いられてきた[58]。光ピンセットは、単一のタンパク質分子をC末端とN末端から引き伸ばし、それを展開して、その後のリフォールディングを研究するために使用されてきた[59]。この手法により、単一分子レベルでフォールディング率を測定できる。例えば、光ピンセットは最近、血液凝固に関与するタンパク質のフォールディングとアンフォールディングの研究に応用されている。ヴォン・ヴィレブランド因子 (von Willebrand factor (vWF)) は、血液凝固プロセスに不可欠な役割を持つタンパク質である。単一分子の光ピンセット測定を使用して、カルシウム結合vWFが血液中でせん断力センサーとして働くことを発見した。せん断力はvWFのA2ドメインのアンフォールディングにつながり、そのリフォールディング速度はカルシウムの存在下で劇的に向上する[60]。最近では、単純な src SH3 ドメインが、力を受けると複数のアンフォールディング経路にアクセスすることも明らかにされた[61]。
ビオチン標識
ビオチン標識 (Biotin painting) は、(未)フォールディング・タンパク質の状態特異的な細胞スナップショットを可能にする。 ビオチン標識は、予測される天然変成タンパク質への偏りを示している[62]。
タンパク質フォールディングの計算科学的研究
タンパク質フォールディングの計算科学的研究には、タンパク質の安定性、速度論、および構造予測に関連する3つの主要な側面が含まれる。以下の最近のレビューは、タンパク質フォールディングに利用可能な計算手法をまとめたものである[63]。
レヴィンタールのパラドックス
1969年、サイラス・レヴィンタールは、折りたたまれていないポリペプチド鎖の自由度が非常に大きいため、タンパク質分子は天文学的な数の起こりうるコンホメーションを持っていることに着目した。彼の論文の中で、3300または10143という推定がなされている[64]。レヴィンタールのパラドックスは、タンパク質が起こりうるすべてのコンホメーションを順次サンプリングして折りたたまれた場合、たとえコンホメーションが高速 (ナノ秒またはピコ秒スケール) でサンプリングされたとしても、天文学的な量の時間がかかるという観察に基づく思考実験である[65]。タンパク質はこれよりもはるかに速く折りたたまれるという観測に基づいて、レヴィンタールは、ランダムなコンホメーション探索は発生しないため、タンパク質は一連の準安定な中間状態を経て折りたたまれなければならないと提案した。
タンパク質フォールディングのエネルギー地形
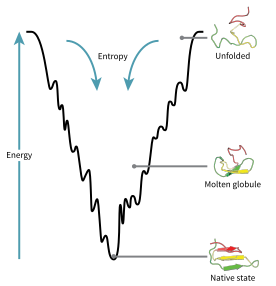
フォールディング中のタンパク質の配位空間は、エネルギー地形 (energy landscape) として可視化できる。Joseph BryngelsonとPeter Wolynesによると、タンパク質は最小フラストレーション原理に従っており、自然に進化したタンパク質はフォールディング時のエネルギー地形を最適化し[66]、自然はタンパク質の折りたたみ状態が十分に安定するようにアミノ酸配列を選択していることを意味している。さらに、折りたたまれた状態の獲得は、十分に高速なプロセスにならなければならない。自然がタンパク質のフラストレーションのレベルを減らしたとしても、タンパク質のエネルギー地形における局所的な最小値が存在することからもわかるように、ある程度のフラストレーションは今のところ残っている。
これらの進化的に選択された配列の結果として、タンパク質は、天然状態に向かうグローバルな「ファンネル(漏斗)状のエネルギー地形」(José Onuchicの造語[67]) を持っていると一般的に考えられている。この「フォールディング・ファンネル」地形により、タンパク質は、単一のメカニズムに限定されるのではなく、多数の経路や中間体のいずれかを介して天然状態にフォールディングできる。この理論は、モデルタンパク質の計算シミュレーション(格子タンパク質)と実験的研究の両方で支持されており[66]、タンパク質構造の予測とタンパク質構造設計のための方法を改善するために使用されてきた[66]。平準化自由エネルギー地形によるタンパク質フォールディングの説明も、熱力学第2法則と合致している[68]。物理的には、エネルギー地形を、地理的な地形のように、単に最大値、鞍点、最小値、ファンネルを持った可視化可能なポテンシャル曲面や全エネルギー曲面の観点から考えることは、あるいはいくらか誤解を招く可能性がある。妥当な記述は、実際には、多様体が様々なより複雑な位相形態をとる可能性のある高次元の位相空間である[69]。
折りたたまれていないポリペプチド鎖は、ファンネルの一番上に位置し、折りたたまれていないバリエーションの数が最も多く、エネルギー状態は高も高くなる。このようなエネルギー地形は、初期の可能性が多数あることを示しているが、可能なのは単一の天然状態のみである。しかし、それは可能な多くのフォールディング経路を明らかにしていない。同じ正確なタンパク質の異なる分子は、同じ天然構造に到達する限り、わずかに異なるフォールディング経路をたどり、異なる低エネルギー中間体を探すことができる場合がある[70]。異なる経路は、各経路の熱力学的な有利性に応じて、異なる利用頻度を持つ可能性がある。これは、ある経路が他の経路よりも熱力学的に有利であることが分かった場合、本来の構造を追求するために、より頻繁に使用される可能性が高いことを意味する[70]。タンパク質が折りたたみ始め、さまざまなコンホメーションをとると、常に以前よりも熱力学的に有利な構造を求め、エネルギーファンネルを通過し続けることになる。二次構造の形成は、タンパク質内の安定性の向上を強く示しており、ポリペプチド骨格によって想定される二次構造の一つの組み合わせだけが最低のエネルギーを持ち、ゆえにタンパク質は天然状態で存在することになる[70]。ポリペプチドが折りたたみを開始すると形成される最初の構造の中には、αヘリックスおよびβターンがあり、αヘリックスはわずか100ナノ秒で形成され、βターンは1マイクロ秒で形成される[29]。
エネルギー・ファンネル地形には、特定のタンパク質の遷移状態が見られる鞍点が存在する[29]。エネルギー・ファンネル図の遷移状態とは、タンパク質が最終的に本来の構造をとることを想定した場合、そのタンパク質のすべての分子がとらなければならないコンホメーションである。どのタンパク質も、最初に遷移状態を通過しなければ、本来の構造をとることはできない[29]。遷移状態は、単なる別の中間段階ではなく、天然状態の変化形または未熟な形と呼ぶことができる[71]。遷移状態のフォールディングは律速であることが示されており、それが本来のフォールディングよりも高いエネルギー状態で存在しているとしても、本来の構造と極めて類似する。遷移状態の中には、タンパク質が折りたたむことができる核(足場)となる構造が存在しており、核の上に構造が段階的に完成してゆく「凝縮核形成」と呼ばれるプロセスによって形成される[71]。
タンパク質フォールディングのモデリング

計算によるタンパク質構造予測のためのデ・ノボ (de novo) または第一原理 (ap initio) 的手法は、どちらもタンパク質フォールディングの実験的研究に関連しているが、厳密には区別されるものである。分子動力学法 (MD) は、タンパク質のフォールディングと動力学をイン・シリコ (in silico) で研究するための重要なツールである[72]。最初の平衡フォールディング・シミュレーションは、暗黙の溶媒モデルとアンブレラ・サンプリング法を用いて行われた[73]。計算コストが高いため、明示的な水を用いた第一原理計算によるフォールディング・シミュレーションは、ペプチドや非常に小さなタンパク質に限定される[74][75]。より大きなタンパク質のMDシミュレーションは、実験的な構造の動力学、または、その高温アンフォールディングに限定される。小さなサイズのタンパク質 (約50残基以上) のフォールディングのような長時間のフォールディングプロセス (約1ミリ秒を超える) は、粗視化モデルを用いて解析することができる[76][77][78]。
スタンフォード大学のVijay Pande氏のグループが作成した100ペタFLOP級の分散コンピューティングプロジェクト Folding@home は、ボランティアのパーソナルコンピュータのCPUとGPUのアイドル処理時間を利用して、タンパク質のフォールディングをシミュレーションする。このプロジェクトは、タンパク質のフォールディングのミスフォールディング(誤った折りたたみ)を理解し、疾患研究のための創薬ドラッグデザインを加速することを目的としている。
D. E. Shaw Research社 (英語版) のカスタムASICと相互接続を中心に設計・構築された超並列スーパーコンピュータAnton(アントン)で、長時間の連続軌道シミュレーションが実行された。Antonを使用して実行されたシミュレーションの公開された最長の結果は、355KでのNTL9の2.936ミリ秒のシミュレーションである[79]。
注釈と出典
- ^ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walters, Peter (2002). “The Shape and Structure of Proteins”. Molecular Biology of the Cell; Fourth Edition. New York and London: Garland Science. ISBN 978-0-8153-3218-3
- ^ “The formation and stabilization of protein structure”. The Biochemical Journal 128 (4): 737–49. (July 1972). doi:10.1042/bj1280737. PMC 1173893. PMID 4565129.
- ^ Berg, Jeremy M.; Tymoczko, John L.; Stryer, Lubert (2002). “3. Protein Structure and Function”. Biochemistry. San Francisco: W. H. Freeman. ISBN 978-0-7167-4684-3
- ^ a b “Folding proteins in fatal ways”. Nature 426 (6968): 900–4. (December 2003). Bibcode: 2003Natur.426..900S. doi:10.1038/nature02264. PMID 14685251.
- ^ Alberts, Bruce; Bray, Dennis; Hopkin, Karen; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter (2010). “Protein Structure and Function”. Essential cell biology (Third ed.). New York, NY: Garland Science. pp. 120–70. ISBN 978-0-8153-4454-4
- ^ “Intermediates in the folding reactions of small proteins”. Annual Review of Biochemistry 59: 631–60. (1990). doi:10.1146/annurev.bi.59.070190.003215. PMID 2197986.
- ^ “How do small single-domain proteins fold?”. Folding & Design 3 (4): R81-91. (1998). doi:10.1016/S1359-0278(98)00033-9. PMID 9710577.
- ^ “The protein folding 'speed limit'”. Current Opinion in Structural Biology 14 (1): 76–88. (February 2004). doi:10.1016/j.sbi.2004.01.013. PMID 15102453.
- ^ “Principles that govern the folding of protein chains”. Science 181 (4096): 223–30. (July 1973). Bibcode: 1973Sci...181..223A. doi:10.1126/science.181.4096.223. PMID 4124164.
- ^ a b c d e f g h Voet, Donald; Voet, Judith G.; Pratt, Charlotte W. (2016). Principles of Biochemistry (Fifth ed.). Wiley. ISBN 978-1-118-91840-1
- ^ “The design and characterization of two proteins with 88% sequence identity but different structure and function”. Proceedings of the National Academy of Sciences of the United States of America 104 (29): 11963–8. (July 2007). Bibcode: 2007PNAS..10411963A. doi:10.1073/pnas.0700922104. PMC 1906725. PMID 17609385.
- ^ “A backbone-based theory of protein folding”. Proceedings of the National Academy of Sciences of the United States of America 103 (45): 16623–33. (November 2006). Bibcode: 2006PNAS..10316623R. doi:10.1073/pnas.0606843103. PMC 1636505. PMID 17075053.
- ^ a b c Fersht, Alan (1999). Structure and Mechanism in Protein Science: A Guide to Enzyme Catalysis and Protein Folding. Macmillan. ISBN 978-0-7167-3268-6
- ^ “Protein Structure”. Scitable. Nature Education. 2016年11月26日閲覧。
- ^ Pratt, Charlotte; Cornely, Kathleen (2004). “Thermodynamics”. Essential Biochemistry. Wiley. ISBN 978-0-471-39387-0 2016年11月26日閲覧。
- ^ Fedorov AN (1997). “Cotranslational Protein Folding”. J. Biol. Chem. 272 (52): 32715-32718. doi:10.1074/jbc.272.52.32715.
- ^ Zhang, Gong; Ignatova, Zoya (2011-02-01). “Folding at the birth of the nascent chain: coordinating translation with co-translational folding”. Current Opinion in Structural Biology 21 (1): 25–31. doi:10.1016/j.sbi.2010.10.008. ISSN 0959-440X. PMID 21111607.
- ^ “Macromolecular crowding perturbs protein refolding kinetics: implications for folding inside the cell”. The EMBO Journal 19 (15): 3870–5. (August 2000). doi:10.1093/emboj/19.15.3870. PMC 306593. PMID 10921869.
- ^ Al-Karadaghi, Salam. “Torsion Angles and the Ramachnadran Plot in Protein Structures”. www.proteinstructures.com. 2016年11月26日閲覧。
- ^ “Forces contributing to the conformational stability of proteins”. FASEB Journal 10 (1): 75–83. (January 1996). doi:10.1096/fasebj.10.1.8566551. PMID 8566551.
- ^ “Protein-spanning water networks and implications for prediction of protein-protein interactions mediated through hydrophobic effects”. Proteins 82 (12): 3312–26. (December 2014). doi:10.1002/prot.24683. PMID 25204743.
- ^ “The hydrophobic effect and the organization of living matter”. Science 200 (4345): 1012–8. (June 1978). Bibcode: 1978Sci...200.1012T. doi:10.1126/science.653353. PMID 653353.
- ^ “Context-dependent contributions of backbone hydrogen bonding to beta-sheet folding energetics”. Nature 430 (6995): 101–5. (July 2004). Bibcode: 2004Natur.430..101D. doi:10.1038/nature02611. PMID 15229605.
- ^ Irbäck, Anders; Sandelin, Erik (November 2000). “On Hydrophobicity Correlations in Protein Chains”. Biophysical Journal 79 (5): 2252–2258. arXiv:cond-mat/0010390. Bibcode: 2000BpJ....79.2252I. doi:10.1016/S0006-3495(00)76472-1. PMC 1301114. PMID 11053106.
- ^ Irbäck, A.; Peterson, C.; Potthast, F. (3 September 1996). “Evidence for nonrandom hydrophobicity structures in protein chains.”. Proceedings of the National Academy of Sciences 93 (18): 9533–9538. arXiv:chem-ph/9512004. Bibcode: 1996PNAS...93.9533I. doi:10.1073/pnas.93.18.9533. PMC 38463. PMID 8790365.
- ^ Wilson, Benjamin A.; Foy, Scott G.; Neme, Rafik; Masel, Joanna (24 April 2017). “Young genes are highly disordered as predicted by the preadaptation hypothesis of de novo gene birth”. Nature Ecology & Evolution 1 (6): 0146–146. doi:10.1038/s41559-017-0146. PMC 5476217. PMID 28642936.
- ^ Willis, Sara; Masel, Joanna (September 2018). “Gene Birth Contributes to Structural Disorder Encoded by Overlapping Genes”. Genetics 210 (1): 303–313. doi:10.1534/genetics.118.301249. PMC 6116962. PMID 30026186.
- ^ Foy, Scott G.; Wilson, Benjamin A.; Bertram, Jason; Cordes, Matthew H. J.; Masel, Joanna (April 2019). “A Shift in Aggregation Avoidance Strategy Marks a Long-Term Direction to Protein Evolution”. Genetics 211 (4): 1345–1355. doi:10.1534/genetics.118.301719. PMC 6456324. PMID 30692195.
- ^ a b c d e f “Protein folding and misfolding”. Nature 426 (6968): 884–90. (December 2003). Bibcode: 2003Natur.426..884D. doi:10.1038/nature02261. PMID 14685248.
- ^ a b c “Molecular chaperones in cellular protein folding”. Nature 381 (6583): 571–9. (June 1996). Bibcode: 1996Natur.381..571H. doi:10.1038/381571a0. PMID 8637592.
- ^ a b “Molecular chaperones in protein folding and proteostasis”. Nature 475 (7356): 324–32. (July 2011). doi:10.1038/nature10317. PMID 21776078.
- ^ “Molecular chaperone functions in protein folding and proteostasis”. Annual Review of Biochemistry 82: 323–55. (2013). doi:10.1146/annurev-biochem-060208-092442. PMID 23746257.
- ^ “The denatured state (the other half of the folding equation) and its role in protein stability”. FASEB Journal 10 (1): 27–34. (January 1996). doi:10.1096/fasebj.10.1.8566543. PMID 8566543.
- ^ “Molecular chaperones in protein quality control”. Journal of Biochemistry and Molecular Biology 38 (3): 259–65. (2005). doi:10.5483/BMBRep.2005.38.3.259. PMID 15943899.
- ^ “Electric field-driven disruption of a native beta-sheet protein conformation and generation of a helix-structure”. Biophysical Journal 99 (2): 595–9. (July 2010). Bibcode: 2010BpJ....99..595O. doi:10.1016/j.bpj.2010.04.040. PMC 2905109. PMID 20643079.
- ^ “Effects of macromolecular crowding on protein folding and aggregation”. The EMBO Journal 18 (24): 6927–33. (December 1999). doi:10.1093/emboj/18.24.6927. PMC 1171756. PMID 10601015.
- ^ “Molecular chaperones: assisting assembly in addition to folding”. Trends in Biochemical Sciences 31 (7): 395–401. (July 2006). doi:10.1016/j.tibs.2006.05.001. PMID 16716593.
- ^ “Cell proliferation at 122 degrees C and isotopically heavy CH4 production by a hyperthermophilic methanogen under high-pressure cultivation”. Proceedings of the National Academy of Sciences of the United States of America 105 (31): 10949–54. (August 2008). Bibcode: 2008PNAS..10510949T. doi:10.1073/pnas.0712334105. PMC 2490668. PMID 18664583.
- ^ a b Marusich EI, Kurochkina LP, Mesyanzhinov VV. Chaperones in bacteriophage T4 assembly. Biochemistry (Mosc). 1998;63(4):399-406
- ^ a b c d “Protein-misfolding diseases and chaperone-based therapeutic approaches”. The FEBS Journal 273 (7): 1331–49. (April 2006). doi:10.1111/j.1742-4658.2006.05181.x. PMID 16689923.
- ^ a b “Amyloids, prions and the inherent infectious nature of misfolded protein aggregates”. Trends in Biochemical Sciences 31 (3): 150–5. (March 2006). doi:10.1016/j.tibs.2006.01.002. PMID 16473510.
- ^ “Prevention of transthyretin amyloid disease by changing protein misfolding energetics”. Science 299 (5607): 713–6. (January 2003). Bibcode: 2003Sci...299..713H. doi:10.1126/science.1079589. PMID 12560553.
- ^ “Protein misfolding, functional amyloid, and human disease”. Annual Review of Biochemistry 75: 333–66. (2006). doi:10.1146/annurev.biochem.75.101304.123901. PMID 16756495.
- ^ “Native state kinetic stabilization as a strategy to ameliorate protein misfolding diseases: a focus on the transthyretin amyloidoses”. Accounts of Chemical Research 38 (12): 911–21. (December 2005). doi:10.1021/ar020073i. PMID 16359163.
- ^ a b Cowtan, Kevin (2001). "Phase Problem in X-ray Crystallography, and Its Solution" (PDF). Encyclopedia of Life Sciences. Macmillan Publishers Ltd, Nature Publishing Group. 2016年11月3日閲覧。
{{cite encyclopedia}}: 不明な引数|name-list-format=は無視されます。(もしかして:|name-list-style=) (説明) - ^ Drenth, Jan (2007-04-05). Principles of Protein X-Ray Crystallography. Springer Science & Business Media. ISBN 978-0-387-33746-3
- ^ Taylor, Garry (2003). “The phase problem”. Acta Crystallographica Section D 59 (11): 1881–90. doi:10.1107/S0907444903017815. PMID 14573942.
- ^ a b c “Principles and equations for measuring and interpreting protein stability: From monomer to tetramer”. Biochimie 121: 29–37. (February 2016). doi:10.1016/j.biochi.2015.11.013. PMID 26607240.
- ^ “Quantitative measurement of protein stability from unfolding equilibria monitored with the fluorescence maximum wavelength”. Protein Engineering, Design & Selection 18 (9): 445–56. (September 2005). doi:10.1093/protein/gzi046. PMID 16087653.
- ^ “Dimeric tyrosyl-tRNA synthetase from Bacillus stearothermophilus unfolds through a monomeric intermediate. A quantitative analysis under equilibrium conditions”. The Journal of Biological Chemistry 273 (29): 18052–9. (July 1998). doi:10.1074/jbc.273.29.18052. PMID 9660761.
- ^ “Multiple folding states and disorder of ribosomal protein SA, a membrane receptor for laminin, anticarcinogens, and pathogens”. Biochemistry 51 (24): 4807–21. (June 2012). doi:10.1021/bi300335r. PMID 22640394.
- ^ “Probing protein folding and conformational transitions with fluorescence”. Chemical Reviews 106 (5): 1769–84. (May 2006). doi:10.1021/cr0404390. PMID 16683754.
- ^ Beatrice M.P. Huyghues-Despointes, C. Nick Pace, S. Walter Englander, and J. Martin Scholtz. "Measuring the Conformational Stability of a Protein by Hydrogen Exchange." Methods in Molecular Biology. Kenneth P. Murphy Ed. Humana Press, Totowa, New Jersey, 2001. pp. 69–92
- ^ Cross, Graham H.; Freeman, Neville J.; Swann, Marcus J. (2008). “Dual Polarization Interferometry: A Real-Time Optical Technique for Measuring (Bio)molecular Orientation, Structure and Function at the Solid/Liquid Interface”. Handbook of Biosensors and Biochips. doi:10.1002/9780470061565.hbb055. ISBN 978-0-470-01905-4
- ^ “Dynamic regimes and correlated structural dynamics in native and denatured alpha-lactalbumin”. Journal of Molecular Biology 312 (4): 865–73. (September 2001). doi:10.1006/jmbi.2001.5006. PMID 11575938.
- ^ “Determining biophysical protein stability in lysates by a fast proteolysis assay, FASTpp”. PLOS ONE 7 (10): e46147. (2012). Bibcode: 2012PLoSO...746147M. doi:10.1371/journal.pone.0046147. PMC 3463568. PMID 23056252.
- ^ “Pulse proteolysis: a simple method for quantitative determination of protein stability and ligand binding”. Nature Methods 2 (3): 207–12. (March 2005). doi:10.1038/nmeth740. PMID 15782190.
- ^ “Chaperone action at the single-molecule level”. Chemical Reviews 114 (1): 660–76. (January 2014). doi:10.1021/cr400326k. PMID 24001118.
- ^ “Protein folding and unfolding under force”. Biopolymers 99 (11): 860–9. (November 2013). doi:10.1002/bip.22321. PMC 4065244. PMID 23784721.
- ^ “Calcium modulates force sensing by the von Willebrand factor A2 domain”. Nature Communications 2: 385. (July 2011). Bibcode: 2011NatCo...2..385J. doi:10.1038/ncomms1385. PMC 3144584. PMID 21750539.
- ^ “Direct observation of a force-induced switch in the anisotropic mechanical unfolding pathway of a protein”. Proceedings of the National Academy of Sciences of the United States of America 109 (44): 17820–5. (October 2012). Bibcode: 2012PNAS..10917820J. doi:10.1073/pnas.1201800109. PMC 3497811. PMID 22949695.
- ^ “Biotinylation by proximity labelling favours unfolded proteins”. bioRxiv. (2018). doi:10.1101/274761.
- ^ “Computational and theoretical methods for protein folding”. Biochemistry 52 (48): 8601–24. (December 2013). doi:10.1021/bi4001529. PMID 24187909.
- ^ “Structural Biochemistry/Proteins/Protein Folding - Wikibooks, open books for an open world”. en.wikibooks.org. 2016年11月5日閲覧。
- ^ Levinthal, Cyrus (1968). “Are there pathways for protein folding?”. Journal de Chimie Physique et de Physico-Chimie Biologique 65: 44–45. Bibcode: 1968JCP....65...44L. doi:10.1051/jcp/1968650044. オリジナルの2009-09-02時点におけるアーカイブ。.
- ^ a b c “Funnels, pathways, and the energy landscape of protein folding: a synthesis”. Proteins 21 (3): 167–95. (March 1995). arXiv:chem-ph/9411008. doi:10.1002/prot.340210302. PMID 7784423.
- ^ Leopold PE; Montal M; Onuchic JN (September 1992). “Protein folding funnels: a kinetic approach to the sequence-structure relationship”. Proceedings of the National Academy of Sciences of the United States of America 89 (18): 8721–5. Bibcode: 1992PNAS...89.8721L. doi:10.1073/pnas.89.18.8721. PMC 49992. PMID 1528885.
- ^ Sharma, Vivek; Kaila, Ville R.I.; Annila, Arto (2009). “Protein folding as an evolutionary process”. Physica A: Statistical Mechanics and Its Applications 388 (6): 851–62. Bibcode: 2009PhyA..388..851S. doi:10.1016/j.physa.2008.12.004.
- ^ Robson, Barry; Vaithilingam, Andy (2008). “Protein Folding Revisited”. Molecular Biology of Protein Folding, Part B. Progress in Molecular Biology and Translational Science. 84. pp. 161–202. doi:10.1016/S0079-6603(08)00405-4. ISBN 978-0-12-374595-8. PMID 19121702
- ^ a b c “The protein-folding problem, 50 years on”. Science 338 (6110): 1042–6. (November 2012). Bibcode: 2012Sci...338.1042D. doi:10.1126/science.1219021. PMID 23180855.
- ^ a b “Transition-state structure as a unifying basis in protein-folding mechanisms: contact order, chain topology, stability, and the extended nucleus mechanism”. Proceedings of the National Academy of Sciences of the United States of America 97 (4): 1525–9. (February 2000). Bibcode: 2000PNAS...97.1525F. doi:10.1073/pnas.97.4.1525. PMC 26468. PMID 10677494.
- ^ “Using simulations to provide the framework for experimental protein folding studies”. Archives of Biochemistry and Biophysics 531 (1–2): 128–35. (March 2013). doi:10.1016/j.abb.2012.12.015. PMC 4084838. PMID 23266569.
- ^ “Solution conformations and thermodynamics of structured peptides: molecular dynamics simulation with an implicit solvation model”. Journal of Molecular Biology 284 (3): 835–48. (December 1998). doi:10.1006/jmbi.1998.2172. PMID 9826519.
- ^ Jones, David. “Fragment-based Protein Folding Simulations”. University College London. 2020年10月12日閲覧。
- ^ “Protein folding” (by Molecular Dynamics). 2020年10月12日閲覧。
- ^ “Coarse-Grained Protein Models and Their Applications”. Chemical Reviews 116 (14): 7898–936. (July 2016). doi:10.1021/acs.chemrev.6b00163. PMID 27333362.
- ^ “Characterization of protein-folding pathways by reduced-space modeling”. Proceedings of the National Academy of Sciences of the United States of America 104 (30): 12330–5. (July 2007). Bibcode: 2007PNAS..10412330K. doi:10.1073/pnas.0702265104. PMC 1941469. PMID 17636132.
- ^ “De novo prediction of protein folding pathways and structure using the principle of sequential stabilization”. Proceedings of the National Academy of Sciences of the United States of America 109 (43): 17442–7. (October 2012). Bibcode: 2012PNAS..10917442A. doi:10.1073/pnas.1209000109. PMC 3491489. PMID 23045636.
- ^ “How fast-folding proteins fold”. Science 334 (6055): 517–20. (October 2011). Bibcode: 2011Sci...334..517L. doi:10.1126/science.1208351. PMID 22034434.
参考文献
- 藤博幸 編「タンパク質の立体構造入門」 講談社 (2010)
- Voet & Voet "Biochemistry" 3rd edition, Wiley (2004)
関連項目
- アントン (スーパーコンピュータ)
- シャペロン
- シェブロン・プロット
- 変性中間点
- ダウンヒル・フォールディング
- 高速並列タンパク質分解
- フォールディング (化学)
- Folding@Home
- Foldit - コンピュータ・ゲーム
- 力場 (化学)
- Pt-barrel
- タンパク質動力学
- タンパク質ミスフォールディング周期的増幅 (PMCA法)
- タンパク質構造予測ソフトウェア
- プロテオパチー
- Rosetta@home
- 分子力学モデリング用ソフトの比較
- 統計的ポテンシャル
- 時間分解質量分析法
