転写 (生物学)
概要
[編集]転写は膨大な種類のタンパク質とRNAを必要な時期に必要な量を正確に合成するための重要な代謝過程である。この重要で難解な作業を成功させるために、生物は遺伝子を活用する。遺伝子は安定なDNAの塩基配列で記録され、生物はそれと同じ配列のRNAを合成することができる。これが転写であり、例えばtRNAなどが転写される。また遺伝子(とそれを写し取ったRNA)の中には特定のタンパク質に対応するものもある。遺伝情報に基づいてタンパク質を作ることを、その遺伝子を発現 expression するというが、その前段階として特に転写は重要である。
タンパク質を合成するために必要なRNAは伝令RNA(メッセンジャーRNA messenger RNA:mRNA) であるが、その合成過程は種によって大いに異なる。原核生物ではmRNAを転写することができるのに対し、真核生物は転写で作ったRNA(一次転写産物 primary transcript )にPre-mRNA スプライシングなどのさらなる段階を経なければmRNAを得ることができない。真核生物におけるmRNAを特に成熟mRNA、成熟する前のRNAをmRNA前駆体と呼ぶ。また、転写から直接合成するRNAを転写産物 transcription product と呼ぶ。
一般に転写には開始、伸長、終結の過程があり[1]、また開始にいたるまでに様々な転写調節因子が転写の活性化や抑制に関わっている。近年はクロマチン構造を通しての転写制御機構が注目されている。基本的な機構はあらゆる生物で共通しているが、各ドメイン(生物を分類する最上位階級)ごとの違いはやや大きい。細菌は細胞質中で転写を行い、転写機構も単純である。一方、真核生物は、転写を細胞核内でのみ行い、多数の酵素が関る複雑な機構を使っている。最後の古細菌は、細胞質中で転写を行う点は細菌と同じだが、転写機構そのものは真核生物に類似している。
転写産物はRNA合成酵素RNAポリメラーゼ RNA polymerase (DNA依存性RNAポリメラーゼ DNA-dependent RNA polymerase )により合成される。この酵素はssDNAと結合すると、それと相補的なRNA鎖を合成する[注釈 1]。したがって、RNAポリメラーゼが結合する方を鋳型鎖template strand または転写鎖;(-)鎖、もう片方は非鋳型鎖 nontemplate strand あるいは非転写鎖;(+)鎖である。また、転写産物が(-)鎖と相補的であるからその塩基配列は(+)鎖に等しい。よって、(+)鎖こそが遺伝情報を保持しているといえ、コード鎖 coding strand またはセンス鎖 sense strand (センス=意味)ともいう。それにちなんで(-)鎖をアンチコード鎖やアンチセンス鎖 antisense strand という。ただし、転写産物と(+)鎖は完全に同じ存在というわけではなく、(+)鎖におけるチミンは転写産物でウラシルに置き換わっているし、DNAとRNAという違いも忘れてはならない。
DNA上のどこからどこまでを転写の範囲とするかは(-)鎖の塩基配列により決まっており、この範囲を転写単位 transcription unit としてオペロン operon という。細菌のDNAには数百のオペロンがあり、真核細胞では数千のオペロンを持つ。
開始段階
[編集]
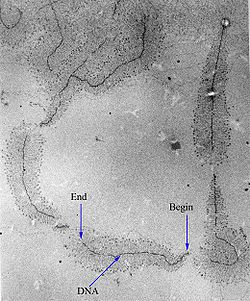
転写における開始段階 initiation ではまずRNAポリメラーゼをはじめ転写に関わる酵素がDNA上の転写開始部位に結合する。この部位はオペロンの5'側末端であり、プロモーター promoter という。結合した各酵素は複合体(ホロ酵素)[注釈 2]を成すが、この構造は順序だって変遷するため開始段階はさらに3つに分けられる。初めは組み込んだDNAが二重らせんのままである閉鎖型複合体 closed complex:PRc であるが、転写はssDNAでないと実行できない。第2段階として、プロモーターをほどいた開放型複合体 open complex:PR0 と、ssDNA領域の転写バブル ribonucleoside triphosphate (先端を解離点 unwinding point 、後端を巻き直し点 rewinding point という)を形成する[注釈 3]。転写産物の始まりとなる約10nt(ヌクレオチド)の合成も最後の段階として行う。RNAポリメラーゼが初活躍するこの時期は初期転写複合体 initial transcribing complex と呼び、伸長段階に向けてプロモーターから脱出する[1]。
伸長
[編集]
伸長 Elongationの段階で本格的に転写産物の合成が行われる。RNAポリメラーゼは結合しているDNAの塩基を正確に識別し、4つのリボヌクレオシド三リン酸 ribonucleoside triphosphate (ATP、GTP、CTP、UTP)の中からそれと相補的なものを選ぶ。そして、直前の開始段階で合成された短いRNA鎖の5'末端に付加する。これが済むとRNAポリメラーゼと転写バブルは次の塩基へと移動し、再びRNA鎖にリボヌクレオシド三リン酸を付加する。この反応には進む方向が決まっており、RNAの5'末端から3'末端へ、逆平行の(-)鎖にとっては3'末端から5'末端へ向かう。通り過ぎたDNAは再会合する。これを繰り返してRNAは伸長し、転写産物が作られていく。
終結
[編集]終結 termination は、RNAポリメラーゼが遺伝子末端にあるターミネーター terminator まで来ると起こる。ターミネーターはRNAポリメラーゼと協力しながら転写産物とDNAとの結合を切り、RNAポリメラーゼからもDNAからも解離させる。
細菌の転写
[編集]細菌における転写機構は大腸菌において特に良く調べられている。基本的な機構は他の生物でも同じであるが、細部には違いが見られる。特に真核生物では機構がかなり複雑である。開始段階における特に大きな違いは、細菌のRNAポリメラーゼは単独で転写を始めることができるが、真核生物はプロモーターに結合するのにも様々なタンパク質を要する。
細菌の転写開始
[編集]細菌の転写開始は次の4つの段階を経る[2]。①RNAポリメラーゼがプロモーターを認識し、閉鎖型複合体を形成する。この複合体のDNAとの結合は可逆的であり、平衡定数(KB)は106〜109 M-1である[3]。②閉鎖型プロモーター複合体を開放型複合体に変換する。不可逆的であり、反応速度定数(k2)は10-3〜10-1である[3]。③さらに初期転写複合体 initial transcribing complex となり[1]、プロモーターに留まっている間に10nt(ヌクレオチド)を合成。④転写産物の先駆けが十分に長くなると、伸長段階に移行するためホロ酵素の立体構造が変化する。この過程をプロモータークリアランス promoter clearance と呼ぶ。④から伸長開始までをプロモータークリアランス時間 promoter clearance time といい、短くても1〜2秒はかかる[4]。
①から②への変化は異性化 isomerization または強い結合 tight binding と呼び、融解は-11から3の短い領域で起きる。σ70と複合体を形成したRNAポリメラーゼの場合、以降にも調節機構があるとはいえ、実質的に不可逆なため済めばほぼ確実に転写が始まる。異性化にはホロ酵素の著しい変化が2つある。第一にホロ酵素のβとβ'サブユニットは下流DNAをしっかり固定する。第二はσの領域1.1が50Å移動して酵素の外側に出ることである。DNAと結合してないとき、領域1.1はホロ酵素の活性中心溝にあり、開放型複合体にある鋳型鎖の通り道を塞ぐ[注釈 4]。DNAと同様に負に荷電しているため、DNAと結合するため強い正荷電の活性中心溝で擬態した分子 molecular mimic として振る舞うためである。移動による開放でDNAは入れるようになる。
③のRNA合成は伸長段階でのそれと比べ、次に紹介する2つの理由により複雑な過程を経る。ほとんどの転写はAから始まる[5]が、合成された直後にこれを支えるのはDNAとの2本の弱い水素結合だけである。このためホロ酵素は1番目と2番目のリボヌクレオチドを、次に来るNTPが化学攻撃できる状態で固定しなければならない[5]。常に同じリボヌクレオチドから始まるのはホロ酵素が特異的に結合するためであろう。
第二の理由は転写開始の失敗 abortive initiation である。開始段階で10nt以上のRNAが合成されるが、しばしばホロ酵素はこれに満たないRNAを放出して、最初から合成をやり直す。放棄された転写産物をアボーティブ転写産物 abortive transcripts といい、この実りのない(意義はよくわかっていない)過程を繰り返さなければ次の伸長段階へと移れない。この間にホロ酵素がどのようにDNA上を移動しているかははっきりしておらず、3つのモデルが提唱されている[5]。
- 行きつ戻りつモデル
- RNAポリメラーゼは転写のために移動し、放出した後で元の開始位置に戻るとする説。
- 尺取虫モデル
- ホロ酵素本体は動かず、転写を実行する活性部位が移動するという説。RNAポリメラーゼ内に柔軟な部分があると仮定し、これが下流へ向かって伸びることで活性部位を持つ前部が連続して転写を実行する。中断時に縮み、活性部位を開始位置に戻す。
- しわ寄せモデル
- 本体も活性部位も動かず、DNAが活性部位へ連続して移動するという説。RNAポリメラーゼは、コードを巻き戻す掃除機のようにDNAを引き込む。内部でDNAはたわんだssDNAとなって溜まる。
現在ではしわ寄せモデルが真実に近いと予想されている。根拠の一つは、転写初期のRNAポリメラーゼはDNA上を静止し、下流DNAをほどいて内部へ引き込むという単分子解析の結果である[5]。
10nt以上のRNA合成に成功すると、RNAポリメラーゼはプロモーターを脱出 escape して伸長段階へ突入する(④)。合成されたRNAはホロ酵素内部で塩基対を形成しているが、これが収まりきらなくなってRNA出口通路へ抜く[6]。また、ホロ酵素とプロモーター間の結合は全て断つ。ホロ酵素の構成要素の一つであるσサブユニットも本体との結合が弱くなる[注釈 5]。脱出に当たり、しわ寄せモデルでほどかれたDNAが巻き戻り、同時に転写バブルが22〜24ntから12〜14ntに縮む[6]。この過程がRNAポリメラーゼ-プロモーターとコア酵素-シグマサブユニット間の結合の切断に要する自由エネルギーを供給する[6]。
細菌の転写伸長
[編集]伸長段階でDNAは開放型複合体の時と同様にホロ酵素内を通る。巨大なβとβ’サブユニットの間[注釈 2]に下流域が入り込み、入り口で分離して別々の道を往く[注釈 4]。それぞれ専用の通路に出た後、ホロ酵素の背で二重らせんを再構成する。基質のリボヌクレオシド三リン酸も専用の入り口から入る。鋳型鎖と塩基対形成しているのは伸長中の3’末端の8か9ntだけで、ほかの剥がれた部分はRNA出口通路から外へ出る[7]。

転写バブルの大きさは伸長段階を通して一定で、ホロ酵素は1bp(塩基対)ほどくと同時に後方で1bp再会合させる[7]。DNAのような二重らせんの高分子を酵素がほどくとき、捻じれてしまうことが大きな問題となる。これを解消しつつ転写バブルを移動させる方法については2通り考えられている。一つは、鋳型鎖の下流が二重らせんを緩めるよう回転し、転写された後に巻き戻るというもの。しかし、回転によってDNAの立体構造に大きなひずみを生むことは避けられない。このひずみは、DNAスーパーコイル(DNA supercoil)あるいはDNA超らせん(DNA superhelix)と呼ばれる。DNA複製にも同じ問題(DNA複製#DNAのよじれの解消を参照)は生じる。これらのひずみを解消するのが、トポイソメラーゼ(topoisomerase)と呼ばれる酵素群である。ほかに、RNAポリメラーゼがdsDNAの捩じれに沿って回転することで巻き戻すという説もある。これならばよじれの問題は起きないが、回転には大きなエネルギーが必要と考えられる。また、転写産物はDNAに巻きついてDNAの再会合を阻むことになるが、これをほどく酵素はいまだ発見されていない[8]。
RNAポリメラーゼは転写中たびたび1〜6秒一時停止する[8]。時には後戻りすることもあり、転写の平均的な速度は遅い。理由は少なくとも2つある。1つ目はもっと遅い翻訳に合わせるためであり、アテニュエーションや翻訳が失敗して転写を中断しなければならない場合に必要である[8]。2つ目は、一時停止が次の項で解説する#細菌の終結における第一段階であるためである。
RNAポリメラーゼは転写産物の塩基配列に誤りがないかの校正を2種類の方法で行う[7]。1つ目は、間違って付加したリボヌクレオシド三リン酸を活性部位の逆反応で除去する加ピロリン酸分解校正 pyrophosphorolytic editing である。リボヌクレオシド三リン酸との反応は3'末端から二リン酸を奪うので、付け直しも行う。正しい塩基対より活性部位に長く留まるため、間違った塩基対がより逆反応を受ける[7]。2つ目は加水分解校正 hydrolytic editing で、1個あるいはそれ以上のヌクレオチドの距離を引き返し、間違いを含む塩基配列を切り離す。
加水分解校正とともに伸長自体を促進するGre因子がある。この因子は伸長反応を効率よくし、転写の難しい所で停滞させないようにする。伸長段階でホロ酵素に加わり、伸長と終結を助けるNusタンパクというタンパク質群も発見されている。
RNAポリメラーゼが途中で転写をやめる場合がある。原因の多くはDNAの損傷であるが、これに出会うとそこから先の転写を続行できないうえに、停止すればほかのポリメラーゼも妨害してしまう。これに対処するため、TRCF transcription-repair coupling factor は停止したRNAポリメラーゼを発見するとヌクレオチド除去修復タンパク(エンドヌクレアーゼUvrA、B、C)を招集し、RNAポリメラーゼをDNAから離す[9][注釈 6]。RNAポリメラーゼは修復タンパクを損傷に近づけるようにすることもある[10]。これを転写と共役した修復 transcription-coupled repair という。
細菌の転写終結
[編集]RNAポリメラーゼが遺伝子配列の転写を終えても反応は終わらない。転写を確実に終わらせるために、遺伝子配列の下流に存在する逆方向反復配列 inverted repeats は転写された時、RNAポリメラーゼに干渉する。この遺伝子配列を写し取ったRNA領域は補塩基同士を結合させてステムループ構造 stem-loop (ヘアピン構造 hairpin loop )を作るが、これが転写終結のカギである。大腸菌の終結の方法は次の2つ。
- ρ非依存性終結 rho-independent termination
- mRNAの3'末端側に連続したDNAとのA-U塩基対が作成され、その結合の弱さから自然にmRNAがDNAから離れていく。同時にコア酵素もゲノムDNAから離脱する。
- ρ依存性終結 rho-dependent termination
- 一方ρ依存性終結ではρ因子 rho factor というタンパク質がステムループ構造の5'側に結合し、mRNAと鋳型DNAの塩基対を破壊して転写が終結する。
mRNAは3'側にも遺伝子配列そのものよりも長い配列を持っており、この配列をトレイラーセグメントと呼ぶ。転写の終結したmRNAは、輸送や修飾などは特に行なわれず、すぐに翻訳過程に向かう。 スプライシング反応をはじめとした様々な修飾を施す真核生物とは大いに異なる。
ρ非依存性終結
[編集]
この場合のターミネーターはタンパク質の補助なしで独自でRNAポリメラーゼに作用し、転写産物を解放させる。内因性ターミネーター intrinsic terminator というこの塩基配列は、20ntほどの逆位反復領域とそのすぐ下流にある8ntほどのA(アデニン)高含有領域で構成される[11]。逆位反復領域と相補的なRNAはヘアピン構造を形成する特徴があり、転写終結の原動力となる。
ρ非依存性終結の典型例として、ペギー・ファーナム Peggy Farnham とテリー・プラット Terry Platt は大腸菌のトリプトファンオペロンを研究した。内因性ターミネーターの2つの要素を兼ね備えるtrp アテニュエーター trp attenuator があるからである。これの逆位反復配列を転写したRNAが形成するヘアピン構造を下図に示す[12]。完全な逆反復ではないが、8bpの塩基対形成が可能であり、そのうち7bpは強いG-C塩基対である。また、ファーナムとプラットは、T高含有配列が転写されるにつれRNAのUとDNAのAとの間に塩基対(rU-dA)が形成すると考えた[13]。この塩基対の融解温度はrU-dAもしくはrU-rAよりも20℃低い[13]。2つの要素についての考察から得られる結論は、T高含有配列でRNAポリメラーゼは一時的に停止し、次にヘアピン構造が形成されることにより弱い結合が切られるというものである。ヘアピン構造はRNAポリメラーゼを押して転写産物から離すか、何らかの立体構造変化を引き起こすものと考えられている[11]。W. S. ヤーネル W. S. Yarnell とジェフリー・ロバーツ Jeffrey Roberts は転写終結において2つの要素は必ずしも必要ではないことを明らかにした[14]。必要なのは、転写産物と極端に弱い塩基対形成する配列と、一時的に転写を停止させる何かである。
A /| U U C G G G C G G A | | | | | | | | A G C C C G C C A U
ρ依存性終結
[編集]
ρ因子は全て同じサブユニットから構成された六量体である。全体は、転写産物とDNAとの二重らせんを通す環構造となっているが、この輪は一部が切れている。そのうえ、切れ目を隔てた2つのサブユニットは12Åほど輪の軸方向に離れていて、すなわち小さならせん(ピッチ[要曖昧さ回避]は45Å)となっている。[11]。サブユニットは全てRNAと結合する部位を持ち、次から次へとRNAに結合するサブユニットをジグザグに変えると考えられている[15]。
ρ因子はDNAの転写終結部位の上流にあるρ因子結合部位 rho loading site で転写産物に結合する。この配列はシトシンを多く含む60〜100ntで、比較的ランダムな二次構造を形成している[15]。ρ因子が実際に結合するには先の条件を満たす40ntの配列が最もいい[9]。このρ因子結合部位を転写することで、実際に迎え入れるrut部位 Rho utilization sites:rut sites という配列がRNAに現れる。ただし、リボソームと結合し、セントラルドグマにおける次の段階の翻訳を受けているものとは結合できない[9]。細菌では転写と翻訳は密に連携し、伸長がまだ終わっていなくてもポリメラーゼから出てきた転写産物はすぐに翻訳を始められる[16]。したがって、ρが集結するのは、遺伝子やオペロンの末端を過ぎても転写が続いている転写産物だけである。
6つのサブユニットは全てATPアーゼである。この活性により、必要以上に長くなってしまった転写産物に結合したときエネルギーが供給され、回転しながら5’から3’へと進む。ターミネーターのヘアピン構造形成で停止したRNAポリメラーゼに追いつくと、転写産物を放出させる。具体的な終結の過程はまだ断定されていない。1987年にテリー・プラットはρ因子がRNA-DNAヘリカーゼ活性を持つことを発見したので、一説では、RNAポリメラーゼと接触したときに二重らせんをほどいて放出させるとする[15]。ほかには、RNAポリメラーゼを後ろから押して転写産物から離すとする説や、RNAポリメラーゼの立体構造を変化させるとする説がある[9]。
真核生物の場合
[編集]真核生物の場合、細菌プロモーターの-10領域に相当する、5'-TATAAA-3'の共通配列を持つ領域(TATAボックス、あるいは、ゴールドバーグ・ホグネスボックス (Goldberg-Hogness box) と呼ばれる)が-25あるいはさらに上流に存在する。転写開始位置はこのTATAボックスが主となって決定している。
この他、-100〜-60の範囲に存在する5'-CCAAT-3'の共通配列を持つ領域(CAATボックスと呼ばれる)や、-60〜-40の範囲に存在する5'-GGCGGG-3'の共通配列を持つ領域(GCボックスと呼ばれる)がよく知られているが、これらは転写の促進に働いていると考えられている。
真核生物の場合、RNAポリメラーゼには3つの種類があり、それぞれPol I,Pol II,Pol IIIと呼ばれている。それぞれ、転写開始に必要となる因子、プロモーター領域の配列、転写の様式が異なっている。大部分の遺伝子は転写をPol IIに依存しているが、rRNAはPol Iに、tRNAはPol IIIに依存している。
Pol I系遺伝子(rRNA)における転写の開始
[編集]RNAポリメラーゼIによる転写においては、プロモーターは、-200〜-65の範囲に存在する上流制御要素(UCE; upstream controling element)と呼ばれる領域と、-45〜+20の範囲に存在するTATAボックスを含むコアプロモーター(CPE; core promoter element)と呼ばれる領域の二つの部分からなる。
転写に関与する因子としては、
- UBF:UCE結合因子
- SL1
の二つが知られている。UBFはUCEに結合し、SL1のTATAボックスへの結合を促進すると言われている。SL1はCPEへの弱い結合能を示すが、転写の開始において必須である。SL1はまた、Pol Iとも会合する。
SL1は更に、
- TBP:TATA結合蛋白質
- TAF:Pol I会合因子
- TAFI 110
- TAFI 68
- TAFI 48
といったの因子の複合体となっている。
また、転写伸長に関わる因子として、
- TIF-1C
も知られている。
Pol II系遺伝子(構造遺伝子など大部分の遺伝子)における転写の開始
[編集]RNAポリメラーゼIIによる転写に必要な酵素以外の基本転写因子蛋白質をTFII (Transcription Factor for Pol II) シリーズと呼び、現在、
- TFIIA: TFIIDのDNAへの結合を促進
- TFIIB: TFIIDとともに転写の最小開始複合体形成に参加
- TFIID: プロモーター領域を認識し、TATAボックスに結合
- TFIIE: TFIIHの制御
- TFIIF: Pol IIと結合。Pol IIの転写開始複合体参加に必須
- TFIIH: DNA依存ATPase。Pol IIをリン酸化し変化させ、転写開始複合体から各因子を遊離させる
- TFII-I:
の七種類が知られている。
また、転写開始後、RNA伸長を促進する因子として、
- TFIIS
も存在する。
転写開始への流れとしては、
- プロモーター領域(TATAボックス近辺)にTFIIAが結合
- TATAボックスにTFIIDが結合、TFIIAと複合体を形成
- TFIIA-TFIID-DNA複合体にTFIIBが結合
- RNAポリメラーゼII(Pol II)にTFIIFが結合
- TFIIF-Pol II複合体にTFIIEが結合
- TFIIF-TFIIE-Pol II複合体にTFIIHが結合
- TFIIF-TFIIE-TFIIH-Pol II複合体がTFIIA-TFIID-TFIIB-DNA複合体と結合
- TFIIHの作用によりPol IIがリン酸化される
- Pol II変形
- TFIIHの作用により、転写開始複合体はTFIIA-TFIID-TFIIB複合体とTFIIF-Pol II複合体に分離される。この際、TFIIE, TFIIHは放出される。(プロモータークリアランスという)
- TFIIF-PolII-DNA複合体にTFIISが結合する
- RNAの転写が進行する
となっている。
TATAボックスは、コアプロモーターエレメント(CPE; core promoter element、コアプロモーター要素、時にcore promoter motifとも呼ばれる)の1つである。
コアプロモーター (core promoter) とは、正確な転写開始を導く働きをもつプロモーター領域のことであり、一般に転写開始点を含む±35塩基ほどの長さの領域を含むが、多くの例から、約40塩基の領域から構成されていると考えられている。コアプロモーターの中には幾つかのシーケンスモチーフが存在し、これをコアプロモーターエレメントと呼ぶ。
コアプロモーターエレメントは、すべてのコアプロモーターに普遍的に存在するものではない。むしろ、個々のコアプロモーターの特異性を与えるものである。教科書等を見ると、TATAボックスが全ての遺伝子のコアプロモーターに存在しているような印象を受けるが、実際は違い、例えば酵母に関する最近の研究においては、TATA-containing core promoter(TATAボックスを含むコアプロモーター)は、わずか約19%であったと報告している。
-2〜+4の範囲に存在するイニシエーターエレメント(Inr; initiator, PyPyANT/APyPy, Aが転写開始位置になる)もコアプロモーターエレメントである。Inrの認識はTFIIDによっておこなわれる。このほかに、DPE (downstream core promoter element)、MTE (motif ten element) が発見されている。
Pol III系遺伝子 (tRNA) における転写の開始
[編集]RNAポリメラーゼIIIによる転写に必要な酵素以外の基本転写因子蛋白質をTFIII (Transcription Factor for Pol III) シリーズと呼び、現在、
- TFIIIA:
- TFIIIB:
- TFIIIC:
の三つが知られている。その他にも、
- TBP:TATA結合蛋白質
- PTF:PSE結合蛋白質
などの因子も存在する。
RNAポリメラーゼIIIによって転写される遺伝子の場合、転写開始の機作が、そのプロモーター領域の構造により
- 遺伝子構造の内部にAブロック(+20の位置)、Bブロック(+51から+113の位置)の二つのコアプロモーター(転写の開始に際し必須の配列)を持つもの。TFIIIB,TFIIICの二つが必要。
- 遺伝子構造の内部にAブロック、Iブロック、Bブロックの二つのコアプロモーター(転写の開始に際し必須の配列)を持つもの。TFIIIA,TFIIIB,TFIIICの三つが必要
- TATAボックス(-25の位置)、PSE(近位配列要素;-55の位置)を持つもの。TBP,TFIIIB,PTFの三つの因子が必要
の三つの様式に分かれる。
古細菌の場合
[編集]古細菌における転写は基本的には真核生物のものを簡単にしたものと考えてよい。転写開始位置付近には真核生物とほぼ同じ位置にBRE、TATAボックス、イニシエーターエレメントなどが配置されている。TATAボックスにRNAポリメラーゼ、TFIIB、TFIID、TFIIEより成る複合体が形成され(TFIIA、TFIIF、TFIIHは見つかっていない)、これらを足掛かりにRNAポリメラーゼが転写を開始する。TFIISも存在する。詳細は#Pol II系遺伝子参照。
転写因子
[編集]転写因子は転写そのものに関わる基本転写因子と、転写の調節を行う転写調節因子(--制御因子)がある。前者はRNAポリメラーゼ複合体やTATA結合タンパク質などが含まれる。転写開始後の伸長反応に機能する転写伸長因子を含むこともある。後者は転写制御配列のDNAに結合し、基本転写因子の活性を制御する特異的転写因子が含まれる。直接 DNA には結合せずクロマチンの構造変換を行うヒストン修飾酵素やクロマチン再構成因子を含むこともある。
基本転写因子
[編集]転写調節因子
[編集]この節の加筆が望まれています。 |
転写伸長因子
[編集]転写伸長に必要とされる。ヌクレオソームは転写の伸長を妨げるため、転写伸長因子が転写装置とともに移動する。
この節の加筆が望まれています。 |
クロマチン構造
[編集]真核生物のDNAはヒストンという蛋白に巻きついている (ヌクレオソーム)。一般的にはヒストンがアセチル化されることでクロマチン構造がゆるみ、結果として発現が活性化される。また、ショウジョウバエなどのGAGA因子と呼ばれるタンパク質はDNAに結合し、その周辺のクロマチン構造を変化させる。NAP-1がヒストンをDNAに貼り付け、ACFがそれを移動させて一定間隔にする。
細菌ではHUタンパクがDNAと結合し核様体を形成するがクロマチン構造はとらない。
一方多くの古細菌はヒストンを持ち、DNAが巻きついてクロマチン様構造をとっている。
DNAのメチル化
[編集]また、DNAのメチル化なども重要なキーワードの一つである。メチル化DNAはゲノムインプリンティングなどに関与するとされる。メチル化DNAを特異的に認識するタンパク質(メチル化DNA結合タンパク質, MBD)は、クロマチン構造を変換する酵素複合体を誘導することが考えられている。
一般にDNAのメチル化は転写の抑制となり、特定の遺伝子が特定の組織で発現するメカニズムの原因であると考えられている。また、DNAのメチル化はヒストンのアセチル化やメチル化と結びついており、細胞分裂の際にコピーされた染色体にヒストンのアセチル化やメチル化が引き継がれる。
逆転写
[編集]RNAウイルスは、DNAから行われる通常の転写とは異なりRNAに遺伝情報を記録し、RNAからmRNA合成を行うが、これも転写の範疇に含まれる。ただし、核酸から核酸への合成はほかにDNA複製の過程でも行われるが、これは生成物がDNAであり、遺伝子の発現に直接関わってもいないため転写と異なる。mRNAからDNAへの合成は逆転写と呼ぶ。
注釈
[編集]- ^ 二重らせんを形成しているDNAを二本鎖DNA double-stranded DNA、一本だけの状態で遊離しているものを一本鎖DNA single-stranded DNA と呼ぶ。それぞれdsDNA、ssDNAと略す。
- ^ a b RNAポリメラーゼが転写を実行するためにDNA上に構築する複合体をRNAポリメラーゼホロ酵素もしくは単にホロ酵素と呼ぶ。α1,α2、β、β’、σ、ωの6つのサブユニットから構成され、全体はカニのはさみに似ている。1つで複合体のほぼ半分を占める2つの爪はそれぞれ巨大なβ、β’サブユニットで、この内部を活性中心溝という。転写の活性部位は活性中心溝の奥、爪の付け根に存在する。
- ^ 開放型プロモーター複合体形成に伴う、ssDNAの領域を転写バブルという。その大きさについては様々な方法での測定例がある。1978年にTao-shih HsiehとJames Wangは、大腸菌のRNAポリメラーゼはT7ファージの初期プロモーターを約10bpほど分解すると算定した。1979年にウルリッチ・ジーベンリスト Ulrich Siebenlist が行った実験では、観測されたのは12bpだった。ただし、この結果は実際よりも小さい可能性が指摘されている。これらの実験も、それ以前のほかの実験もRNAポリメラーゼとDNAだけが結びついた複合体を対象としている。すなわち、RNA合成前の転写初期におけるRNAポリメラーゼである。
- ^ a b RNAポリメラーゼにはDNA用の出入り口が5つある。NTP取り込み通路は[[基質 (化学)|]]であるリボヌクレオチドを触媒活性中心に迎え入れる。RNA出口通路は後の伸長段階で合成したRNA鎖の部分を出すためにある。下流のdsDNAは下流DNA用通路から活性中心溝に入り、ssDNAに分かれる。非鋳型鎖は非鋳型鎖用通路(NT通路)から、鋳型鎖は活性部位を通って鋳型鎖用通路(T通路)から外に出る。2つの一本鎖はホロ酵素の後方で二重らせんに戻る。
- ^ σサブユニットは転写開始に最も重要で、ホロ酵素をプロモーターへと誘導するなどの必須な役割を持つ。転写開始が転写において最も遅い段階であるため、σ因子は役割を終えると再利用のため直ちにホロ酵素から分離される。そして、別のコア酵素へと供給される。この循環をσサイクルという。
- ^ TRCFはdsDNAに結合すると、 ATPアーゼ活性を活用して転写と同じ5’から3’の方向へ滑るように移動する。停止したRNAポリメラーゼがあれば衝突し、解離させる。損傷の先へ押されて伸長を再開することもある。
出典
[編集]- ^ a b c 『ワトソン 遺伝子の分子生物学 第6版』、著者:James D. Watsonほか、監訳者:中村桂子、発行:学校法人東京電機大学出版局(2010)、p382
- ^ 『ウィーバー 分子生物学 第4版』、化学同人、著者:Robert F. Weaver、監訳者:杉山弘、2008、p42
- ^ a b 『遺伝子第8版』、p230
- ^ 『遺伝子 第8版』、著者:Benjamin Lewin、訳者:菊池菊池韶彦(あきひこ)、東京化学同人、2006、p229
- ^ a b c d 『ワトソン 遺伝子の分子生物学』、p389
- ^ a b c 『ワトソン 遺伝子の分子生物学』、p390
- ^ a b c d 『ワトソン 遺伝子の分子生物学』、p391
- ^ a b c 『ウィーバー 分子生物学第4版』、p172
- ^ a b c d 『ワトソン 遺伝子の分子生物学』、p394
- ^ 『ワトソン 遺伝子の分子生物学』、p274
- ^ a b c 『ワトソン 遺伝子の分子生物学』、p395
- ^ 『ウィーバー 分子生物学』、p173
- ^ a b 『ウィーバー 分子生物学』、p174
- ^ 『ウィーバー 分子生物学』、p176
- ^ a b c 『ウィーバー 分子生物学』、p179
- ^ 『ワトソン 遺伝子の分子生物学』、p395
関連映像
[編集]- 理化学研究所オミックス基盤研究領域「HD セントラルドグマ -synra editon- 日本語ナレーション版」2008年、YouTube(CG ムー ビー)
- 理化学研究所ゲノム科学総合研究センター遺伝子構造・機能研究グループ「セントラルドグマ ~ゲノム情報からタンパク質ができるまで~ / The Central Dogma」2007年、YouTube(CG ムー ビー)
参考文献
[編集]- 『ワトソン 遺伝子の分子生物学 第6版』、著者:James D. Watsonほか、監訳者:中村桂子、東京電機大学出版局(2010)
- 『ウィーバー 分子生物学 第4版』著者:Robert F. Weaver、監訳者:杉山弘、化学同人、2008
- 『遺伝子 第8版』、著者:Benjamin Lewin、訳者:菊池韶彦、 水野猛, 榊佳之, 伊庭英夫 、東京化学同人、2006
