RNA治療
RNA治療(英: RNA therapeutics)またはRNA治療薬は、リボ核酸(RNA)に基づく薬物の一群である。主な種類には、メッセンジャーRNA(mRNA)、アンチセンスRNA(asRNA)、RNA干渉(RNAi)、およびRNAアプタマーに基づくものがある。
4種類のうち、mRNAに基づく治療は、細胞内のタンパク質合成をトリガーした唯一の治療法であり、ワクチン開発で特に有用であると考えられている[1]。2020年には、新型コロナウイルス感染症(COVID-19パンデミック)の対策に向けてmRNAワクチンが開発されている[2]。アンチセンスRNAは、mRNAのコードに相補的で、mRNAの不活性化を引き起こしてタンパク質翻訳で利用されないようにするために使用される[3]。 RNAiベースのシステムも同様のメカニズムを使用しており、mRNAの翻訳を防ぐために低分子干渉RNA(siRNA)とマイクロRNA(miRNA)の両方を使用している[4][5]。しかし、RNAアプタマーは、定向進化によって生成された短い一本鎖RNA分子で、さまざまな生体分子標的に高い親和性で結合することで、生体内(in vivo)での正常な活性に影響を及ぼすl[6][7][8]。
RNAはRNAポリメラーゼ酵素によって鋳型DNAから合成され、メッセンジャーRNA(mRNA)はDNAの発現とタンパク質の翻訳の間の中間生体分子として機能している。その独特の特性(典型的な一本鎖の性質やその2'OH基など)や、多くの異なる二次構造/三次構造の形状をとる能力のために、ボットコーディングとノンコーディングRNAは、医学の分野で特別な注目を集めている。RNAを治療に利用する可能性を探る研究が始まっており、RNA治療の創薬と実施の間には、ユニークな課題が発生している[9]。
mRNA[編集]
メッセンジャーRNA(mRNA)は、遺伝子のDNA鎖の1本に相補的に結合している一本鎖RNA分子である[10]。mRNA分子は、タンパク質を作るためにDNAコードの一部を細胞の他の部分に転送する[11]。DNA治療薬は、RNAに転写されるために核へのアクセスを必要とし、その機能は細胞分裂中の核エンベロープの破壊に依存する。しかし、mRNA治療薬は、細胞質に到達するとすぐに翻訳されるので、機能するために核に入る必要はない[12]。さらに、プラスミドやウイルスベクターとは異なり、mRNAはゲノムに組み込まれないため挿入変異を誘発するリスクがなく[13]、がんワクチンや腫瘍免疫療法、感染症予防などの用途に適している。[14]。
発見と開発[編集]
1953年、アルフレッド・ハーシーは、細菌がファージに感染した直後に高いレベルでRNAの形を作り出し、このRNAも急速に分解されることを報告した[15]。しかし、mRNAを最初に明確に示したのは、1956年、大腸菌にT2バクテリオファージを感染させ、それらを32Pを添加した培地に入れた、エリオット・ヴォルキンとラザロ・アストラチャンの研究である。彼らは、大腸菌のタンパク質合成が停止し、ファージのタンパク質が合成されることを発見した[16]。その後、1961年5月、共同研究者のシドニー・ブレナー、フランソワ・ジャコブ、ジム・ワトソンがmRNAの単離を発表した[17][18]。mRNAの発見から数十年の間、人々はmRNAの構造、機能、代謝経路の側面を理解することに焦点を当てていた。しかし、1990年、Jon A. Wolffは、マウスの骨格筋にインビトロ転写(IVT)mRNAまたはプラスミドDNA(pDNA)を直接注入し、注入された筋肉内でコード化されたタンパク質を発現させることで、核酸コード化された薬物のアイデアを実証した[19][20]。
IVT mRNAが細胞質に到達すると、mRNAは即座に翻訳される。したがって、それが機能するために核に入る必要はない[21]。また、それはゲノムに組み込まれないため、挿入変異誘発のリスクもない[22]。さらに、IVT mRNAは一過性にしか活性化されず、生理的な代謝経路を介して完全に分解される[23]。このような理由から、IVT mRNAに関する前臨床研究が広範囲で行われている。
機構[編集]
インビトロ転写(IVT)は、標的のコード配列を含む線状化DNAプラスミド鋳型を用いて行われる。次に、裸のmRNAやナノ粒子に複合化したmRNAは、全身または局所的に送達される。その後、外因性の裸のmRNAまたは複合体化されたmRNAの一部が細胞特異的な機構を経ている。細胞質に入ると、IVT mRNAはタンパク質合成機構によって翻訳される[24][25]。
RNAセンサーとして、Toll様受容体(TLR)とRIG-I様受容体ファミリーの2つが同定されている。TLRは、樹状細胞(DC)やマクロファージなどの細胞のエンドソーム区画に局在している[26]。RIG-I様ファミリーは、パターン認識受容体(PRR)である[27]。しかし、細胞センサーによるmRNAワクチン認識の免疫応答機構やプロセス、センサー活性化の機構は未だ不明である[25]。
応用例[編集]
がん免疫療法[編集]
1995年、ロバート・コンリーは、癌胎児性抗原をコードする裸のRNAを筋肉内注射すると、抗原特異的な抗体応答が得られることを実証した[28]。次に、特定の抗原をコードするmRNAや腫瘍細胞から抽出した全mRNAに曝露した樹状細胞(DC)を担癌マウスに注入すると、T細胞免疫応答が引き起こされ、腫瘍の増殖が抑制されることを実証し、さらに発展させた[29]。その後、研究者は、体外(ex vivo)IVT mRNAトランスフェクトDCに基づくワクチンを用いて、mRNAトランスフェクトDCへのアプローチを開始した[30]。一方、Argos Therapeutics社は2015年に進行性腎細胞癌のDCを用いた第III相臨床試験を開始していたが(NCT01582672)、有効性が認められず中止となった[31]。
さらなる応用のために、IVT mRNAを生体内(in vivo)でのDCのin situトランスフェクション用に最適化した。これにより、IVT mRNAの翻訳効率と安定性を改善させ、MHCクラスIおよびクラスII分子上でのmRNAコード化抗原の提示を強化した[32][33]。そして、裸のIVT mRNAをリンパ節に直接注入することが、T細胞応答を引き起こす最も効果的な方法であることを発見した[34]。この発見をもとに、BioNTech社によるがん抗原をコードする裸のIVT mRNAのリンパ節への直接注入のヒト初回試験が、メラノーマ患者を対象に開始された(NCT01684241)[35]。
最近、自己送達RNA(sd-rxRNA)と養子細胞移植(ACT)療法を組み合わせた新しいがん免疫療法が、フィオ・ファーマスーティカルズ社とカロリンスカ研究所によって発明された。この治療法では、sd-rxRNAが治療用免疫細胞における免疫抑制受容体やタンパク質の発現を除去するため、免疫細胞が腫瘍細胞を破壊する能力を向上させた。次に、PD-1を標的としたsd-rxRNAは、メラノーマ細胞に対する腫瘍浸潤リンパ球(TIL)の抗腫瘍活性を高めた[36][37]。このような考えに基づき、mRNA-4157は試験され、第I相臨床試験に合格している[38]。
ワクチン[編集]
1993年、インフルエンザの核タンパク質をコードするIVT mRNAをリポソームでカプセル化し、ウイルス特異的なT細胞を誘導することにより、マウスを用いたmRNAワクチンの最初の成功が報告された[39]。その後、合成脂質ナノ粒子を用いてIVT mRNAを製剤化し、マウスの呼吸器合胞体ウイルス(RSV)およびインフルエンザウイルスに対する防御抗体反応を誘導した[40]。
IVT mRNAに基づいた感染症用ワクチン開発には、いくつかの種類がある。成功した種類の一つは、プラス鎖RNAウイルスの配列を持つ自己増幅型IVT mRNAを用いたものである。もともとはフラビウイルス用に開発されたもので、皮内注射で使用可能であった。他の方法の1つは、インフルエンザヘマグルチニン抗原をコードするmRNAアジュバント(免疫賦活剤)と裸のIVT mRNAのみを含む、またはIVT mRNAをコードするノイラミニダーゼとの組み合わせを含む、2液混合ワクチンを注射する方法がある[41]。
たとえば、HIV治療のために、HIVタンパク質をコードするIVT mRNAをトランスフェクトしたDCを用いたワクチンが使用されている。IVT mRNAをコードする組み合わせを用いた第I相および第II相臨床試験がいくつか行われており、抗原特異的なCD8+およびCD4+ T細胞応答が誘導できることが示されている。しかし、臨床試験では抗ウイルス効果は観察されていない[42][43]。
他のmRNAワクチンの一つにCOVID-19ワクチンがある。2019年12月に重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)が大流行し、世界中に広がり、コロナウイルス感染症2019(COVID-19)と命名された呼吸器疾患のパンデミックを引き起こした[44]。モデルナ社が製造するmRNA-1273ワクチン候補は、脂質ナノ粒子(LNP)でカプセル化されたmRNAに基づくワクチンで、膜貫通型アンカーを持つSARS-CoV-2の完全長融合前安定化スパイク(S)-2P抗原をコードしている[45][46]。2020年11月16日、モデルナは、COVID19ワクチン候補のmRNA-1273が、第III相COVID-19試験の第1次中間分析において、94.5%のワクチン有効率で主要な有効性評価項目を満たしたと発表した[47]。2020年12月21日、アメリカ疾病対策予防センター(CDC)は、モデルナのmRNA-1273の成人への緊急使用許可を推奨した[48]。
制限事項[編集]
mRNAは、非常に大きくて重い分子であるため(105〜106 Da)、薬物への翻訳を成功させるためには多くの課題がある[49]。さらに、mRNAは不安定でヌクレアーゼによって分解されやすく、免疫系も活性化する[50]。そのうえ、mRNAは負電荷密度が高く、細胞膜を通過するmRNAの透過性は低下する。このような理由から、適切な送達システムがないとmRNAは分解されやすく、送達システムがない場合のmRNAの半減期は7時間程度しかない[51]。化学修飾によってある程度の課題は克服できても、mRNAの送達は依然として大きな障害として残っている。mRNAの送達システムを改善するために研究されてきた方法としては、マイクロインジェクション、RNAパッチ(溶解マイクロニードルにmRNAを装填したもの)、遺伝子銃、プロタミン縮合、RNAアジュバント、脂質を含むナノ粒子へのmRNAのカプセル化などがある[49][52][53]。
インビトロ転写(IVT) mRNAに送達剤を添加した場合、分解に対する耐性が改善されたとはいえ、生体内(in vivo)での裸のmRNAの送達効率を向上させるためには、さらなる研究が必要である[20]。
アンチセンスRNA[編集]
アンチセンスRNAは、mRNAのコーディング配列に相補的なノンコーディング一本鎖RNAである。これは、mRNAがタンパク質に翻訳される能力を阻害する[54]。短いアンチセンスRNA転写物は、二本鎖RNA前駆体を21~26ヌクレオチド長のRNA種に切断する酵素ダイサーの作用によって核内で生成される[3]。
アンチセンスに基づく発見戦略、スクリーニングアッセイの理論的根拠および設計、ならびに天然物抽出物のスクリーニングおよび脂肪酸縮合酵素阻害剤の発見のためのアッセイ応用がある[55]。アンチセンスRNAは、がんの治療、転移の抑制、アンチセンス隔離のためのベクターとして使用されている。特にマイクロRNA(miRs)15および16は、がんの診断および予防のための治療を必要とする患者に使用される[56]。アンチセンス薬は、アンチセンスRNAがmRNAとハイブリダイズして不活性化することに基づいている。これらの薬物は、mRNAに結合し、特定の遺伝子がコードするタンパク質を産生しないようにする短いRNAの配列である。アンチセンス薬は、肺がんや糖尿病、関節炎や喘息など主要な炎症性要素を伴う疾患の治療薬として開発されている[57]。MLLT4アンチセンスRNA 1(MLLT4-AS1)の発現低下は、胃がんの予後不良のバイオマーカーとなりうることを示している。これまでに、アンチセンスRNAの抗ウイルス、抗がん剤治療への応用や、植物や微生物における関連遺伝子の発現調節への応用が検討されてきた[58]。
アンチセンス RNA を細胞膜を介して細胞質や核に送達するために、非ウイルスベクター、レトロウイルスやアデノウイルスなどのウイルスベクター、リポソームなどが使用されてきた。その中でも、ウイルスベクターを用いた送達は、さまざまな送達システムの中で、高いトランスフェクション効果を持つことから、最も有利であることが明らかになった[59]。しかし、アンチセンスRNAを標的部位にのみ送達することは困難であり、また、アンチセンスRNAの大きさや安定性の問題もあり、利用には限界がある。送達の問題点を改善するために、化学修飾や新規オリゴヌクレオチドの設計などが研究されており、薬物分布、副作用、忍容性の強化が図られている[60][61]。
RNAi[編集]
RNA干渉(Interfering RNA)は、翻訳または翻訳後の遺伝子発現を抑制するように作用する短いノンコーディングRNAの一群である[62][4]。これらの発見と、その後の転写後遺伝子調節の鍵となるエフェクターとしての同定により、低分子干渉RNA(siRNA)とマイクロRNA(miRNA)は、全身性疾患の潜在的な治療薬として可能性があると考えられている[62][4][63]。RNAiシステムは、1990年にペチュニアに着色遺伝子を導入する研究をしていたヨルゲンセンらによって発見されたもので[63][64]、もともとは二本鎖RNAウイルスに対する自然免疫の手段として発展されたと考えられている[65]。
siRNA[編集]

低分子干渉RNA(siRNA)とは、19~23塩基対(2塩基の3'オーバーハングを持つ)の短い二本鎖RNAのことで、遺伝子サイレンシングのためのRNA誘導サイレンシング複合体(RISC)に関与している[4][63]。具体的には、siRNAは、ATP加水分解を利用して巻き戻されるRISC複合体に結合している[63][66][67]。次に、スライサー酵素により、標的mRNAと相補的な塩基対関係に基づいて、標的mRNAを分解するためのガイドとして利用される[63][66][67]。治療薬として、siRNAは目や鼻から局所的に送達され、さまざまな疾患を治療することができる[4]。局所的送達の利点は、単純な製剤化と薬物送達、および薬物の高いバイオアベイラビリティーである[4]。がんやその他の疾患を標的とするには、全身送達が必要である[4]。局所的に送達された場合のsiRNAの標的化は、siRNA治療薬における主要な課題の一つである[4]。siRNA治療薬を送達するために静脈内注射を使用することは可能であるが、注射で使用される大量の薬剤は多くの場合、全血液量の20〜30%でなければならないため、懸念が生じている[63]。その他の送達方法としては、リポソームパッケージング、膜透過性ペプチドへの結合、組織/臓器への直接エレクトロポレーション(電気穿孔法)などがある[63]。さらに、外因性siRNAは、生体内(in vivo)では数日(非分裂細胞ではせいぜい数週間)しか持続しないことがわかっている[68][69]。siRNAが標的にうまく到達できれば、RISCシステムを介してmRNA標的と塩基対を形成し、その分解を促進することで、遺伝子発現を治療的に制御する可能性があると考えられている[4][63]。現在、siRNAに基づいた治療法は、加齢黄斑変性症を対象とした第I相臨床試験が行われているほか[63]、がん治療への応用も検討されている[70]。たとえば、siRNAは、VEGF受容体やテロメラーゼ酵素などの腫瘍成長を促進するタンパク質をコードするmRNAを標的にして使用することができる[63]。
miRNA[編集]
マイクロRNA(miRNA)は、19~23塩基対長の短いRNAオリゴヌクレオチドであり、RNA誘導サイレンシング複合体に関与している[63][5]。具体的には、いったんアルゴノート酵素にロードされると、miRNAはmRNAと協働して翻訳を抑制したり、翻訳後にmRNAを不安定化させたりする[5]。それらは機能的にはsiRNAと似ているが、miRNAはmRNAサイレンシングのために広範な塩基対を必要としないため(標的との塩基対はわずか7個)[71][72]、より広範囲のmRNA標的に影響を与えることができる[73]。細胞内では、miRNAはスイッチ、チューニング、そしてニュートラルな相互作用を利用して、遺伝子抑制を細かく調整する[5]。治療薬として、miRNAは、生体内の生化学的経路に影響を与える可能性がある[62]。
ヒトでは400以上のmiRNAが同定されており、その抑制の標的遺伝子を見極めることが最初の課題となっている[5]。miRNA seedマッチングを利用して、TargetScanなどの複数のデータベースが構築されている[62]。生体外(in vitro)アッセイは、miRNAの表現型への影響を決定するのに役立つが[62]、遺伝子制御の複雑な性質のため、同定されたすべてのmiRNAが期待される影響を持つわけではない[5]。さらに、発がん性のmiR-155およびmiR-17-92のように、いくつかのmiRNAは、生体内(in vivo)で腫瘍抑制因子または発がん遺伝子として作用することがわかっている[73]。
臨床試験では、miRNAはさまざまな疾患のバイオマーカーとして一般的に使用されており、早期診断、疾患の進行、病期、遺伝的関連性の提供が期待されている[62]。現在、がんやその他の疾患の患者を対象に、miRNA mimic(遺伝子を発現させる)およびmiRNA(遺伝子を抑制する)の試験が第I相および第II相試験として行われている[62]。特に、mimic miRNAは、腫瘍抑制因子として作用するmiRNAをがん組織に導入するために用いられ、miRNAアンタゴニストは、がん促進型miRNAを標的にしてがん化促進作用を阻害するために用いられている[73]。患者のmiRNAレベルを過剰発現または不安定化させることが知られている一般的な治療法(例えば、がん治療薬)に加えて、治療用miRNAも使用される[62]。マウス研究において、肺がん腫瘍の増殖を抑制する効果を示したmimic miRNA治療の例として、miR-34aがある[73][74]。
miRNAに基づく治療法に対する1つの懸念事項は、外因性のmiRNAが正常な体細胞内のmiRNAサイレンシング機構に影響を与え、それによって正常な細胞の生化学的経路に影響を与える可能性である[73]。しかし、生体内(in vivo)での研究では、miRNAは非標的の組織/器官ではほとんど、またはまったく効果を示さないことが示されている[74][75]。
RNAアプタマー[編集]

おおまかに、アプタマーは一本鎖DNAまたはRNAのいずれかで構成される低分子であり、一般的には20〜100ヌクレオチド長[6][7][77]、つまり3〜60 kDaである[77][78]。アプタマーは一本鎖であるため、鎖内の塩基対の相互作用により、シュードノット、ステムループ、およびバルジ(膨らみ)などの多くの二次構造を形成することができる[77]。アプタマーに存在する二次構造の組み合わせは、アプタマーに特定の三次構造を与え、それによって、アプタマーが選択的に結合する特定の標的が決定される[77][79]。アプタマーは選択的に結合する能力を持つため、医薬品への応用が期待される生体分子として検討されている[6][7][77]。さらに、アプタマーは標的への強固な結合を示し、解離定数はpM~nMの範囲にあることが多くある[8][6]。アプタマーは強力な結合力の他に、ファージディスプレイや抗体によって生成された小型ペプチドでは結合できない標的にも利用でき、構造異性体とアミノ酸置換を区別できる点も評価されている[77][80][81]。また、アプタマーは核酸に基づくため直接合成することができ、抗体作成のように細胞ベースでの発現や抽出が不要である[7][82]。特にRNAアプタマーは、無数の異なる構造を作り出すことができるため、DNAアプタマーに比べて標的親和性の識別性が高いと推測されている[77][83]。
発見と開発[編集]
アプタマーは、1990年にLary GoldとCraig TuerkがSELEXとして知られている定向進化の方法を利用して、T4バクテリオファージDNAポリメラーゼに結合することができる小さな一本鎖RNA分子を単離したときに最初に発見された[7][84]。さらに、「アプタマー」という用語は、アンドリュー・エリントンによって造語されたもので[77][85]、彼はジャック・ショスタクと協同で、特定の有機色素分子に強固に結合することができるRNAアプタマーを選択した。この用語自体は、ラテン語の "aptus" または "to fit" とギリシャ語の "meros" または "part" の複合語である[77][85]。
RNAアプタマーは「作られた」のではなく「選ばれた」のである。分子標的に選択的に結合できるRNAアプタマーを開発するために、試験管内進化法 (SELEX)として知られている方法を使用して、1013~1016の異なるアプタマーのプール(別名ライブラリとして知られる)からユニークなRNAアプタマーを単離する[6][7][77][84][85]。次に、潜在的なアプタマーオリゴヌクレオチドのライブラリは、非標的種とインキュベートして、非特異的な結合を示すアプタマーを除去する[6]。その後、非特異的アプタマーを除去した後、残りのライブラリーメンバーは次に、タンパク質、ペプチド、細胞型、あるいは器官(生きた動物ベースのSELEXの場合)などの目的の標的に曝露される[6][77][86][87][88][89][90][91]。そこから、標的に結合したRNAアプタマーをcDNAに転写し、PCRによりcDNAを増幅し、PCR産物をRNAに再転写する[77]。これらの新しいRNA転写物は、その後、選択サイクルを何度も繰り返すために用いられ、最終的には、特異的が高く、親和性が高い標的結合が可能なRNAアプタマーの均一なプールを生成する[6]。
例[編集]
RNAアプタマーは、アンタゴニスト、アゴニスト、またはいわゆる「RNAデコイ・アプタマー」として作用するように設計することができる[77][92]。アンタゴニストの場合、RNAアプタマーは、特定のタンパク質がその細胞膜受容体に結合するのを阻止するため、またはタンパク質の標的に結合してそのタンパク質がその活性を発揮することを止ぐために使用される[77]。現在、臨床試験に進んでいるRNAアプタマーに基づく治療法は、アンタゴニストとして作用するもののみである[77]。RNAアプタマーがアゴニストとして作用するように設計されている場合には、免疫細胞の活性化を促進する共刺激分子として、生体防御システムの動員を助けることになる[77][93]。RNAデコイ・アプタマーの場合、合成RNAアプタマーはネイティブRNA分子に類似している[77][92]。そのため、ネイティブRNAターゲットに結合するタンパク質が、代わりにRNAアプタマーに結合し、特定の疾患の生体分子経路を阻害する可能性がある[77][92]。RNAアプタマーは、直接的な治療薬としての有用性に加えて、他の治療上の役割も検討されている。例えば、RNAアプタマーを薬物化合物に結合させることで、RNAアプタマーは薬物を標的とした送達システムとして機能することができる[6]。このようなRNAアプタマーはApDCとして知られている[6]。また、放射性同位元素や蛍光色素分子との共役により、画像診断に有用な場合もある[6][94][95]。
RNAアプタマーの選択にSELEXプロセスを利用しているため、多くの潜在的な標的に対してRNAアプタマーを生成することができる。SELEX中にRNAアプタマーを直接標的に導入することで、非常に選択性の高い、高親和性の均質なRNAアプタマーのプールを生成できる。このように、RNAアプタマーは、小さなペプチドやタンパク質だけでなく、細胞断片、細胞全体、さらには特定の組織を標的とするよう作成することができる[6][77][96][97][89][98]。RNAアプタマー分子標的および潜在的標的の例には、血管内皮増殖因子[99]、骨芽細胞[100]、およびC-X-Cケモカインリガンド12(CXCL12)がある[6][7][101]。
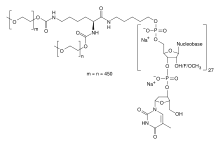
RNAアプタマー治療薬の一例として、FDAが承認した唯一のRNAアプタマー治療薬であるペガプタニブ(商品名マキュゲン®)がある[6][7][77]。2004年に、加齢黄斑変性症の治療薬として承認されたペガプタニブは、VEGFアンタゴニストとして作用する28塩基のRNAアプタマーである[6][7][77]。しかし、ベバシズマブやラニビズマブなどの抗体系治療薬ほどの効果はない[77][102][103]。RNAアプタマー治療薬のもう一つの例としては、慢性リンパ性白血病、膵臓がん、およびその他のがんで臨床試験が行われている45ヌクレオチドRNAアプタマーであるNOX-A12がある[7]。NOX-A12は、腫瘍の増殖に関与するケモカインであるCXCL12/SDF-1に対するアンタゴニストとして作用する[7]。
制限事項[編集]
RNAアプタマーは、高選択性で強固に結合することから医薬品としての利用が注目されているが、生体内での利用を妨げる多くの問題がある。一つには、RNAアプタマーは、体内に導入されてもそのままではヌクレアーゼによって数分で分解されてしまうことである[7][77][104][105]。また、RNAアプタマーはその大きさが小さいため、腎臓系によって血中から取り除かれてしまう[7][77][78][104][105]。RNAアプタマーは負電荷を帯びているため、血液中のタンパク質と結合し、非標的組織への送達と毒性を引き起こすことも知られている[77][106][107]。繰り返しシトシン-リン酸-グアニン(CpG)配列を含むアプタマーは、Toll様受容体経路を介して免疫系の活性化を引き起こすため、RNAアプタマーを単離する際にも注意が必要である[7][108][109]。
RNAアプタマーの生体内(in vivo)での制限のいくつかに対抗するために、ヌクレオチドに対してさまざまな修飾を加え、アプタマーの有効性を助けることができる。例えば、ポリエチレングリコール(PEG)部位を付加してアプタマーのサイズを大きくし、腎糸球体による血流からの除去を防ぐことができる[110][111]。ただし、PEGは生体内試験中のアレルギー反応に関与している[77][112][113]。さらに、ヌクレアーゼ分解を防ぐために、2'フルオロまたはアミノ基、3'転化チミジンなどの修飾を加えることができる[7][77][114][115]。さらに、リボース糖がD型ではなくL型になるようにアプタマーを合成することで、ヌクレアーゼの認識をさらに妨げることもできる[6][77][116][117]。このようなアプタマーは、シュピーゲルマー(Spiegelmer)として知られている[6][117]。Toll様受容体経路の活性化を防ぐためには、アプタマー内のシトシン核酸塩基をメチル化することができる[7]。しかしながら、生体内での有効性の低下に対するこれらの潜在的な解決策にもかかわらず、アプタマーを化学的に修飾すると、標的に対するその結合親和性を弱める可能性がある[77][118]。
関連項目[編集]
脚注[編集]
- ^ DeFrancesco L (March 2017). “The 'anti-hype' vaccine”. Nature Biotechnology 35 (3): 193–197. doi:10.1038/nbt.3812. PMID 28244993.
- ^ “Our COVID-19 Vaccine Study – What's Next? | Pfizer”. www.pfizer.com. 2020年11月29日閲覧。
- ^ a b Iida T, Nakayama J, Moazed D (July 2008). “siRNA-mediated heterochromatin establishment requires HP1 and is associated with antisense transcription”. Molecular Cell 31 (2): 178–89. doi:10.1016/j.molcel.2008.07.003. PMC 2575423. PMID 18657501.
- ^ a b c d e f g h i Nikam RR, Gore KR (August 2018). “Journey of siRNA: Clinical Developments and Targeted Delivery”. Nucleic Acid Therapeutics 28 (4): 209–224. doi:10.1089/nat.2017.0715. PMID 29584585.
- ^ a b c d e f Bartel DP (January 2009). “MicroRNAs: target recognition and regulatory functions”. Cell 136 (2): 215–33. doi:10.1016/j.cell.2009.01.002. PMC 3794896. PMID 19167326.
- ^ a b c d e f g h i j k l m n o p q r Zhu G, Chen X (September 2018). “Aptamer-based targeted therapy”. Advanced Drug Delivery Reviews 134: 65–78. doi:10.1016/j.addr.2018.08.005. PMC 6239901. PMID 30125604.
- ^ a b c d e f g h i j k l m n o p Kaur H, Bruno JG, Kumar A, Sharma TK (2018-07-01). “Aptamers in the Therapeutics and Diagnostics Pipelines”. Theranostics 8 (15): 4016–4032. doi:10.7150/thno.25958. PMC 6096388. PMID 30128033.
- ^ a b Dunn MR, Jimenez RM, Chaput JC (2017-10-04). “Analysis of aptamer discovery and technology” (英語). Nature Reviews Chemistry 1 (10): 1–16. doi:10.1038/s41570-017-0076. ISSN 2397-3358.
- ^ Coutinho MF, Matos L, Santos JI, Alves S (2019) (英語). The mRNA Metabolism in Human Disease. 1157. Cham: Springer International Publishing. 133–177. doi:10.1007/978-3-030-19966-1_7. ISBN 978-3-030-19965-4. PMID 31342441
- ^ “Messenger RNA (mRNA)”. Genome.gov. 2020年10月5日閲覧。
- ^ “mRNA” (英語). Biology Dictionary (2016年11月25日). 2020年11月30日閲覧。
- ^ Herndon MK, Quirk CC, Nilson JH (January 2016). “Chapter 2 - Control of Hormone Gene Expression”. Endocrinology: Adult and Pediatric (Seventh ed.). Philadelphia: W.B. Saunders. pp. 16–29.e2. doi:10.1016/B978-0-323-18907-1.00002-0. ISBN 978-0-323-18907-1
- ^ Yamamoto A, Kormann M, Rosenecker J, Rudolph C (March 2009). “Current prospects for mRNA gene delivery”. European Journal of Pharmaceutics and Biopharmaceutics 71 (3): 484–9. doi:10.1016/j.ejpb.2008.09.016. PMID 18948192.
- ^ Huang L, Zhang L, Li W, Li S, Wen J, Li H, Liu Z (July 2020). “Advances in Development of mRNA-Based Therapeutics”. Current Topics in Microbiology and Immunology (Berlin, Heidelberg: Springer): 1–20. doi:10.1007/82_2020_222. PMID 32683507.
- ^ Hershey AD (September 1953). “Nucleic acid economy in bacteria infected with bacteriophage T2”. The Journal of General Physiology 37 (1): 1–23. doi:10.1085/jgp.37.1.1. PMC 2147426. PMID 13084888.
- ^ Astrachan L, Colowick CP, Kaplan NO (April 1956). “Phosphorus incorporation in Escherichia coli ribonucleic acid after infection with bacteriophage T2”. Virology 2 (2): 149–161. doi:10.1016/0042-6822(56)90016-2. PMID 13312220.
- ^ Brenner S, Jacob F, Meselson M (May 1961). “An unstable intermediate carrying information from genes to ribosomes for protein synthesis”. Nature 190 (4776): 576–581. Bibcode: 1961Natur.190..576B. doi:10.1038/190576a0. PMID 20446365.
- ^ Gros F, Hiatt H, Gilbert W, Kurland CG, Risebrough RW, Watson JD (May 1961). “Unstable ribonucleic acid revealed by pulse labelling of Escherichia coli”. Nature 190 (4776): 581–5. Bibcode: 1961Natur.190..581G. doi:10.1038/190581a0. PMID 13708983.
- ^ Wolff JA, Malone RW, Williams P, Chong W, Acsadi G, Jani A, Felgner PL (March 1990). “Direct gene transfer into mouse muscle in vivo”. Science 247 (4949 Pt 1): 1465–8. Bibcode: 1990Sci...247.1465W. doi:10.1126/science.1690918. PMID 1690918.
- ^ a b Sahin U, Karikó K, Türeci Ö (October 2014). “mRNA-based therapeutics--developing a new class of drugs”. Nature Reviews. Drug Discovery 13 (10): 759–80. doi:10.1038/nrd4278. PMID 25233993.
- ^ Karikó K (April 2019). “In vitro-Transcribed mRNA Therapeutics: Out of the Shadows and Into the Spotlight”. Molecular Therapy 27 (4): 691–692. doi:10.1016/j.ymthe.2019.03.009. PMC 6453554. PMID 30905578.
- ^ Mandl CW, Aberle JH, Aberle SW, Holzmann H, Allison SL, Heinz FX (December 1998). “In vitro-synthesized infectious RNA as an attenuated live vaccine in a flavivirus model”. Nature Medicine 4 (12): 1438–40. doi:10.1038/4031. PMID 9846585.
- ^ Hoerr I, Obst R, Rammensee HG, Jung G (January 2000). “In vivo application of RNA leads to induction of specific cytotoxic T lymphocytes and antibodies”. European Journal of Immunology 30 (1): 1–7. doi:10.1002/1521-4141(200001)30:1<1::aid-immu1>3.0.co;2-#. PMID 10602021.
- ^ Van Hoecke L, Roose K (February 2019). “How mRNA therapeutics are entering the monoclonal antibody field”. Journal of Translational Medicine 17 (1): 54. doi:10.1186/s12967-019-1804-8. PMC 6387507. PMID 30795778.
- ^ a b Zhang C, Maruggi G, Shan H, Li J (2019). “Advances in mRNA Vaccines for Infectious Diseases” (英語). Frontiers in Immunology 10: 594. doi:10.3389/fimmu.2019.00594. PMC 6446947. PMID 30972078.
- ^ Hua Z, Hou B (March 2013). “TLR signaling in B-cell development and activation”. Cellular & Molecular Immunology 10 (2): 103–6. doi:10.1038/cmi.2012.61. PMC 4003046. PMID 23241902.
- ^ Kato H, Oh SW, Fujita T (May 2017). “RIG-I-Like Receptors and Type I Interferonopathies”. Journal of Interferon & Cytokine Research 37 (5): 207–213. doi:10.1089/jir.2016.0095. PMC 5439449. PMID 28475461.
- ^ Conry RM, LoBuglio AF, Wright M, Sumerel L, Pike MJ, Johanning F, Benjamin R, Lu D, Curiel DT (April 1995). “Characterization of a messenger RNA polynucleotide vaccine vector”. Cancer Research 55 (7): 1397–400. PMID 7882341.
- ^ Boczkowski D, Nair SK, Snyder D, Gilboa E (August 1996). “Dendritic cells pulsed with RNA are potent antigen-presenting cells in vitro and in vivo”. The Journal of Experimental Medicine 184 (2): 465–72. doi:10.1084/jem.184.2.465. PMC 2192710. PMID 8760800.
- ^ Nair SK, Heiser A, Boczkowski D, Majumdar A, Naoe M, Lebkowski JS, Vieweg J, Gilboa E (September 2000). “Induction of cytotoxic T cell responses and tumor immunity against unrelated tumors using telomerase reverse transcriptase RNA transfected dendritic cells”. Nature Medicine 6 (9): 1011–7. doi:10.1038/79519. PMID 10973321.
- ^ Argos Therapeutics (2018-06-13). An International Phase 3 Randomized Trial of Autologous Dendritic Cell Immunotherapy (AGS-003) Plus Standard Treatment of Advanced Renal Cell Carcinoma (ADAPT).
- ^ Kreiter S, Konrad T, Sester M, Huber C, Türeci O, Sahin U (October 2007). “Simultaneous ex vivo quantification of antigen-specific CD4+ and CD8+ T cell responses using in vitro transcribed RNA”. Cancer Immunology, Immunotherapy 56 (10): 1577–87. doi:10.1007/s00262-007-0302-7. PMID 17361438.
- ^ Kreiter S, Selmi A, Diken M, Sebastian M, Osterloh P, Schild H, Huber C, Türeci O, Sahin U (January 2008). “Increased antigen presentation efficiency by coupling antigens to MHC class I trafficking signals”. Journal of Immunology 180 (1): 309–18. doi:10.4049/jimmunol.180.1.309. PMID 18097032.
- ^ Kuhn AN, Diken M, Kreiter S, Selmi A, Kowalska J, Jemielity J, Darzynkiewicz E, Huber C, Türeci O, Sahin U (August 2010). “Phosphorothioate cap analogs increase stability and translational efficiency of RNA vaccines in immature dendritic cells and induce superior immune responses in vivo”. Gene Therapy 17 (8): 961–71. doi:10.1038/gt.2010.52. PMID 20410931.
- ^ BioNTech RNA Pharmaceuticals GmbH (2020-01-14). Clinical First-in-human Dose Escalation Study Evaluating the Safety and Tolerability of Intranodal Administration of an RNA-based Cancer Vaccine Targeting Two Tumor-associated Antigens in Patients With Advanced Melanoma.
- ^ Ligtenberg MA, Pico de Coaña Y, Shmushkovich T, Yoshimoto Y, Truxova I, Yang Y, Betancur-Boissel M, Eliseev AV, Wolfson AD, Kiessling R (June 2018). “Self-Delivering RNAi Targeting PD-1 Improves Tumor-Specific T Cell Functionality for Adoptive Cell Therapy of Malignant Melanoma”. Molecular Therapy 26 (6): 1482–1493. doi:10.1016/j.ymthe.2018.04.015. PMC 5986970. PMID 29735366.
- ^ Lian S, Xie R, Ye Y, Xie X, Li S, Lu Y, Li B, Cheng Y, Katanaev VL, Jia L (April 2019). “Simultaneous blocking of CD47 and PD-L1 increases innate and adaptive cancer immune responses and cytokine release”. EBioMedicine 42: 281–295. doi:10.1016/j.ebiom.2019.03.018. PMC 6491392. PMID 30878596.
- ^ “mRNA-4157 | Moderna, Inc.”. www.modernatx.com. 2020年11月30日閲覧。
- ^ Martinon F, Krishnan S, Lenzen G, Magné R, Gomard E, Guillet JG, Lévy JP, Meulien P (July 1993). “Induction of virus-specific cytotoxic T lymphocytes in vivo by liposome-entrapped mRNA”. European Journal of Immunology 23 (7): 1719–22. doi:10.1002/eji.1830230749. PMID 8325342.
- ^ Hekele A, Bertholet S, Archer J, Gibson DG, Palladino G, Brito LA, Otten GR, Brazzoli M, Buccato S, Bonci A, Casini D, Maione D, Qi ZQ, Gill JE, Caiazza NC, Urano J, Hubby B, Gao GF, Shu Y, De Gregorio E, Mandl CW, Mason PW, Settembre EC, Ulmer JB, Craig Venter J, Dormitzer PR, Rappuoli R, Geall AJ (August 2013). “Rapidly produced SAM(®) vaccine against H7N9 influenza is immunogenic in mice”. Emerging Microbes & Infections 2 (8): e52. doi:10.1038/emi.2013.54. PMC 3821287. PMID 26038486.
- ^ Petsch B, Schnee M, Vogel AB, Lange E, Hoffmann B, Voss D, Schlake T, Thess A, Kallen KJ, Stitz L, Kramps T (December 2012). “Protective efficacy of in vitro synthesized, specific mRNA vaccines against influenza A virus infection”. Nature Biotechnology 30 (12): 1210–6. doi:10.1038/nbt.2436. PMID 23159882.
- ^ Routy JP, Boulassel MR, Yassine-Diab B, Nicolette C, Healey D, Jain R, Landry C, Yegorov O, Tcherepanova I, Monesmith T, Finke L, Sékaly RP (February 2010). “Immunologic activity and safety of autologous HIV RNA-electroporated dendritic cells in HIV-1 infected patients receiving antiretroviral therapy”. Clinical Immunology 134 (2): 140–7. doi:10.1016/j.clim.2009.09.009. PMC 2818410. PMID 19889582.
- ^ Van Gulck E, Vlieghe E, Vekemans M, Van Tendeloo VF, Van De Velde A, Smits E, Anguille S, Cools N, Goossens H, Mertens L, De Haes W, Wong J, Florence E, Vanham G, Berneman ZN (February 2012). “mRNA-based dendritic cell vaccination induces potent antiviral T-cell responses in HIV-1-infected patients”. Aids 26 (4): F1-12. doi:10.1097/qad.0b013e32834f33e8. PMID 22156965.
- ^ Helmy YA, Fawzy M, Elaswad A, Sobieh A, Kenney SP, Shehata AA (April 2020). “The COVID-19 Pandemic: A Comprehensive Review of Taxonomy, Genetics, Epidemiology, Diagnosis, Treatment, and Control”. Journal of Clinical Medicine 9 (4): 1225. doi:10.3390/jcm9041225. PMC 7230578. PMID 32344679.
- ^ “Drug”. Default. 2020年11月30日閲覧。
- ^ Jackson LA, Anderson EJ, Rouphael NG, Roberts PC, Makhene M, Coler RN, McCullough MP, Chappell JD, Denison MR, Stevens LJ, Pruijssers AJ, McDermott A, Flach B, Doria-Rose NA, Corbett KS, Morabito KM, O'Dell S, Schmidt SD, Swanson PA, Padilla M, Mascola JR, Neuzil KM, Bennett H, Sun W, Peters E, Makowski M, Albert J, Cross K, Buchanan W, Pikaart-Tautges R, Ledgerwood JE, Graham BS, Beigel JH (November 2020). “An mRNA Vaccine against SARS-CoV-2 - Preliminary Report”. The New England Journal of Medicine 383 (20): 1920–1931. doi:10.1056/NEJMoa2022483. PMC 7377258. PMID 32663912.
- ^ “Moderna's Work on a COVID-19 Vaccine Candidate | Moderna, Inc.”. www.modernatx.com. 2020年11月30日閲覧。
- ^ “US CDC panel recommends Moderna's Covid-19 vaccine for use in adults”. www.pharmaceutical-technology.com. 2020年12月21日閲覧。
- ^ a b Houseley J, Tollervey D (February 2009). “The many pathways of RNA degradation”. Cell 136 (4): 763–76. doi:10.1016/j.cell.2009.01.019. PMID 19239894.
- ^ Kowalski PS, Rudra A, Miao L, Anderson DG (April 2019). “Delivering the Messenger: Advances in Technologies for Therapeutic mRNA Delivery”. Molecular Therapy 27 (4): 710–728. doi:10.1016/j.ymthe.2019.02.012. PMC 6453548. PMID 30846391.
- ^ Sharova LV, Sharov AA, Nedorezov T, Piao Y, Shaik N, Ko MS (February 2009). “Database for mRNA half-life of 19 977 genes obtained by DNA microarray analysis of pluripotent and differentiating mouse embryonic stem cells”. DNA Research 16 (1): 45–58. doi:10.1093/dnares/dsn030. PMC 2644350. PMID 19001483.
- ^ Deering RP, Kommareddy S, Ulmer JB, Brito LA, Geall AJ (June 2014). “Nucleic acid vaccines: prospects for non-viral delivery of mRNA vaccines”. Expert Opinion on Drug Delivery 11 (6): 885–99. doi:10.1517/17425247.2014.901308. PMID 24665982.
- ^ Golombek S, Pilz M, Steinle H, Kochba E, Levin Y, Lunter D, Schlensak C, Wendel HP, Avci-Adali M (June 2018). “Intradermal Delivery of Synthetic mRNA Using Hollow Microneedles for Efficient and Rapid Production of Exogenous Proteins in Skin”. Molecular Therapy. Nucleic Acids 11: 382–392. doi:10.1016/j.omtn.2018.03.005. PMC 5992458. PMID 29858073.
- ^ “7.19A: RNA Regulation and Antisense RNA” (英語). Biology LibreTexts (2017年6月6日). 2020年12月1日閲覧。
- ^ Singh SB, Phillips JW, Wang J (March 2007). “Highly sensitive target-based whole-cell antibacterial discovery strategy by antisense RNA silencing”. Current Opinion in Drug Discovery & Development 10 (2): 160–6. PMID 17436551.
- ^ US 2017283805, Bonci D, De Maria R, "Antisense RNA for Treating Cancer and Inhibition of Metastasis and Vectors for Antisense Sequestration", published 2017, assigned to Istituto Superiore di Sanità
- ^ Wade JT (2013). “Antisense RNA”. Brenner's Encyclopedia of Genetics. doi:10.1016/B978-0-12-809633-8.06068-4. ISBN 978-0-12-809633-8
- ^ Xu JZ, Zhang JL, Zhang WG (2018). “Antisense RNA: the new favorite in genetic research”. Journal of Zhejiang University. Science. B 19 (10): 739–749. doi:10.1631/jzus.B1700594. PMC 6194357. PMID 30269442.
- ^ Farooqi AA, Rehman ZU, Muntane J (November 2014). “Antisense therapeutics in oncology: current status”. OncoTargets and Therapy 7: 2035–42. doi:10.2147/ott.s49652. PMC 4224095. PMID 25395862.
- ^ “Antisense Oligonucleotide-based Therapeutics”, Gene Therapy (CRC Press): pp. 365–392, (May 2000), ISBN 978-0-429-13193-6
- ^ Annual Review of Pharmacology and Toxicology 0. (2007-01-10). doi:10.1146/pharmtox.2009.49.issue-1.
- ^ a b c d e f g h Hanna J, Hossain GS, Kocerha J (2019). “The Potential for microRNA Therapeutics and Clinical Research” (英語). Frontiers in Genetics 10: 478. doi:10.3389/fgene.2019.00478. PMC 6532434. PMID 31156715.
- ^ a b c d e f g h i j k Devi GR (September 2006). “siRNA-based approaches in cancer therapy”. Cancer Gene Therapy 13 (9): 819–29. doi:10.1038/sj.cgt.7700931. PMID 16424918.
- ^ Napoli C, Lemieux C, Jorgensen R (April 1990). “Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans”. The Plant Cell 2 (4): 279–289. doi:10.1105/tpc.2.4.279. PMC 159885. PMID 12354959.
- ^ Hannon GJ (July 2002). “RNA interference”. Nature 418 (6894): 244–51. Bibcode: 2002Natur.418..244H. doi:10.1038/418244a. PMID 12110901.
- ^ a b Tomari Y, Zamore PD (March 2005). “Perspective: machines for RNAi”. Genes & Development 19 (5): 517–29. doi:10.1101/gad.1284105. PMID 15741316.
- ^ a b Banan M, Puri N (October 2004). “The ins and outs of RNAi in mammalian cells”. Current Pharmaceutical Biotechnology 5 (5): 441–50. doi:10.2174/1389201043376643. PMID 15544492.
- ^ Song E, Lee SK, Dykxhoorn DM, Novina C, Zhang D, Crawford K, Cerny J, Sharp PA, Lieberman J, Manjunath N, Shankar P (July 2003). “Sustained small interfering RNA-mediated human immunodeficiency virus type 1 inhibition in primary macrophages”. Journal of Virology 77 (13): 7174–81. doi:10.1128/JVI.77.13.7174-7181.2003. PMC 164789. PMID 12805416.
- ^ Song E, Lee SK, Wang J, Ince N, Ouyang N, Min J, Chen J, Shankar P, Lieberman J (March 2003). “RNA interference targeting Fas protects mice from fulminant hepatitis”. Nature Medicine 9 (3): 347–51. doi:10.1038/nm828. PMID 12579197.
- ^ Martynov A, Didenko G, Farber B, Farber S, Cruts O (2018). “The anticancer activity of antisense micro RNA (fRNA) in combination with the lectin from Bacillus subtilis B‐7025”. Journal of Pharmacy and Pharmacology 70 (6): 732–39. doi:10.1111/jphp.12898. PMID 29520790.
- ^ Lai EC (April 2002). “Micro RNAs are complementary to 3' UTR sequence motifs that mediate negative post-transcriptional regulation”. Nature Genetics 30 (4): 363–4. doi:10.1038/ng865. PMID 11896390.
- ^ Jackson AL, Bartz SR, Schelter J, Kobayashi SV, Burchard J, Mao M, Li B, Cavet G, Linsley PS (June 2003). “Expression profiling reveals off-target gene regulation by RNAi”. Nature Biotechnology 21 (6): 635–7. doi:10.1038/nbt831. PMID 12754523.
- ^ a b c d e Bader AG, Brown D, Winkler M (September 2010). “The promise of microRNA replacement therapy”. Cancer Research 70 (18): 7027–30. doi:10.1158/0008-5472.CAN-10-2010. PMC 2940943. PMID 20807816.
- ^ a b Wiggins JF, Ruffino L, Kelnar K, Omotola M, Patrawala L, Brown D, Bader AG (July 2010). “Development of a lung cancer therapeutic based on the tumor suppressor microRNA-34”. Cancer Research 70 (14): 5923–30. doi:10.1158/0008-5472.CAN-10-0655. PMC 2913706. PMID 20570894.
- ^ Kota J, Chivukula RR, O'Donnell KA, Wentzel EA, Montgomery CL, Hwang HW, Chang TC, Vivekanandan P, Torbenson M, Clark KR, Mendell JR, Mendell JT (June 2009). “Therapeutic microRNA delivery suppresses tumorigenesis in a murine liver cancer model”. Cell 137 (6): 1005–17. doi:10.1016/j.cell.2009.04.021. PMC 2722880. PMID 19524505.
- ^ a b Huang H, Suslov NB, Li NS, Shelke SA, Evans ME, Koldobskaya Y, Rice PA, Piccirilli JA (August 2014). “A G-quadruplex-containing RNA activates fluorescence in a GFP-like fluorophore”. Nature Chemical Biology 10 (8): 686–91. doi:10.1038/nchembio.1561. PMC 4104137. PMID 24952597.
- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z aa ab ac Zhou J, Rossi J (March 2017). “Aptamers as targeted therapeutics: current potential and challenges”. Nature Reviews. Drug Discovery 16 (3): 181–202. doi:10.1038/nrd.2016.199. PMC 5700751. PMID 27807347.
- ^ a b Guo P (December 2010). “The emerging field of RNA nanotechnology”. Nature Nanotechnology 5 (12): 833–42. Bibcode: 2010NatNa...5..833G. doi:10.1038/nnano.2010.231. PMC 3149862. PMID 21102465.
- ^ Gelinas AD, Davies DR, Janjic N (February 2016). “Embracing proteins: structural themes in aptamer-protein complexes”. Current Opinion in Structural Biology 36: 122–32. doi:10.1016/j.sbi.2016.01.009. PMID 26919170.
- ^ Geiger A, Burgstaller P, von der Eltz H, Roeder A, Famulok M (March 1996). “RNA aptamers that bind L-arginine with sub-micromolar dissociation constants and high enantioselectivity”. Nucleic Acids Research 24 (6): 1029–36. doi:10.1093/nar/24.6.1029. PMC 145747. PMID 8604334.
- ^ Chen L, Rashid F, Shah A, Awan HM, Wu M, Liu A, Wang J, Zhu T, Luo Z, Shan G (August 2015). “The isolation of an RNA aptamer targeting to p53 protein with single amino acid mutation”. Proceedings of the National Academy of Sciences of the United States of America 112 (32): 10002–7. Bibcode: 2015PNAS..11210002C. doi:10.1073/pnas.1502159112. PMC 4538674. PMID 26216949.
- ^ Keefe AD, Pai S, Ellington A (July 2010). “Aptamers as therapeutics”. Nature Reviews. Drug Discovery 9 (7): 537–50. doi:10.1038/nrd3141. PMC 7097324. PMID 20592747.
- ^ Shu Y, Pi F, Sharma A, Rajabi M, Haque F, Shu D, Leggas M, Evers BM, Guo P (February 2014). “Stable RNA nanoparticles as potential new generation drugs for cancer therapy”. Advanced Drug Delivery Reviews 66: 74–89. doi:10.1016/j.addr.2013.11.006. PMC 3955949. PMID 24270010.
- ^ a b Tuerk C, Gold L (August 1990). “Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase”. Science 249 (4968): 505–10. Bibcode: 1990Sci...249..505T. doi:10.1126/science.2200121. PMID 2200121.
- ^ a b c Ellington AD, Szostak JW (August 1990). “In vitro selection of RNA molecules that bind specific ligands”. Nature 346 (6287): 818–22. Bibcode: 1990Natur.346..818E. doi:10.1038/346818a0. PMID 1697402.
- ^ Guo KT, Ziemer G, Paul A, Wendel HP (April 2008). “CELL-SELEX: Novel perspectives of aptamer-based therapeutics”. International Journal of Molecular Sciences 9 (4): 668–78. doi:10.3390/ijms9040668. PMC 2635693. PMID 19325777.
- ^ Ohuchi S (2012). “Cell-SELEX Technology”. BioResearch Open Access 1 (6): 265–272. doi:10.1089/biores.2012.0253. PMC 3559206. PMID 23515081.
- ^ Cerchia L, Giangrande PH, McNamara JO, de Franciscis V (2009). “Cell-specific aptamers for targeted therapies”. Nucleic Acid and Peptide Aptamers. Methods in Molecular Biology. 535. Totowa, NJ: Humana Press. pp. 59–78. doi:10.1007/978-1-59745-557-2_5. ISBN 978-1-59745-557-2. PMC 4443708. PMID 19377980
- ^ a b Tang Z, Shangguan D, Wang K, Shi H, Sefah K, Mallikratchy P, Chen HW, Li Y, Tan W (July 2007). “Selection of aptamers for molecular recognition and characterization of cancer cells”. Analytical Chemistry 79 (13): 4900–7. doi:10.1021/ac070189y. PMID 17530817.
- ^ Xiao Z, Shangguan D, Cao Z, Fang X, Tan W (2008-02-18). “Cell-specific internalization study of an aptamer from whole cell selection”. Chemistry 14 (6): 1769–75. doi:10.1002/chem.200701330. PMID 18092308.
- ^ Phillips JA, Lopez-Colon D, Zhu Z, Xu Y, Tan W (July 2008). “Applications of aptamers in cancer cell biology”. Analytica Chimica Acta 621 (2): 101–8. doi:10.1016/j.aca.2008.05.031. PMID 18573375.
- ^ a b c Kohn DB, Bauer G, Rice CR, Rothschild JC, Carbonaro DA, Valdez P, Hao QL, Zhou C, Bahner I, Kearns K, Brody K, Fox S, Haden E, Wilson K, Salata C, Dolan C, Wetter C, Aguilar-Cordova E, Church J (July 1999). “A clinical trial of retroviral-mediated transfer of a rev-responsive element decoy gene into CD34(+) cells from the bone marrow of human immunodeficiency virus-1-infected children”. Blood 94 (1): 368–71. doi:10.1182/blood.V94.1.368.413a47_368_371. PMID 10381536.
- ^ Khedri M, Rafatpanah H, Abnous K, Ramezani P, Ramezani M (December 2015). “Cancer immunotherapy via nucleic acid aptamers”. International Immunopharmacology 29 (2): 926–936. doi:10.1016/j.intimp.2015.10.013. PMID 26603636.
- ^ Shi H, He X, Wang K, Wu X, Ye X, Guo Q, Tan W, Qing Z, Yang X, Zhou B (March 2011). “Activatable aptamer probe for contrast-enhanced in vivo cancer imaging based on cell membrane protein-triggered conformation alteration”. Proceedings of the National Academy of Sciences of the United States of America 108 (10): 3900–5. Bibcode: 2011PNAS..108.3900S. doi:10.1073/pnas.1016197108. PMC 3054025. PMID 21368158.
- ^ Zhang J, Smaga LP, Satyavolu NS, Chan J, Lu Y (December 2017). “DNA Aptamer-Based Activatable Probes for Photoacoustic Imaging in Living Mice”. Journal of the American Chemical Society 139 (48): 17225–17228. doi:10.1021/jacs.7b07913. PMC 5724028. PMID 29028325.
- ^ Mi J, Liu Y, Rabbani ZN, Yang Z, Urban JH, Sullenger BA, Clary BM (January 2010). “In vivo selection of tumor-targeting RNA motifs”. Nature Chemical Biology 6 (1): 22–4. doi:10.1038/nchembio.277. PMC 2795795. PMID 19946274.
- ^ Shangguan D, Li Y, Tang Z, Cao ZC, Chen HW, Mallikaratchy P, Sefah K, Yang CJ, Tan W (August 2006). “Aptamers evolved from live cells as effective molecular probes for cancer study”. Proceedings of the National Academy of Sciences of the United States of America 103 (32): 11838–43. doi:10.1073/pnas.0602615103. PMC 1567664. PMID 16873550.
- ^ Chen HW, Medley CD, Sefah K, Shangguan D, Tang Z, Meng L, Smith JE, Tan W (June 2008). “Molecular recognition of small-cell lung cancer cells using aptamers”. ChemMedChem 3 (6): 991–1001. doi:10.1002/cmdc.200800030. PMC 3544301. PMID 18338423.
- ^ Gragoudas ES, Adamis AP, Cunningham ET, Feinsod M, Guyer DR (December 2004). “Pegaptanib for neovascular age-related macular degeneration”. The New England Journal of Medicine 351 (27): 2805–16. doi:10.1056/NEJMoa042760. PMID 15625332.
- ^ Liang C, Guo B, Wu H, Shao N, Li D, Liu J, Dang L, Wang C, Li H, Li S, Lau WK, Cao Y, Yang Z, Lu C, He X, Au DW, Pan X, Zhang BT, Lu C, Zhang H, Yue K, Qian A, Shang P, Xu J, Xiao L, Bian Z, Tan W, Liang Z, He F, Zhang L, Lu A, Zhang G (March 2015). “Aptamer-functionalized lipid nanoparticles targeting osteoblasts as a novel RNA interference-based bone anabolic strategy”. Nature Medicine 21 (3): 288–94. doi:10.1038/nm.3791. PMC 5508976. PMID 25665179.
- ^ Hoellenriegel J, Zboralski D, Maasch C, Rosin NY, Wierda WG, Keating MJ, Kruschinski A, Burger JA (February 2014). “The Spiegelmer NOX-A12, a novel CXCL12 inhibitor, interferes with chronic lymphocytic leukemia cell motility and causes chemosensitization”. Blood 123 (7): 1032–9. doi:10.1182/blood-2013-03-493924. PMC 4123413. PMID 24277076.
- ^ Mousa SA, Mousa SS (June 2010). “Current status of vascular endothelial growth factor inhibition in age-related macular degeneration”. BioDrugs 24 (3): 183–94. doi:10.2165/11318550-000000000-00000. PMID 20210371.
- ^ Ferrara N, Adamis AP (June 2016). “Ten years of anti-vascular endothelial growth factor therapy”. Nature Reviews. Drug Discovery 15 (6): 385–403. doi:10.1038/nrd.2015.17. PMID 26775688.
- ^ a b Healy JM, Lewis SD, Kurz M, Boomer RM, Thompson KM, Wilson C, McCauley TG (December 2004). “Pharmacokinetics and biodistribution of novel aptamer compositions”. Pharmaceutical Research 21 (12): 2234–46. doi:10.1007/s11095-004-7676-4. PMID 15648255.
- ^ a b Abeydeera ND, Egli M, Cox N, Mercier K, Conde JN, Pallan PS, Mizurini DM, Sierant M, Hibti FE, Hassell T, Wang T, Liu FW, Liu HM, Martinez C, Sood AK, Lybrand TP, Frydman C, Monteiro RQ, Gomer RH, Nawrot B, Yang X (September 2016). “Evoking picomolar binding in RNA by a single phosphorodithioate linkage”. Nucleic Acids Research 44 (17): 8052–64. doi:10.1093/nar/gkw725. PMC 5041495. PMID 27566147.
- ^ Henry SP, Giclas PC, Leeds J, Pangburn M, Auletta C, Levin AA, Kornbrust DJ (May 1997). “Activation of the alternative pathway of complement by a phosphorothioate oligonucleotide: potential mechanism of action”. The Journal of Pharmacology and Experimental Therapeutics 281 (2): 810–6. PMID 9152389.
- ^ Farman CA, Kornbrust DJ (2016-11-17). “Oligodeoxynucleotide studies in primates: antisense and immune stimulatory indications”. Toxicologic Pathology 31 Suppl: 119–22. doi:10.1080/01926230390174995. PMID 12597439.
- ^ Avci-Adali M, Steinle H, Michel T, Schlensak C, Wendel HP (2013-07-23). “Potential capacity of aptamers to trigger immune activation in human blood”. PLOS ONE 8 (7): e68810. Bibcode: 2013PLoSO...868810A. doi:10.1371/journal.pone.0068810. PMC 3720859. PMID 23935890.
- ^ Mena A, Nichani AK, Popowych Y, Godson DL, Dent D, Townsend HG, Mutwiri GK, Hecker R, Babiuk LA, Griebel P (October 2003). “Innate immune responses induced by CpG oligodeoxyribonucleotide stimulation of ovine blood mononuclear cells”. Immunology 110 (2): 250–7. doi:10.1046/j.1365-2567.2003.01722.x. PMC 1783041. PMID 14511239.
- ^ Musumeci D, Montesarchio D (November 2012). “Polyvalent nucleic acid aptamers and modulation of their activity: a focus on the thrombin binding aptamer”. Pharmacology & Therapeutics 136 (2): 202–15. doi:10.1016/j.pharmthera.2012.07.011. PMID 22850531.
- ^ Soule EE, Bompiani KM, Woodruff RS, Sullenger BA (February 2016). “Targeting Two Coagulation Cascade Proteases with a Bivalent Aptamer Yields a Potent and Antidote-Controllable Anticoagulant”. Nucleic Acid Therapeutics 26 (1): 1–9. doi:10.1089/nat.2015.0565. PMC 4753633. PMID 26584417.
- ^ Ganson NJ, Povsic TJ, Sullenger BA, Alexander JH, Zelenkofske SL, Sailstad JM, Rusconi CP, Hershfield MS (May 2016). “Pre-existing anti-polyethylene glycol antibody linked to first-exposure allergic reactions to pegnivacogin, a PEGylated RNA aptamer”. The Journal of Allergy and Clinical Immunology 137 (5): 1610–1613.e7. doi:10.1016/j.jaci.2015.10.034. PMC 5819876. PMID 26688515.
- ^ Lincoff AM, Mehran R, Povsic TJ, Zelenkofske SL, Huang Z, Armstrong PW, Steg PG, Bode C, Cohen MG, Buller C, Laanmets P, Valgimigli M, Marandi T, Fridrich V, Cantor WJ, Merkely B, Lopez-Sendon J, Cornel JH, Kasprzak JD, Aschermann M, Guetta V, Morais J, Sinnaeve PR, Huber K, Stables R, Sellers MA, Borgman M, Glenn L, Levinson AI, Lopes RD, Hasselblad V, Becker RC, Alexander JH (January 2016). “Effect of the REG1 anticoagulation system versus bivalirudin on outcomes after percutaneous coronary intervention (REGULATE-PCI): a randomised clinical trial”. Lancet 387 (10016): 349–356. doi:10.1016/s0140-6736(15)00515-2. PMID 26547100.
- ^ Sharma TK, Bruno JG, Dhiman A (2017-03-01). “ABCs of DNA aptamer and related assay development”. Biotechnology Advances 35 (2): 275–301. doi:10.1016/j.biotechadv.2017.01.003. PMID 28108354.
- ^ Pieken WA, Olsen DB, Benseler F, Aurup H, Eckstein F (July 1991). “Kinetic characterization of ribonuclease-resistant 2'-modified hammerhead ribozymes”. Science 253 (5017): 314–7. Bibcode: 1991Sci...253..314P. doi:10.1126/science.1857967. PMID 1857967.
- ^ Vater A, Klussmann S (March 2003). “Toward third-generation aptamers: Spiegelmers and their therapeutic prospects”. Current Opinion in Drug Discovery & Development 6 (2): 253–61. PMID 12669461.
- ^ a b Rusconi CP, Roberts JD, Pitoc GA, Nimjee SM, White RR, Quick G, Scardino E, Fay WP, Sullenger BA (November 2004). “Antidote-mediated control of an anticoagulant aptamer in vivo”. Nature Biotechnology 22 (11): 1423–8. doi:10.1038/nbt1023. PMID 15502817.
- ^ Aaldering LJ, Tayeb H, Krishnan S, Fletcher S, Wilton SD, Veedu RN (2015-04-03). “Smart functional nucleic acid chimeras: enabling tissue specific RNA targeting therapy”. RNA Biology 12 (4): 412–25. doi:10.1080/15476286.2015.1017234. PMC 4615226. PMID 25849197.
外部リンク[編集]
- RNA therapeutics on the rise, Nature (April 2020}
