3C様プロテアーゼ
| SARSコロナウイルスメインプロテアーゼ | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| 識別子 | |||||||||
| EC番号 | 3.4.22.69 | ||||||||
| データベース | |||||||||
| IntEnz | IntEnz view | ||||||||
| BRENDA | BRENDA entry | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | KEGG entry | ||||||||
| MetaCyc | metabolic pathway | ||||||||
| PRIAM | profile | ||||||||
| PDB構造 | RCSB PDB PDBj PDBe PDBsum | ||||||||
| |||||||||
| ペプチダーゼC30, コロナウイルスエンドペプチダーゼ | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 識別子 | |||||||||
| 略号 | Peptidase_C30 | ||||||||
| Pfam | PF05409 | ||||||||
| InterPro | IPR008740 | ||||||||
| PROSITE | PS51442 | ||||||||
| MEROPS | C30 | ||||||||
| SCOP | d1q2wb1 | ||||||||
| SUPERFAMILY | d1q2wb1 | ||||||||
| |||||||||
3C様プロテアーゼ(3シーようプロテアーゼ、英: 3C-like protease、3CLpro)またはMproは、正式にはC30エンドペプチダーゼとして知られており、コロナウイルスに見られる主要なプロテアーゼである。コロナウイルスのポリタンパク質を11個の保存部位で切断する。システインプロテアーゼであり、プロテアーゼのPAクランのメンバーである。活性部位にシステイン-ヒスチジン触媒二残基を持ち、Gln-(Ser/Ala/Gly) ペプチド結合を切断する。
国際生化学・分子生物学連合 酵素委員会(EC)は、このファミリーをSARSコロナウイルスメインプロテアーゼ(Mpro; EC 3.4.22.69)と呼んでいる。3C様プロテアーゼは、コロナウイルス非構造タンパク質5(nsp5)に相当する。慣用名「3C」は、ピコルナウイルスに見られる相同プロテアーゼである3Cプロテアーゼ(3Cpro)を意味する。
機能
[編集]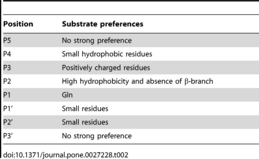
3C様プロテアーゼは、P1位のグルタミンとP1'位の小分子アミノ酸(セリン、アラニン、グリシン)の間のペプチド結合を触媒的に切断することができる(右図を参照)。SARSコロナウイルス3CLproは、たとえば以下のようにペプチドを自己切断することができる[1][2][3]。
このプロテアーゼは、コロナウイルスレプリカーゼポリタンパク質(UniPlot P0C6U8)のプロセシングに重要である。これは、コロナウイルスの主要なプロテアーゼであり、非構造タンパク質5(nsp5)に相当する[4]。これは、コロナウイルスのポリタンパク質を11個の保存部位で切断する。3C様プロテアーゼは、その活性部位にシステイン-ヒスチジン触媒二残基を持っている[2]。システインの硫黄は求核剤として働き、ヒスチジンのイミダゾール環は一般塩基として働く[5]。
命名法
[編集]酵素委員会(EC)が提供する代替名として、3CLpro、3C様プロテアーゼ、コロナウイルス3C様プロテアーゼ、Mpro、SARS 3C様プロテアーゼ、SARSコロナウイルス3CLプロテアーゼ、SARSコロナウイルスメインペプチダーゼ、SARSコロナウイルスメインプロテアーゼ、SARS-CoV 3CLpro酵素、SARS-CoVメインプロテアーゼ、SARS-CoV Mpro、重症急性呼吸器症候群コロナウイルスメインプロテアーゼがある。
英語版:
Alternative names provided by the EC include 3CLpro, 3C-like protease, coronavirus 3C-like protease, Mpro, SARS 3C-like protease, SARS coronavirus 3CL protease, SARS coronavirus main peptidase, SARS coronavirus main protease, SARS-CoV 3CLpro enzyme, SARS-CoV main protease, SARS-CoV Mpro and severe acute respiratory syndrome coronavirus main protease.
治療標的として
[編集]

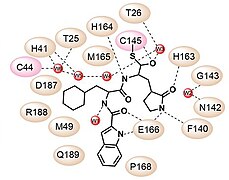
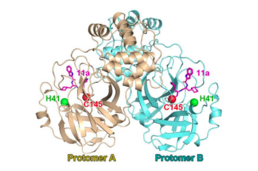
プロテアーゼ3CLproは、ウイルスRNAから翻訳されたポリタンパク質をプロセシングするのに不可欠な役割を果たしているため、コロナウイルス感染症の潜在的な創薬ターゲットである。リガンド非結合SARS-CoV-2プロテアーゼ3CLproと、α-ケトアミド阻害剤との複合体のX線構造は、SARS-CoV-2感染症の治療のためのα-ケトアミド阻害剤[6]の設計の基礎となる[7][8][9][10][11]。3CLproおよび相同3Cproに対して開発されているプロテアーゼ阻害剤には、CLpro-1、GC376、ルピントリビル(rupintrivir)、PF-07304814、PF-07321332、化学物質11a(chemical 11a)、化学物質11b(chemical 11b)がある[12][13][14][15]。2020年9月、静脈内投与のプロドラッグ PF-07304814 が臨床試験に入り[16]、2021年2月、経口投与のフォローアップ薬PF-07321332が被験者の募集を開始した。長期分子動力学シミュレーション(1.50 µs)の研究では、ウーロンホモビスフラバンA(茶の生理活性物質)が、これまでに提案されていた再開発の抗HIV薬よりも強力なSARS-CoV-2のMpro阻害剤として報告された[17]。報告では、堅牢な計算戦略によって、研究室内で合成されたアクリジンジオンアナログDSPD-2およびDSPD-6は、抗ウイルス剤(サキナビル、saquinavir)よりも有利なMM-PBSA相互作用エネルギーを示し、Mproの結合ポケット内の奥深くに収まったと述べている。これらのアクリジンジオンアナログは、許容できるADMET値と低毒性プロファイルを持っている。SARS-CoV-2 Mproの結合部位への分子の結合能は、結合ポケットのS1サブサイトの残基とより効率的に相互作用するように分子を標的化することで高めることができる[18]。

-
SARS-CoV-2の3CLpro阻害剤の設計戦略と、化学構造11aおよび11b。
-
3CLpro阻害剤と化学物質11a
-
3CLpro阻害剤と化学物質11b
他の3C(様)プロテアーゼ
[編集]3C様プロテアーゼ(3C(L)pro)は、(+)ssRNAウイルスに広く見られる。これらはすべて、キモトリプシン様フォールド(PAクラン)を持つシステインプロテアーゼで、触媒二残基または触媒三残基を使用する。これらは、基質特異性と阻害剤効果について、いくつかの一般的な類似点がある。これらは配列類似性によってサブファミリーに分けられ、それらが見られるウイルスのファミリーに対応づけられる[19]。
- コロナウイルスは、3CLproを持っている。
- ピコルナウイルス科は、ピコルナウイルス3Cpro(EC 3.4.22.28; InterPro: IPR000199; MEROPS C03)を持っている。これは、もっとも早く研究されたファミリーである。ポリオウイルスやライノウイルスに見られるものがその例である(どちらもエンテロウイルス属のメンバー)。
- カリシウイルス科は、3CLpro(InterPro: IPR001665; MEROPS C37)を持っている。例としては、ノーウォークウイルスに見られるものがある。
その他のメンバーは、ポティウイルス科および非コロナウイルス科のニドウイルス目から知られている[20]。
参照項目
[編集]- 3CLpro-1- 3C様プロテアーゼ阻害剤として作用するルピントリビルに関する抗ウイルス薬
- カルモフール - 抗悪性腫瘍薬として用いられるピリミジン系アナログ
- エブセレン- 抗炎症作用、抗酸化作用、細胞保護作用を持つ有機セレン系の合成薬剤分子
- GC376- ヒトおよび動物の治療用に開発中の広域スペクトル抗ウイルス薬
- ルピントリビル- 3Cおよび3C様プロテアーゼ阻害剤として作用するペプチド模倣型の抗ウイルス薬
- テアフラビンジガレート- 紅茶に含まれる抗酸化作用のある天然フェノールであるテアフラビン誘導体
脚注
[編集]- ^ Goetz DH, Choe Y, Hansell E, Chen YT, McDowell M, Jonsson CB, Roush WR, McKerrow J, Craik CS (July 2007). “Substrate specificity profiling and identification of a new class of inhibitor for the major protease of the SARS coronavirus”. Biochemistry 46 (30): 8744–52. doi:10.1021/bi0621415. PMID 17605471.
- ^ a b Fan K, Wei P, Feng Q, Chen S, Huang C, Ma L, Lai B, Pei J, Liu Y, Chen J, Lai L (January 2004). “Biosynthesis, purification, and substrate specificity of severe acute respiratory syndrome coronavirus 3C-like proteinase”. The Journal of Biological Chemistry 279 (3): 1637–42. doi:10.1074/jbc.m310875200. PMC 7980035. PMID 14561748.
- ^ Akaji K, Konno H, Onozuka M, Makino A, Saito H, Nosaka K (November 2008). “Evaluation of peptide-aldehyde inhibitors using R188I mutant of SARS 3CL protease as a proteolysis-resistant mutant”. Bioorganic & Medicinal Chemistry 16 (21): 9400–8. doi:10.1016/j.bmc.2008.09.057. PMC 7126698. PMID 18845442.
- ^ Fehr AR, Perlman S (2015). “Coronaviruses: an overview of their replication and pathogenesis”. Coronaviruses. Methods in Molecular Biology. 1282. Springer. pp. 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385. PMID 25720466. "See section: Virion Structure."
- ^ Ryu, Young Bae; Park, Su-Jin; Kim, Young Min; Lee, Ju-Yeon; Seo, Woo Duck; Chang, Jong Sun; Park, Ki Hun; Rho, Mun-Chual et al. (2010-03-15). “SARS-CoV 3CLpro inhibitory effects of quinone-methide triterpenes from Tripterygium regelii” (英語). Bioorganic & Medicinal Chemistry Letters 20 (6): 1873–1876. doi:10.1016/j.bmcl.2010.01.152. ISSN 0960-894X. PMC 7127101. PMID 20167482.
- ^ Ocain TD, Rich DH (February 1992). “alpha-Keto amide inhibitors of aminopeptidases”. Journal of Medicinal Chemistry 35 (3): 451–6. doi:10.1021/jm00081a005. PMID 1738140.
- ^ Anand K, Ziebuhr J, Wadhwani P, Mesters JR, Hilgenfeld R (June 2003). “Coronavirus main proteinase (3CLpro) structure: basis for design of anti-SARS drugs”. Science 300 (5626): 1763–7. Bibcode: 2003Sci...300.1763A. doi:10.1126/science.1085658. PMID 12746549.
- ^ Pacifico S, Ferretti V, Albanese V, Fantinati A, Gallerani E, Nicoli F, Gavioli R, Zamberlan F, Preti D, Marastoni M (July 2019). “Synthesis and Biological Activity of Peptide α-Ketoamide Derivatives as Proteasome Inhibitors”. ACS Medicinal Chemistry Letters 10 (7): 1086–1092. doi:10.1021/acsmedchemlett.9b00233. PMC 6627721. PMID 31312413.
- ^ Kusov Y, Tan J, Alvarez E, Enjuanes L, Hilgenfeld R (October 2015). “A G-quadruplex-binding macrodomain within the "SARS-unique domain" is essential for the activity of the SARS-coronavirus replication-transcription complex”. Virology 484: 313–22. doi:10.1016/j.virol.2015.06.016. PMC 4567502. PMID 26149721.
- ^ Zhang L, Lin D, Kusov Y, Nian Y, Ma Q, Wang J, von Brunn A, Leyssen P, Lanko K, Neyts J, de Wilde A, Snijder EJ, Liu H, Hilgenfeld R (February 2020). “α-Ketoamides as Broad-Spectrum Inhibitors of Coronavirus and Enterovirus Replication: Structure-Based Design, Synthesis, and Activity Assessment”. Journal of Medicinal Chemistry 63 (9): 4562–4578. doi:10.1021/acs.jmedchem.9b01828. PMC 7098070. PMID 32045235.
- ^ Zhang L, Lin D, Sun X, Curth U, Drosten C, Sauerhering L, Becker S, Rox K, Hilgenfeld R (March 2020). “Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors”. Science 368 (6489): 409–412. Bibcode: 2020Sci...368..409Z. doi:10.1126/science.abb3405. PMC 7164518. PMID 32198291.
- ^ Morse JS, Lalonde T, Xu S, Liu WR (March 2020). “Learning from the Past: Possible Urgent Prevention and Treatment Options for Severe Acute Respiratory Infections Caused by 2019-nCoV”. ChemBioChem 21 (5): 730–738. doi:10.1002/cbic.202000047. PMC 7162020. PMID 32022370.
- ^ Liu C, Zhou Q, Li Y, Garner LV, Watkins SP, Carter LJ, Smoot J, Gregg AC, Daniels AD, Jervey S, Albaiu D (March 2020). “Research and Development on Therapeutic Agents and Vaccines for COVID-19 and Related Human Coronavirus Diseases”. ACS Central Science 6 (3): 315–331. doi:10.1021/acscentsci.0c00272. PMC 7094090. PMID 32226821.
- ^ Ramajayam R, Tan KP, Liang PH (October 2011). “Recent development of 3C and 3CL protease inhibitors for anti-coronavirus and anti-picornavirus drug discovery”. Biochemical Society Transactions 39 (5): 1371–5. doi:10.1042/BST0391371. PMID 21936817.
- ^ Dai, Wenhao; Zhang, Bing; Jiang, Xia-Ming; Su, Haixia; Li, Jian; Zhao, Yao; Xie, Xiong; Jin, Zhenming et al. (2020-06-19). “Structure-based design of antiviral drug candidates targeting the SARS-CoV-2 main protease” (英語). Science 368 (6497): 1331–1335. doi:10.1126/science.abb4489. ISSN 0036-8075. PMC 7179937. PMID 32321856.
- ^ Pfizer (2021-06-23). A PHASE 1B, 2-PART, DOUBLE-BLIND, PLACEBO-CONTROLLED, SPONSOR-OPEN STUDY, TO EVALUATE THE SAFETY, TOLERABILITY AND PHARMACOKINETICS OF SINGLE ASCENDING (24-HOUR, PART 1) AND MULTIPLE ASCENDING (120-HOUR, PART 2) INTRAVENOUS INFUSIONS OF PF-07304814 IN HOSPITALIZED PARTICIPANTS WITH COVID-19.
- ^ Bhardwaj VK, Singh R, Sharma J, Rajendran V, Purohit R, Kumar S (May 2020). “Identification of bioactive molecules from tea plant as SARS-CoV-2 main protease inhibitors”. Journal of Biomolecular Structure and Dynamics: 1–10. doi:10.1080/07391102.2020.1766572. PMC 7256349. PMID 32397940.
- ^ a b Bhardwaj VK, Singh R, Das P, Purohit R (January 2021). “Evaluation of acridinedione analogs as potential SARS-CoV-2 main protease inhibitors and their comparison with repurposed anti-viral drugs”. Computers in Biology and Medicine 128 (1104117): 104117. doi:10.1016/j.compbiomed.2020.104117. PMC 7659809. PMID 33217661.
- ^ Kim Y, Lovell S, Tiew KC, Mandadapu SR, Alliston KR, Battaile KP, Groutas WC, Chang KO (November 2012). “Broad-spectrum antivirals against 3C or 3C-like proteases of picornaviruses, noroviruses, and coronaviruses”. Journal of Virology 86 (21): 11754–62. doi:10.1128/JVI.01348-12. PMC 3486288. PMID 22915796.
- ^ Ziebuhr J, Bayer S, Cowley JA, Gorbalenya AE (January 2003). “The 3C-like proteinase of an invertebrate nidovirus links coronavirus and potyvirus homologs”. Journal of Virology 77 (2): 1415–26. doi:10.1128/jvi.77.2.1415-1426.2003. PMC 140795. PMID 12502857.
推薦文献
[編集]- Chuck CP, Chow HF, Wan DC, Wong KB (2 November 2011). “Profiling of substrate specificities of 3C-like proteases from group 1, 2a, 2b, and 3 coronaviruses”. PLOS ONE 6 (11): e27228. Bibcode: 2011PLoSO...627228C. doi:10.1371/journal.pone.0027228. PMC 3206940. PMID 22073294.
外部リンク
[編集]- SARS coronavirus main proteinase - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス
- Peptidase C30/C16 in coronavirus, InterPro: IPR013016. The MEROPS C16 one is the "papain-like" PL-PRO.