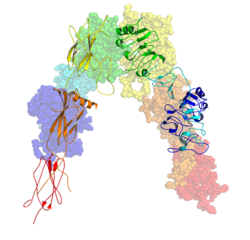
インスリン受容体
インスリン受容体 (インスリンじゅようたい、英: Insulin receptor、IR) は、インスリンとインスリン様成長因子によって活性化される膜貫通タンパク質受容体で、受容体型チロシンキナーゼのクラスに属する[5]。代謝的観点では、インスリン受容体はヒトなどにおいて血糖値の恒常性の調節という重要な役割を果たし、機能の悪化によって糖尿病やがんを含む一連の臨床症状がもたらされる[6][7]。インスリンのシグナルは、多くの細胞において血中にあるグルコースへのアクセスを制御する[注釈 1]。インスリンの血中濃度が低下した時、特にインスリン感受性が高い場合には、体細胞は膜を越えて輸送する必要のない脂質にだけアクセスするようになる。このように、インスリンは脂肪の代謝においても主要な調節因子である。生化学的観点では、インスリン受容体は単一のINSR遺伝子によってコードされ、選択的スプライシングによってIR-AまたはIR-Bのアイソフォームが生じる[8]。これらは翻訳後のタンパク質分解によってαとβのサブユニットへ切断される。これらのアイソフォームはホモ二量体またはヘテロ二量体化し、ジスルフィド結合で連結された約320 kDaの膜貫通インスリン受容体が形成される[8]。
構造
[編集]INSR遺伝子のスプライスバリアントの翻訳によって、2種類の単量体のアイソフォーム(IR-A、B)が形成される。IR-Aは11番目のエクソンが除去されたものであり、IR-Bには11番目のエクソンが含まれている。11番目のエクソンが含まれることにより、IR-Bにはフーリンによる切断部位の上流に12個のアミノ酸が挿入される。

N末端側のα鎖とC末端側のβ鎖へ切断されると、12個のアミノ酸はα鎖のC末端 (αCT) に位置することとなる。この部位は受容体とリガンドの相互作用に影響を与えていると予測されている[9]。
各単量体は、構造上8つの異なるドメインに組織化される。ロイシンリッチ反復ドメイン(leucine-rich repeat domain、L1、1–157番残基)、システインリッチ領域(cysteine-rich region、CR、158–310番残基)、2つ目のロイシンリッチ反復ドメイン(L2、311–470番残基)、3つのフィブロネクチンIII型ドメイン FnIII-1(471–595番残基)、FnIII-2(596–808番残基)、FnIII-3(809–906番残基)、さらに、FnIII-2ドメイン内にはα/βフーリン切断部位を含む挿入ドメイン(insert domain、ID、638–756番残基)があり、切断によってIDα、IDβドメインとなる。β鎖にはFnIII-3ドメインの下流に膜貫通ヘリックス (transmembrane helix、TH)、細胞内の膜近接領域 (juxtamembrane region、JM) があり、その下流には細胞内のチロシンキナーゼ触媒ドメイン (tyrosine kinase catalytic domain、TK) が存在し、細胞内のシグナル伝達を担っている[10]。
各単量体はα鎖とβ鎖へ切断されるが、受容体のホモまたはヘテロ二量体構造は、各単量体内のα鎖とβ鎖間に形成される1つのジスルフィド結合と、各単量体のα鎖間に形成される2つのジスルフィド結合によって共有結合的に維持される。細胞外領域全体には4つのリガンド結合部位があり、その立体構造は逆V字型をしている。各単量体は逆V字に平行な軸に関して擬似2回対称であり、各単量体のL2ドメインとFnIII-1ドメインが逆V字の頂上部を形成している[10][11]。
リガンドの結合
[編集]
インスリン受容体 (IR) の内在性リガンドには、インスリン、インスリン様成長因子 (IGF-I、IGF-II) が含まれる。IRの細胞外領域へのリガンドの結合によって受容体内部の構造変化が誘導され、細胞内のβ鎖のTKドメイン内のさまざまなチロシン残基が自己リン酸化される。これらの変化によって、インスリン受容体基質 (IRS)、SH2-B (Src Homology 2-B)、APSといった特定のアダプタータンパク質や、PTP1Bのようなプロテインホスファターゼが呼び寄せられ、血中グルコース濃度の恒常性に関与する下流過程が促進される[13]。

厳密に言えば、IRとリガンドの関係は複雑なアロステリック性を示す。これはスキャッチャードプロットによって示され、IRに結合しているリガンドと結合していないリガンドの比はIRに結合しているリガンド濃度の変化に対して線形関係になく、IRとリガンドは協調的結合を行う関係にあることが示唆されている[14]。さらに、IRとリガンドの解離速度は結合していないリガンドの添加によって加速され、このことは負の協同性があることを意味している。すなわち、IRへの1つ目のリガンドの結合によって2番目の活性部位への結合が阻害される、というアロステリック阻害が起こることが示されている[14]。
IRへのリガンドの結合の正確なメカニズムはまだ構造的に明らかにされていないが、システム生物学によるアプローチによって、現在利用可能なIRの細胞外領域の構造に基づいた、生物学的に妥当な条件下でのIR-リガンド(インスリン/IGF-I)動態についての予測がなされている[10][11]。
これらのモデルでは、IRの単量体には2つのインスリン結合表面(site 1、2)があるとされる。Site 1はL1ドメインとαCTから構成される「classical」なインスリン結合表面で、site 2はFnIII-1とFnIII-2の接合部に位置し、インスリンの六量体形成面に結合する「novel」な結合表面である。IRの細胞外領域の各単量体は鏡像的相補性を示し、一方の単量体のN末端側のsite 1は、他方の単量体のC末端側のsite 2と向かい合い、反対側も同様となる。現在の文献では、2番目の単量体のsite 1とsite 2をsite 3とsite 4、またはsite 1' とsite 2' と命名することでこの相補的な結合表面を区別している[5][13]。インスリンが特定の位置(site 1とsite 4/2' またはsite 3/1' とsite 2)に結合すると、リガンドによる結合表面間の「架橋」によって、2つの単量体はより近接する。現在のIR-インスリン動態の数学的モデリングからは、インスリンによる架橋によって2つの重要な帰結がもたらされる。1つ目は、IRへのさらなるリガンドの結合が減少するという、上述したIR-リガンド間の負の協調性である。2つ目は、架橋による物理的な運動によって、細胞内領域がチロシンのリン酸化が起こるコンホメーションとなることである。すなわち、これらの出来事が受容体の活性化と最終的な血中グルコース濃度の恒常性の維持に必要とされるのである[13]。
アゴニスト
[編集]シグナル伝達経路
[編集]インスリン受容体は受容体型チロシンキナーゼで、アゴニストの結合に伴い各サブユニットが結合パートナーのチロシン残基をリン酸化する。リン酸基の付加によってインスリン受容体基質 (IRS-1) の結合部位が形成され、IRS-1もリン酸化されて活性化される。活性化されたIRS-1はシグナルの伝達を開始し、PI3キナーゼを結合して活性化を行う。PI3キナーゼは、ホスファチジルイノシトール4,5-ビスリン酸 (PIP2) からホスファチジルイノシトール-3,4,5-トリスリン酸 (PIP3) への変換を触媒する。PIP3はセカンドメッセンジャーとして機能し、ホスホイノシチド依存性キナーゼ1 (PDPK1) の活性化を誘導する。このキナーゼは、よく知られたプロテインキナーゼB (PKB/Akt) など、いくつかのキナーゼを活性化する。PKBはグルコーストランスポーターGLUT4を含む小胞を、SNAREタンパク質を介して細胞膜へ輸送させる。これによってグルコースの細胞内への拡散が促進される。またPKBは、グリコーゲンシンターゼを阻害する酵素GSK-3をリン酸化して阻害する。つまりPKBは、グリコーゲン合成過程を開始させ、最終的には血中グルコース濃度を減少させる機能を持つ[15]。
- インスリンシグナルの伝達
-
グルコースの取り込みと代謝におけるインスリンの影響: インスリンが受容体に結合し(1)、多くのタンパク質活性化カスケードが開始される(2)。グルコーストランスポーターGlut4の細胞膜への移動とグルコースの流入(3)、グリコーゲン合成(4)、解糖(5)、脂肪酸の合成(6)などが活性化される。
-
インスリンシグナルの伝達: シグナル伝達過程では、活性化されたタンパク質は膜に埋め込まれたPIP2に結合する。
機能
[編集]遺伝子発現の調節
[編集]活性化されたIRS-1は、インスリンによって調節される遺伝子の転写を促進するための、細胞内のセカンドメッセンジャーとして機能する。まず、Grb2タンパク質のSH2ドメインがIRS-1のリン酸化チロシン残基に結合する。Grb2はSOSに結合できるようになり、SOSはGタンパク質であるRasに結合しているGDPのGTPへの交換を触媒する。これによって活性化されたRasはリン酸化カスケードを開始し、最終的に活性化されたMAPKは核へ移行して核内のさまざまな転写因子 (Elk1など) をリン酸化する。
インスリンの分解
[編集]インスリン分子は受容体に結合してその作用を果たした後、細胞外環境へ送り返されるか、細胞内で分解される。通常、分解はインスリン-受容体複合体のエンドサイトーシスを伴い、その後インスリン分解酵素によって分解される。ほとんどのインスリン分子は肝細胞で分解される。典型的なインスリン分子は、血液循環への最初の放出から約71分で最終的な分解が行われる[16]。
免疫系
[編集]代謝における機能に加え、インスリン受容体は、マクロファージ、B細胞、T細胞といった免疫細胞でも発現している。T細胞におけるインスリン受容体の発現は休止状態では検出されないが、T細胞受容体の活性化に伴って発現上昇が起こる。事実、インスリンの外的供給によってin vitroでのT細胞の増殖が促進されることが動物モデルで示されている。インスリン受容体によるシグナル伝達は、急性の感染や炎症時にT細胞の潜在的影響力を最大化するために重要である[17][18]。
病理
[編集]インスリン受容体の活性化の主要な役割は、グルコースの取り込みの誘導である。そのため、「インスリン非感受性」もしくはインスリン受容体シグナル伝達の低下によって細胞はグルコースを取り込むことができなくなり、2型糖尿病がもたらされる。その帰結は高血糖と、糖尿病に起因するすべての後遺症である。
インスリン抵抗性の患者は黒色表皮腫を発症することがある。INSR遺伝子のホモ接合変異によって、ドナヒュー症候群 (妖精症) が引き起こされる。この常染色体劣性異常によって、インスリン受容体は完全に機能を持たなくなる。患者には、低位置でしばしば突出した耳、怒り鼻、厚い唇、そして重度の発育遅滞がみられる。ほとんどの場合予後は極めて悪く、出生後1年以内に死に至る。同じ遺伝子の他の変異では、より重症度の低いラブソン-メンデンホール症候群が引き起こされ、患者には特徴的な歯の異常、歯肉の肥大、松果体の増大がみられる。どちらの疾患でも血中グルコース濃度の大幅な変動が見られ、食事後にいったん極めて高値となり、その後異常な低値まで急速に低下する[19]。
相互作用
[編集]インスリン受容体はこれらと相互作用することが示されている。
注釈
[編集]- ^ ただし細胞にグルコースを取り込むトランスポータにも何種類か存在しており、GLUT1やGLUT2のようにインスリンのシグナルとは無関係に細胞外からグルコースを取り込むトランスポータも存在する。逆に、GLUT4のように、インスリンのシグナルが入ると動き出して高効率でグルコースを取り込むトランスポータも存在する。
出典
[編集]- ^ a b c GRCh38: Ensembl release 89: ENSG00000171105 - Ensembl, May 2017
- ^ a b c GRCm38: Ensembl release 89: ENSMUSG00000005534 - Ensembl, May 2017
- ^ Human PubMed Reference:
- ^ Mouse PubMed Reference:
- ^ a b “Ligand-induced activation of the insulin receptor: a multi-step process involving structural changes in both the ligand and the receptor”. BioEssays 31 (4): 422–34. (April 2009). doi:10.1002/bies.200800210. PMID 19274663.
- ^ “The human insulin receptor cDNA: the structural basis for hormone-activated transmembrane signalling”. Cell 40 (4): 747–58. (April 1985). doi:10.1016/0092-8674(85)90334-4. PMID 2859121.
- ^ “Proinsulin binds with high affinity the insulin receptor isoform A and predominantly activates the mitogenic pathway”. Endocrinology 153 (5): 2152–63. (May 2012). doi:10.1210/en.2011-1843. PMID 22355074.
- ^ a b “Insulin receptor isoforms and insulin receptor/insulin-like growth factor receptor hybrids in physiology and disease”. Endocrine Reviews 30 (6): 586–623. (October 2009). doi:10.1210/er.2008-0047. PMID 19752219.
- ^ “Insight into the molecular basis for the kinetic differences between the two insulin receptor isoforms”. The Biochemical Journal 440 (3): 397–403. (December 2011). doi:10.1042/BJ20110550. PMID 21838706.
- ^ a b c “Structural resolution of a tandem hormone-binding element in the insulin receptor and its implications for design of peptide agonists”. Proceedings of the National Academy of Sciences of the United States of America 107 (15): 6771–6. (April 2010). Bibcode: 2010PNAS..107.6771S. doi:10.1073/pnas.1001813107. PMC 2872410. PMID 20348418.
- ^ a b “Structure of the insulin receptor ectodomain reveals a folded-over conformation”. Nature 443 (7108): 218–21. (September 2006). Bibcode: 2006Natur.443..218M. doi:10.1038/nature05106. PMID 16957736.
- ^ “Visualization of ligand-induced transmembrane signaling in the full-length human insulin receptor”. The Journal of Cell Biology 217 (5): 1643–1649. (May 2018). doi:10.1083/jcb.201711047. PMID 29453311.
- ^ a b c “Harmonic oscillator model of the insulin and IGF1 receptors' allosteric binding and activation”. Molecular Systems Biology 5 (5): 243. (Feb 2009). doi:10.1038/msb.2008.78. PMC 2657531. PMID 19225456.
- ^ a b “Insulin interactions with its receptors: experimental evidence for negative cooperativity”. Biochemical and Biophysical Research Communications 55 (1): 154–61. (November 1973). doi:10.1016/S0006-291X(73)80072-5. PMID 4361269.
- ^ Berg, Jeremy M.; Tymoczko, John L.; Stryer, Lubert; Berg, Jeremy M.; Tymoczko, John L.; Stryer, Lubert (2002). Biochemistry (5th ed.). W H Freeman. ISBN 0716730510
- ^ “Insulin degradation: progress and potential”. Endocrine Reviews 19 (5): 608–24. (October 1998). doi:10.1210/er.19.5.608. PMID 9793760.
- ^ “Insulin Receptor-Mediated Stimulation Boosts T Cell Immunity during Inflammation and Infection”. Cell Metabolism. (August 2018). doi:10.1016/j.cmet.2018.08.003. PMID 30174303.
- ^ “The Insulin Receptor Plays a Critical Role in T Cell Function and Adaptive Immunity”. Journal of Immunology 198 (5): 1910–1920. (March 2017). doi:10.4049/jimmunol.1601011. PMID 28115529.
- ^ “Genotype-phenotype correlation in inherited severe insulin resistance”. Human Molecular Genetics 11 (12): 1465–75. (June 2002). doi:10.1093/hmg/11.12.1465. PMID 12023989.
- ^ “Membrane glycoprotein PC-1 inhibition of insulin receptor function occurs via direct interaction with the receptor alpha-subunit”. Diabetes 49 (1): 13–9. (January 2000). doi:10.2337/diabetes.49.1.13. PMID 10615944.
- ^ “Identification of Grb10 as a direct substrate for members of the Src tyrosine kinase family”. Oncogene 19 (25): 2895–903. (June 2000). doi:10.1038/sj.onc.1203616. PMID 10871840.
- ^ “Interaction between the Grb10 SH2 domain and the insulin receptor carboxyl terminus”. The Journal of Biological Chemistry 271 (15): 8882–6. (April 1996). doi:10.1074/jbc.271.15.8882. PMID 8621530.
- ^ “Grb-IR: a SH2-domain-containing protein that binds to the insulin receptor and inhibits its function”. Proceedings of the National Academy of Sciences of the United States of America 92 (22): 10287–91. (October 1995). Bibcode: 1995PNAS...9210287L. doi:10.1073/pnas.92.22.10287. PMC 40781. PMID 7479769.
- ^ “Grb10 interacts differentially with the insulin receptor, insulin-like growth factor I receptor, and epidermal growth factor receptor via the Grb10 Src homology 2 (SH2) domain and a second novel domain located between the pleckstrin homology and SH2 domains”. The Journal of Biological Chemistry 273 (12): 6860–7. (March 1998). doi:10.1074/jbc.273.12.6860. PMID 9506989.
- ^ “Human GRB-IRbeta/GRB10. Splice variants of an insulin and growth factor receptor-binding protein with PH and SH2 domains”. The Journal of Biological Chemistry 272 (5): 2659–67. (January 1997). doi:10.1074/jbc.272.5.2659. PMID 9006901.
- ^ “Evidence for an interaction between the insulin receptor and Grb7. A role for two of its binding domains, PIR and SH2”. Oncogene 19 (16): 2052–9. (April 2000). doi:10.1038/sj.onc.1203469. PMID 10803466.
- ^ “Phosphorylation of Ser307 in insulin receptor substrate-1 blocks interactions with the insulin receptor and inhibits insulin action”. The Journal of Biological Chemistry 277 (2): 1531–7. (January 2002). doi:10.1074/jbc.M101521200. PMID 11606564.
- ^ “Insulin receptor substrate-2 binds to the insulin receptor through its phosphotyrosine-binding domain and through a newly identified domain comprising amino acids 591-786”. The Journal of Biological Chemistry 271 (11): 5980–3. (March 1996). doi:10.1074/jbc.271.11.5980. PMID 8626379.
- ^ “Interaction of MAD2 with the carboxyl terminus of the insulin receptor but not with the IGFIR. Evidence for release from the insulin receptor after activation”. The Journal of Biological Chemistry 272 (15): 10035–40. (April 1997). doi:10.1074/jbc.272.15.10035. PMID 9092546.
- ^ “Insulin induces specific interaction between insulin receptor and protein kinase C delta in primary cultured skeletal muscle”. Molecular Endocrinology 15 (4): 565–74. (April 2001). doi:10.1210/mend.15.4.0612. PMID 11266508.
- ^ “Differential effects of tumor necrosis factor-alpha on protein kinase C isoforms alpha and delta mediate inhibition of insulin receptor signaling”. Diabetes 51 (6): 1921–30. (June 2002). doi:10.2337/diabetes.51.6.1921. PMID 12031982.
- ^ “Insulin receptor kinase phosphorylates protein tyrosine phosphatase containing Src homology 2 regions and modulates its PTPase activity in vitro”. Biochemical and Biophysical Research Communications 199 (2): 780–5. (March 1994). doi:10.1006/bbrc.1994.1297. PMID 8135823.
- ^ “Adapter function of protein-tyrosine phosphatase 1D in insulin receptor/insulin receptor substrate-1 interaction”. The Journal of Biological Chemistry 270 (49): 29189–93. (December 1995). doi:10.1074/jbc.270.49.29189. PMID 7493946.
- ^ “SH2-Balpha is an insulin-receptor adapter protein and substrate that interacts with the activation loop of the insulin-receptor kinase”. The Biochemical Journal 335 ( Pt 1) (1): 103–9. (October 1998). doi:10.1042/bj3350103. PMC 1219757. PMID 9742218.
- ^ “Alternative splicing, gene localization, and binding of SH2-B to the insulin receptor kinase domain”. Mammalian Genome 10 (12): 1160–7. (December 1999). doi:10.1007/s003359901183. PMID 10594240.
関連文献
[編集]- “Protein kinase phosphorylation site sequences and consensus specificity motifs: tabulations”. Methods in Enzymology 200: 62–81. (1991). doi:10.1016/0076-6879(91)00127-I. PMID 1956339.
- “Structural and functional heterogeneity of insulin receptors”. Cellular Signalling 7 (2): 85–91. (February 1995). doi:10.1016/0898-6568(94)00071-I. PMID 7794689.
- “Insulin-like growth factor II (IGF-II)”. The International Journal of Biochemistry & Cell Biology 30 (7): 767–71. (July 1998). doi:10.1016/S1357-2725(98)00048-X. PMID 9722981.
- “Differential regulation of signaling pathways for insulin and insulin-like growth factor I”. Acta Biochimica Polonica 46 (1): 51–60. (1999). PMID 10453981.
- “The functional significance of Shc in insulin signaling as a substrate of the insulin receptor”. Endocrine Journal 47 (4): 373–81. (August 2000). doi:10.1507/endocrj.47.373. PMID 11075717.
- “Insulin receptor--structural and functional characteristics”. Medical Science Monitor 7 (1): 169–77. (2001). PMID 11208515.
- “Phosphorylation of calmodulin. Functional implications”. European Journal of Biochemistry / FEBS 269 (15): 3619–31. (August 2002). doi:10.1046/j.1432-1033.2002.03038.x. PMID 12153558.
外部リンク
[編集]- Insulin receptor - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス