ベクター (遺伝子工学)
ベクター (vector) とは、外来遺伝物質を別の細胞に人為的に運ぶために利用されるDNAまたはRNA分子である。任意の遺伝子やDNA、RNA配列を導入先の細胞内で増幅・維持・導入させる、いわゆる遺伝子組換え技術に用いられる。具体的には、プラスミドやコスミド、ラムダファージ、および人工染色体等を指す。これらのベクターは実験操作を簡便にする目的で、複製起点、マルチクローニングサイト、および選択マーカーを持つことが多い。語源はラテン語の運び屋 (vehere) に由来する。外来DNAを含むベクターは通常、組換えDNAとみなされる。
概要
[編集]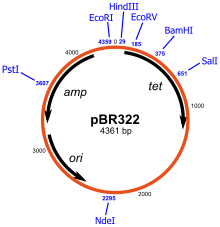
ベクター自体は単純なDNAおよびRNA配列であり、インサート配列(導入遺伝子)を他の細胞等に導入するための輸送器(バックボーン)として機能する。一般的に細胞へ遺伝情報を移入する目的の一つは、標的細胞で導入遺伝子を単離 (isolate) したり、あるいは増幅 (cloning) させたり、発現 (express) させたりすることが多い。原理的にはあらゆるベクターはクローニングに使用することができるが、クローニングの目的に特化して設計されたベクター(クローニングベクター)も存在し、一般的にクローニング用途やライブラリー作成としてはそれらが利用されることが多い。一方で、導入遺伝子の転写やタンパク質発現などの目的のために設計されたベクターも存在する。標的細胞における導入遺伝子の発現を目的として設計されたベクターのことを発現ベクターと呼び、これらは一般的に導入遺伝子の発現を促進するようにプロモーター配列を組み込むことで、挿入したDNA断片からタンパク質の翻訳を促進させている。他方、転写ベクターと呼ばれるより単純なベクターは転写のみを目的としており、導入先の細胞内で複製を進めることはできるが、導入遺伝子の翻訳は行うことができない。そのため転写ベクターは、挿入遺伝子をmRNAで増幅する目的で使用される。また、挿入するDNA断片の大きさや挿入の目的によって、それを挿入するために様々な特徴を付加された媒体がベクターとして使い分けられる。
DNAの操作は通常、大腸菌での維持に必要な要素を含む、いわゆる大腸菌ベクターで行われる。他方、大腸菌に加えて酵母や植物、哺乳動物等に由来する細胞系で維持するためのベクターも存在し、 シャトルベクターと呼ばれる。このようなベクターでは、非細菌の宿主生物に移入可能な細菌・ウイルス的な要素を有する[1]。
ベクターを標的細胞へ入れることは通常、細菌細胞の形質転換[2]、真核細胞であればトランスフェクション [3]と呼ばれ、ウイルスベクターの場合では形質導入と形容されることもある[4]。
プラスミドベクター
[編集]プラスミドを改変し、ベクターとして利用したものである。プラスミドは、宿主細胞の複製機構を使用して複製できる、二本鎖の染色体外のDNA配列を指し、一般には環状である[5]。プラスミドベクターは、宿主内でプラスミドを半独立的に複製させるための複製起点を必ず持っている。プラスミドは大腸菌を含む多くの細菌・古細菌に広く見出されるだけでなく、例えば、酵母やサッカロミセス・セレビシエ(Saccharomyces cerevisiae)といったいくつかの真核生物においても見つかっている[6] バクテリアのプラスミドは接合性(伝染性)のものと非接合性の2種類に大別できる。接合性のものは、細胞同士の接合を介してDNAの転移が媒介されるため、個体群の細菌細胞間で急速に広がる。例えば、Fプラスミド、Rプラスミド、いくつかのcolプラスミドが知られている。一方で、非接合性のものは接合によってDNAを媒介せず、Rおよびcolプラスミドで多く知られている。
一般的なクローニング目的では、この用途に特化して機能をデザインされたプラスミドを利用する場合が多い。これらのプラスミドは、一般に非接合性であり、他の様々な機能が付加されている。例えばマルチクローニングサイトと呼ばれる、複数の制限酵素切断部位が連なる部位は、導入遺伝子配列の挿入領域として利用することができる。クローニングベクターを導入された細菌は、数時間で何百万ものベクターのコピーを細菌内で生成し、増幅されたベクターを細菌から抽出して下流の実験操作に利用することができる。一方で転写ベクターとして利用されるプラスミドは、遺伝子発現を行わないように、あえてタンパク質発現に重要な配列を欠いている場合がある。また、タンパク質発現に使用される発現ベクターのプラスミドでは、リボソーム結合部位 、開始コドン、停止コドンなど、タンパク質の翻訳のための要素が含まれている。
クローニングベクター
[編集]- lac promoter: ラクトースオペロンの広義のプロモーター。イソプロピル-1-チオ-β-ガラクトシド (IPTG) 等の存在下でlacZ αとMCSに挿入された配列を転写する。
- MCS: マルチクローニングサイト。様々な制限酵素に認識される配列が存在する部位。これを適当な制限酵素で切断し、外来のDNA配列を挿入する。
- lacZ α: lacZのα断片。MCSに外来DNAが挿入されると機能するlacZ αタンパク質が翻訳されなくなる。このため、lacZ ωをもつ宿主に形質転換した際にブルー・ホワイトセレクションで挿入断片の有無を簡易検定できる。
- ampicillin resistance gene: 抗生物質耐性遺伝子の一種。アンピシリン存在下でも宿主の大腸菌が増殖することを可能にする。従って、このベクターをもっている大腸菌のみを選択的に得ることができるようになる。他の抗生物質の耐性遺伝子を持つベクターもある。
- ColE1: 大腸菌内でプラスミドを複製するための複製起点。比較的多数のプラスミドを1細胞内に保持する。
pBR322
[編集]1977年に構築された、初期型(古典型)クローニングベクターの最終形。アンピシリン耐性遺伝子とテトラサイクリン耐性遺伝子をもち、制限酵素サイトを利用して目的遺伝子のクローニングが可能。ColE1型で、1細胞当たり15–20のプラスミドが保持される。このpBR322をもとに改良型の多くのプラスミドベクターがつくられた。
pUCプラスミドベクター
[編集]pUC19/pUC18などが代表的である。アンピシリン耐性遺伝子を持ち、挿入DNA断片の有無をブルー・ホワイトセレクションで判別できるため、通常サブクローニング実験やプラスミドの大量調製に利用されている。ColE1型で、1細胞当たり500–700のプラスミドが保持される。
pET系プラスミドベクター
[編集]T7プロモーター支配下に目的遺伝子を連結し、T7 RNA polymeraseによるT7プロモーターからの特異的転写を利用した誘導型高発現プラスミドとして主に利用される。アンピシリンあるいはカナマイシン耐性遺伝子を持つ。ColE1型で、1細胞当たり15–20のプラスミドが保持される。
コスミドベクター
[編集]フォスミドベクター
[編集]ウイルスベクター
[編集]非感染性に改変されたウイルスやバクテリオファージ(細菌に感染するウイルス)の複製系を利用する。ウイルスプロモーターを介することで、導入遺伝子の転写翻訳を行うことができる。
完全な複製能を持つλファージベクターやRSVベクターなどのウイルスベクターを除き、組換えウイルスは多くの場合、単独ではウイルス粒子へとパッケージングされない。そのため、欠失遺伝子を補う目的でパッケージング細胞を作成し、それに感染させる必要がある。この細胞から生じた組換えウイルスは宿主に感染すると外来遺伝子を発現できるが、パッケージングに必要な遺伝子を持たないので自己複製はできない。この性質を利用して、ヒトに対する遺伝子治療に用いられることもある(レトロウイルス、アデノウイルス、アデノ随伴ウイルスなど)。
また、ウイルスベクターはしばしば、宿主ゲノムへの挿入物の恒久的な組み込みのために設計されている。この場合、導入遺伝子を組み込んだ後には、宿主ゲノムに別個の遺伝子マーカーが残る。例えばレトロウイルスでは、遺伝子挿入後には特徴的なレトロウイルス組み込みパターンが残り、ウイルスベクターが宿主ゲノムに組み込まれたことを後から検出することができる。
人工染色体ベクター
[編集]人工染色体は大きく3種類に大別でき、それぞれ酵母人工染色体 (YAC)、細菌人工染色体 (BAC)、ヒト人工染色体 (HAC)である。人工染色体は他のベクターよりもはるかに大きなDNA断片を運ぶことができる[7]。YACおよびBACは、最大300,000塩基長のDNA断片を運ぶことができる。それぞれの人工染色体には、複製起点、セントロメア、およびテロメア末端配列という構造的に必須な要素が含まれる[8]。
YAC (Yeast artificial chromosome)
[編集]出芽酵母を宿主とする人工染色体ベクター。クローン化できるDNA断片が数 Mbと巨大なので長大なコンティグを作成する際に適するが、キメラクローンを生じやすいこと、クローン化した断片を安定に保持できないことが欠点。
BAC (bacterial artificial chromosome)
[編集]大腸菌を宿主とする人工染色体ベクター。大腸菌プラスミドの一種Fプラスミドの複製系を利用する。最大約300 kbの断片を安定にクローン化することができる。
PAC (P1-derived artificial chromosome)
[編集]ファージP1の複製系を利用したクローニングベクター。BACと同様、最大約300 kbの断片を安定にクローン化することができる。
転写
[編集]導入遺伝子の発現を目的とする場合、まずその導入遺伝子の転写を促進することが必要となる。遺伝子はまず転写によって、複数のmRNAに増幅コピーされる[9]。より多くのmRNAを生産することで、より多くの量のタンパク質を発現することができる。そして、生成されるmRNAのコピー数は、ベクター内部で使用されるプロモーターに依存する[10]。発現は2タイプに分類でき、一つは恒常的(タンパク質がバックグラウンドで常に生成される)、もう一つは誘導可能(例えば化学的誘導物質が添加されたときといった、特定の条件下でのみタンパク質が発現する)なものである。この異なる2タイプの発現は、使用するプロモーターとオペレーターのタイプによって選択することができる。
ウイルスプロモーターは、多くの細胞株やタイプで一定量の転写を強制するため、プラスミドやウイルスベクターを用いた導入遺伝子の恒常的発現によく利用される[11]。一方で、誘導可能な発現は、誘導条件に応答するプロモーターに依存する。たとえば、 マウス乳腺腫瘍ウイルスのプロモーターはデキサメタゾンの添加後にのみ転写を開始し、またDrosophilia熱ショックプロモーターは高温に晒すことで転写を開始する。
いくつかのベクターは、in vitroでのmRNA産生(転写)という唯一の目的のために設計されており、転写ベクターと呼ばれる。このベクターではポリアデニル化と終結に必要な配列を欠いている場合があり、タンパク質生産に使うことはできない。
発現
[編集]発現ベクターは、インサート配列の転写とそれに続くmRNAの翻訳によってタンパク質を生成する。したがって、単純な転写のみのベクターよりもより多くの機能がベクター中に必要となる。異なる宿主生物で発現させるためには、それに応じた異なる機能が必要になる。具体的には、転写開始プロモーターや翻訳開始のためのリボソーム結合部位 、終結シグナルなどが該当する。
原核生物の発現ベクターの特徴
[編集]- プロモーター:一般的に使用される誘導プロモーターは、 lacオペロンとT7プロモーターに由来するプロモーターである。また、他の強力なプロモーターとして、 TrpプロモーターとLac Operonプロモーターの両方のハイブリッドであるTrpプロモーターとTac-Promoterが挙げられる。
- リボソーム結合部位 (RBS):プロモーターに従い、目的のタンパク質の効率的な翻訳を促進する。
- 翻訳開始サイト:AUG開始コドンの8塩基対上流に、RBSに囲まれたShine-Dalgarno配列を含む。
真核生物の発現ベクターの特徴
[編集]真核生物の発現ベクターには上記に加え、以下をコードする配列が必要となる。
- ポリアデニル化テール :転写されたmRNA前駆体の最後にポリアデニル化テール作成し、エキソヌクレアーゼからmRNAを保護し、mRNA産生を安定化する。
- 最小UTR長:UTRには、転写または翻訳を妨げる可能性のある特定の特性が含まれているため、最適な発現ベクターでは、最短のUTRが含まれるか、あるいはまったく含まれない。
- コザック配列 :コザック配列は、mRNAの翻訳のためにリボソームと結合する領域となる。
ベクター中に付加される機能の例
[編集]人工的に構築されたベクターには、予め必須コンポーネントは含まれており、一部のベクターについてはその他の追加機能が含まれている。
- 複製起点:宿主細胞内でのベクターの複製と維持に必要となる。
- プロモーター:プロモーターは、ベクターの導入遺伝子および抗生物質耐性遺伝子などのベクター内の他の遺伝子の転写を促進するために使用される。一部のクローニングベクターは、クローニングされたインサートのプロモーターを必要としないが、クローニングされた産物を発現させるため発現ベクターにおいては必須の構成要素となる。
- クローニングサイト:ライゲーションを通じて外来DNAをベクターに挿入できる、マルチクローニングサイトの領域。
- 遺伝的マーカー:ベクターが宿主ゲノムDNAに統合されていることを確認するための遺伝的マーカー。
- 抗生物質耐性:抗生物質耐性遺伝子を備えたベクターは、抗生物質を含む成長培地で培養することで、ベクターを取り込んだ細胞を選択することができる。
- エピトープ:一部のベクターには、発現したタンパク質に組み込むことができる特定のエピトープの配列が含まれている場合がある。これにより、標的タンパク質を発現する細胞の抗体同定を可能にする。
- レポーター遺伝子:一部のベクターには、挿入配列を含んでいるプラスミドを同定するためのレポーター遺伝子が含まれている場合がある。代表的な例は、ガラクトースを消化する酵素であるβ-ガラクトシダーゼのN末端フラグメントをコードするlacZ-αである。マルチクローニングサイトをlacZ-α内部に含むことで、ベクターへのライゲーションが成功すると遺伝子配列が破壊されβ-ガラクトシダーゼを不活性させることができる。細胞をガラクトース類似体(X-gal)を含む培地で培養することで、コロニーの色に青/白の変化がつくため、容易に挿入配列が入ったベクターのみを識別して選択することができる(ブルー・ホワイトセレクション)。すなわち、β-ガラクトシダーゼを発現している細胞(インサートは含まれない)は青いコロニーになる反面、挿入物を含む可能性があるものは白いコロニーとなる。 その他の一般的に使用されるレポーターには、 緑色蛍光タンパク質とルシフェラーゼが挙げられる。
- ターゲティング配列:発現したタンパク質を、細胞内の特定の細胞小器官や細菌のペリプラズム空間などの特定の場所に誘導するように、タンパク質にターゲティング配列を付加する場合がある。
- タンパク質精製タグ:一部の発現ベクターには、発現したタンパク質の精製を容易にするタンパク質またはペプチド配列が含まれている。 例としては、ポリヒスチジンタグ 、グルタチオン-S-トランスフェラーゼ、マルトース結合タンパク質などが挙げられる。これらのタグのいくつかは、標的タンパク質の溶解度を高めることもできる。標的タンパク質はタンパク質タグと融合するが、タンパク質とタグの間のポリペプチドリンカー領域に配置されたプロテアーゼ切断部位により、タグを後から除去することができる。
参考文献
[編集]- 『生化学辞典 第4版』東京化学同人、2007年。ISBN 9784807906703。
- pUCプラスミドにまつわるエトセトラ 生物工学基礎講座 バイオよもやま話 (PDF) - 日本生物工学会
引用文献
[編集]- ^ Acquaah, George (16 August 2012). Principles of Plant Genetics and Breeding. John Wiley & Sons Inc. ISBN 978-1-118-31369-5
- ^ “Bacterial transformation: distribution, shared mechanisms and divergent control”. Nature Reviews. Microbiology 12 (3): 181–96. (March 2014). doi:10.1038/nrmicro3199. PMID 24509783.
- ^ “MeSH Browser” (英語). meshb.nlm.nih.gov. 2018年4月16日閲覧。
- ^ Hartl, Daniel L; Jones, Elizabeth W (1998). Genetics: principles and analysis (4th ed.). Sudbury, Mass.: Jones and Bartlett Publishers. ISBN 978-0-7637-0489-6. OCLC 45730915
- ^ del Solar, Gloria; Giraldo, Rafael; Ruiz-Echevarría, María Jesús; Espinosa, Manuel; Díaz-Orejas, Ramón (June 1998). “Replication and Control of Circular Bacterial Plasmids”. Microbiology and Molecular Biology Reviews 62 (2): 434–464. doi:10.1128/MMBR.62.2.434-464.1998. ISSN 1092-2172. PMC 98921. PMID 9618448.
- ^ “Chapter 2 - Vectors for Gene Cloning: Plasmids and Bacteriophages”. Gene Cloning and DNA Analysis: An Introduction (6th ed.). Wiley-Blackwell. (2010). ISBN 978-1-4051-8173-0
- ^ Julin, Douglas (2014). “Artificial Chromosomes” (英語). Molecular Life Sciences. Springer, New York, NY. pp. 1–3. doi:10.1007/978-1-4614-6436-5_91-3. ISBN 978-1-4614-6436-5
- ^ Murray, Andrew; Szostak, Jack (November 1987). “Artificial Chromosomes”. Scientific American 257 (5): 62–68. doi:10.1038/scientificamerican1187-62. PMID 3317814.
- ^ Solomon, Eldra Pearl; Berg, Linda R; Martin, Diana W (2005). Biology (8th ed.). Belmont, CA: Brooks/Cole Thomson Learning. ISBN 978-0-495-31714-2. OCLC 123008833
- ^ “A comparative analysis of constitutive promoters located in adeno-associated viral vectors”. PLOS One 9 (8): e106472. (2014-08-29). doi:10.1371/journal.pone.0106472. PMC 4149579. PMID 25170953.
- ^ “Viral promoters can initiate expression of toxin genes introduced into Escherichia coli”. BMC Biotechnology 5: 19. (June 2005). doi:10.1186/1472-6750-5-19. PMC 1181807. PMID 15967027.

