「触媒三残基」の版間の差分
編集の要約なし |
英語版Catalytic triadの2020-12-11T17:07:25(UTC)版から一部(Mechanism)を翻訳して追加 |
||
| (同じ利用者による、間の3版が非表示) | |||
| 1行目: | 1行目: | ||
[[File:Catalytic triad of TEV protease.png|thumb|360px|酵素[[TEVプロテアーゼ]]は、その[[活性部位]]に残基 (赤色) の触媒残基の一例を含んでいる。この三残基は、[[アスパラギン酸]] ([[酸]])、[[ヒスチジン]] ([[塩基]]) 、[[セリン]] ([[求核剤]]) で構成されている。[[基質 (化学)|基質]] (黒) は[[結合部位]]で結合しており、この3つの残基の隣に配向する。({{PDB|1LVM}})]] |
|||
{{出典の明記|date=2011年8月13日 (土) 01:23 (UTC)}} |
|||
| ⚫ | |||
'''触媒三残基'''(しょくばいさんざんき、{{lang-en-short|Catalytic triad}})とは、[[酵素]]の[[活性部位]]に必要な[[アミノ酸]][[残基]]である[[セリン]]、[[ヒスチジン]]、[[アスパラギン酸]]の組み合わせを意味する。 |
|||
触媒三残基(しょくばいさんざんき、英: Catalytic triad)とは、いくつかの[[酵素]]の[[活性部位]]に見られる3つの配位[[アミノ酸]]のセットである<ref name=":2">{{cite journal|vauthors=Dodson G, Wlodawer A|date=1998|title=Catalytic triads and their relatives|journal=[[Trends in Biochemical Sciences|Trends Biochem. Sci.]]|volume=23|issue=9|pages=347–52|doi=10.1016/S0968-0004(98)01254-7|pmid=9787641}}</ref><ref name="buller1">{{cite journal|vauthors=Buller AR, Townsend CA|date=2013|title=Intrinsic evolutionary constraints on protease structure, enzyme acylation, and the identity of the catalytic triad|journal=[[Proceedings of the National Academy of Sciences of the United States of America|Proc. Natl. Acad. Sci. U.S.A.]]|volume=110|issue=8|pages=E653–61|bibcode=2013PNAS..110E.653B|doi=10.1073/pnas.1221050110|pmc=3581919|pmid=23382230}}</ref>。触媒三残基は、[[ヒドロラーゼ]]および[[トランスフェラーゼ]]酵素 (例えば、[[プロテアーゼ]]、[[アミダーゼ]]、[[エステラーゼ]]、アシルラーゼ、[[リパーゼ]]および[[β-ラクタマーゼ]]) で最も一般的に見られる。[[酸]]-[[塩基]]-[[求核剤]] 三残基は、[[酵素反応|共有結合触媒]]のための求核性残基を生成するための一般的なモチーフである。この[[残基]]は電荷リレーネットワークを形成して求核剤を分極および活性化し、[[基質 (化学)|基質]]を攻撃して[[共有結合]]中間体を形成し、[[加水分解]]されて{{仮リンク|生成物 (化学)|en|Product (chemistry)|label=生成物}}を放出し、遊離酵素を再生する。求核剤は、最も一般的には[[セリン]]や[[システイン]]アミノ酸であるが、時には[[スレオニン]]または[[セレノシステイン]]も含まれている。酵素の[[三次構造|三次元構造]]は、配列上 ([[一次構造]]) では離れていても、三残基が正確な方向にまとまっていることを示している<ref name="Stryer_Ch9">{{cite book|url=https://www.ncbi.nlm.nih.gov/books/NBK21154/|title=Biochemistry|vauthors=Stryer L, Berg JM, Tymoczko JL|publisher=W.H. Freeman|year=2002|isbn=9780716749554|edition=5th|location=San Francisco|chapter=9 Catalytic Strategies|chapter-url=https://www.ncbi.nlm.nih.gov/books/NBK22526/}}</ref>。 |
|||
機能 (および三残基の求核剤) の{{仮リンク|分岐進化|en|Divergent evolution|label=}}と同様に、触媒三残基は[[収斂進化]]の最良の例のいくつかを示している。触媒作用に対する化学的制約により、同じ触媒解明が、少なくとも23の別々の[[スーパーファミリー]]で独立して展開している。その[[作用機序]]は、[[生化学]]的に最もよく研究されているものの一つである<ref name="Perutz 1992">{{cite book|title=Protein structure. New approaches to disease and therapy|last=Perutz|first=Max|publisher=W.H. Freeman and Co|year=1992|isbn=9780716770213|location=New York|name-list-style=vanc}}</ref><ref name="cite journal|pmid=7849591">{{cite journal|date=1994|title=Proteolytic enzymes past and present: the second golden era. Recollections, special section in honor of Max Perutz|journal=[[Protein Science|Protein Sci.]]|volume=3|issue=10|pages=1734–9|doi=10.1002/pro.5560031013|pmid=7849591|pmc=2142620|vauthors=Neurath H}}</ref>。 |
|||
== 歴史<!-- History --> == |
|||
[[トリプシン]]と[[キモトリプシン]]は、1930年代に初めて精製された酵素である<ref>{{cite journal|date=1990|title=Endothelin-induced vasoconstriction and release of atrial natriuretic peptides in the rat|journal=[[Acta Physiologica Scandinavica|Acta Physiol. Scand.]]|volume=138|issue=4|pages=549–56|doi=10.1111/j.1748-1716.1990.tb08883.x|pmid=2141214|vauthors=Ohman KP, Hoffman A, Keiser HR}}</ref>。1950年代には、トリプシンとキモトリプシンのそれぞれのセリンが触媒的求核剤として同定された (ジイソプロピルフルオロリン酸修飾)<ref>{{cite journal|date=1958|title=Amino Acid Sequence in the Region of Diisopropyl Phosphoryl Binding in Dip-Trypsin|journal=[[Journal of the American Chemical Society|J. Am. Chem. Soc.]]|volume=80|issue=5|pages=1260–1|doi=10.1021/ja01538a059|vauthors=Dixon GH, Kauffman DL, Neurath H}}</ref>。キモトリプシンの構造は、1960年代に[[X線回折|X線結晶構造解析]]により解明され、活性部位に触媒三残基が配向していることが明らかになった<ref>{{cite journal|date=1967|title=Three-dimensional structure of tosyl-α-chymotrypsin|journal=[[Nature (journal)|Nature]]|volume=214|issue=5089|pages=652–656|bibcode=1967Natur.214..652M|doi=10.1038/214652a0|pmid=6049071|display-authors=3|vauthors=Matthews BW, Sigler PB, Henderson R, Blow DM}}</ref>。他のプロテアーゼの配列が決定され、整理されて、現在はS1ファミリーと呼ばれる関連するプロテアーゼのファミリーが明らかにされた<ref>{{cite journal|date=1964|title=Trypsinogen and Chymotrypsinogen as Homologous Proteins|journal=[[Proceedings of the National Academy of Sciences of the United States of America|Proc. Natl. Acad. Sci. U.S.A.]]|volume=52|issue=4|pages=884–9|bibcode=1964PNAS...52..884W|doi=10.1073/pnas.52.4.884|pmid=14224394|pmc=300366|vauthors=Walsh KA, Neurath H}}</ref><ref>{{cite journal|date=1975|title=The phylogeny of trypsin-related serine proteases and their zymogens. New methods for the investigation of distant evolutionary relationships|journal=[[Journal of Molecular Biology|J. Mol. Biol.]]|volume=92|issue=2|pages=225–59|doi=10.1016/0022-2836(75)90225-9|pmid=1142424|vauthors=de Haën C, Neurath H, Teller DC}}</ref><ref>{{cite journal|date=1996|title=Conservation and variability in the structures of serine proteinases of the chymotrypsin family|journal=[[Journal of Molecular Biology|J. Mol. Biol.]]|volume=258|issue=3|pages=501–37|doi=10.1006/jmbi.1996.0264|pmid=8642605|vauthors=Lesk AM, Fordham WD}}</ref>。同時に、進化的に無関係な[[パパイン]]および[[サブチリシン]]プロテアーゼの構造は類似の三残基を含んでいることが見いだされた。他の三残基メンバーによる求核剤の活性化のための「電荷リレー」メカニズムは、1960年代後半に提案された<ref>{{cite journal|date=1969|title=Role of a buried acid group in the mechanism of action of chymotrypsin|journal=[[Nature (journal)|Nature]]|volume=221|issue=5178|pages=337–40|bibcode=1969Natur.221..337B|doi=10.1038/221337a0|pmid=5764436|vauthors=Blow DM, Birktoft JJ, Hartley BS}}</ref>。1970年代から80年代にかけて、X線結晶構造解析によってより多くのプロテアーゼの構造が解明され、[[相同]]な三残基 ([[TEVプロテアーゼ]]のような) および[[アナログ|類似]]した三残基 (パパインのような) が発見された<ref>{{cite journal|date=1986|title=Poliovirus-encoded proteinase 3C: a possible evolutionary link between cellular serine and cysteine proteinase families|journal=[[FEBS Letters|FEBS Lett.]]|volume=194|issue=2|pages=253–7|doi=10.1016/0014-5793(86)80095-3|pmid=3000829|vauthors=Gorbalenya AE, Blinov VM, Donchenko AP}}</ref><ref>{{cite journal|date=1988|title=Viral cysteine proteases are homologous to the trypsin-like family of serine proteases: structural and functional implications|journal=[[Proceedings of the National Academy of Sciences of the United States of America|Proc. Natl. Acad. Sci. U.S.A.]]|volume=85|issue=21|pages=7872–6|bibcode=1988PNAS...85.7872B|doi=10.1073/pnas.85.21.7872|pmid=3186696|pmc=282299|vauthors=Bazan JF, Fletterick RJ}}</ref><ref>{{cite journal|date=2002|title=Structural basis for the substrate specificity of tobacco etch virus protease|journal=[[Journal of Biological Chemistry|J. Biol. Chem.]]|volume=277|issue=52|pages=50564–72|doi=10.1074/jbc.M207224200|pmid=12377789|display-authors=3|vauthors=Phan J, Zdanov A, Evdokimov AG, Tropea JE, Peters HK, Kapust RB, Li M, Wlodawer A, Waugh DS}}</ref>。1990年代から2000年代にかけて、プロテアーゼを{{仮リンク|立体構造ホモロジー|en|Structural homology|label=構造的に関連}}した酵素[[スーパーファミリー]]に分類した{{仮リンク|MEROPS|en|MEROPS|label=}}分類システムは、20以上のスーパーファミリーにおける三残基の収斂進化のデータベースとしての役割を果たしている<ref>{{cite journal|date=1993|title=Evolutionary families of peptidases|journal=[[Biochemical Journal|Biochem. J.]]|volume=290|issue=1|pages=205–18|doi=10.1042/bj2900205|pmid=8439290|pmc=1132403|vauthors=Rawlings ND, Barrett AJ}}</ref><ref name="pmid19892822">{{cite journal|date=2010|title=MEROPS: the peptidase database|journal=[[Nucleic Acids Research|Nucleic Acids Res.]]|volume=38|issue=supl_1|pages=D227–33|doi=10.1093/nar/gkp971|pmid=19892822|pmc=2808883|vauthors=Rawlings ND, Barrett AJ, Bateman A}}</ref>。進化に対する化学的制約が、どのようにして同じ三残基の幾何形状に非常に多くの酵素ファミリーの収斂のにつながったのか理解は、2010年代に発展した<ref name="buller1">{{cite journal|vauthors=Buller AR, Townsend CA|date=2013|title=Intrinsic evolutionary constraints on protease structure, enzyme acylation, and the identity of the catalytic triad|journal=[[Proceedings of the National Academy of Sciences of the United States of America|Proc. Natl. Acad. Sci. U.S.A.]]|volume=110|issue=8|pages=E653–61|bibcode=2013PNAS..110E.653B|doi=10.1073/pnas.1221050110|pmc=3581919|pmid=23382230}}</ref>。 |
|||
それらの最初の発見以来、酵素の正確な触媒機構について詳細な研究が増えた。1990年代と2000年代には、[[低障壁水素結合]]が触媒作用に寄与しているのか<ref>{{cite journal|date=1994|title=A low-barrier hydrogen bond in the catalytic triad of serine proteases|journal=[[Science (journal)|Science]]|volume=264|issue=5167|pages=1927–30|bibcode=1994Sci...264.1927F|doi=10.1126/science.7661899|pmid=7661899|vauthors=Frey PA, Whitt SA, Tobin JB}}</ref><ref>{{cite journal|date=1997|title=A low-barrier hydrogen bond in the catalytic triad of serine proteases? Theory versus experiment|journal=[[Science (journal)|Science]]|volume=278|issue=5340|pages=1128–32|bibcode=1997Sci...278.1128A|doi=10.1126/science.278.5340.1128|pmid=9353195|display-authors=3|vauthors=Ash EL, Sudmeier JL, De Fabo EC, Bachovchin WW}}</ref><ref>{{cite journal|date=2018|title=Direct evidence of a low barrier hydrogen bond in the catalytic triad of a Serine protease|journal=[[Scientific Reports|Sci. Rep.]]|volume=8|issue=1|pages=10078|bibcode=2018NatSR...810078A|doi=10.1038/s41598-018-28441-7|pmid=29973622|pmc=6031666|vauthors=Agback P, Agback T}}</ref>、それとも通常の[[水素結合]]で機構を説明するのに十分なのか<ref name=":0">{{cite journal|date=1996|title=Energy considerations show that low-barrier hydrogen bonds do not offer a catalytic advantage over ordinary hydrogen bonds|journal=[[Proceedings of the National Academy of Sciences of the United States of America|Proc. Natl. Acad. Sci. U.S.A.]]|volume=93|issue=24|pages=13665–70|bibcode=1996PNAS...9313665W|doi=10.1073/pnas.93.24.13665|pmid=8942991|pmc=19385|vauthors=Warshel A, Papazyan A}}</ref>、ということが特に議論された<ref name=":8">{{cite journal|date=2004|title=The low barrier hydrogen bond (LBHB) proposal revisited: the case of the Asp... His pair in serine proteases|journal=[[Proteins (journal)|Proteins]]|volume=55|issue=3|pages=711–23|doi=10.1002/prot.20096|pmid=15103633|vauthors=Schutz CN, Warshel A}}</ref>。触媒三残基が使用する電荷リレー型共有結合触媒に関する膨大な研究の結果、生化学の中で最も特徴的な機構が解明された<ref name="Perutz 1992" /><ref name="cite journal|pmid=7849591" /><ref name=":0" />。 |
|||
== 機能<!-- Function --> == |
|||
触媒三残基を含む酵素は、基質を分割する ([[加水分解]]酵素) か、基質の一部分を第二の基質に移す ([[転移酵素]]) かの2つの反応タイプのいずれかに三残基を使用する。三残基は、酵素の[[活性部位]]にある相互に依存する残基の集合であり、他の残基 (例えば、[[結合部位]]や{{仮リンク|オキシアニオンホール|en|Oxyanion hole|label=}}) と協働して求核的触媒作用{{Enlink|Nucleophilic catalysis|英語版}}を達成するために作用する。これらの三残基が一緒に作用して[[求核剤]]メンバーの{{仮リンク|反応性 (化学)|en|Reactivity (chemistry)|label=反応性}}を高め、基質との共有結合中間体を生成し、それが分解されて触媒反応が完了する。 |
|||
=== 機構 <!-- Mechanism -->=== |
|||
触媒三残基は、残基を求核剤として用いて{{仮リンク|共有結合触媒|en|Covalent catalysis|label=}}反応を行う。求核性残基の反応性は、他の三残基メンバーの[[官能基]]によって増加する。求核性残基は、それ自体が酸によって結合および安定化されている塩基により、分極および配向される。 |
|||
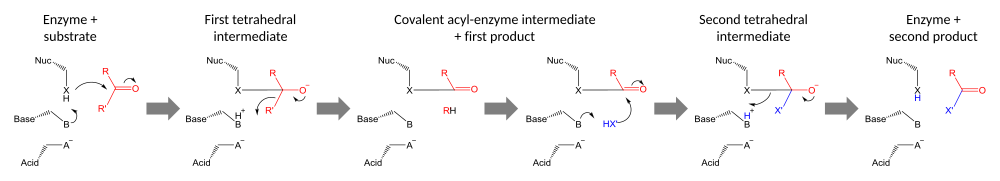
触媒反応は2段階で行われる。まず、活性化された求核剤が[[カルボニル基|カルボニル]]炭素を攻撃し、カルボニル酸素に電子を受け入れさせ、四面体[[反応中間体|中間体]]を形成する。この中間体への負電荷の蓄積は、通常、活性部位内の{{仮リンク|オキシアニオンホール|en|Oxyanion hole|label=}}によって安定化される。次に、中間体は崩壊してカルボニルに戻り、基質の前半を放出し、後半は{{仮リンク|アシル酵素中間体|en|Acyl-enzyme intermediate|label=}}として酵素に共有結合したままになる。この最初の[[脱離基]]の放出は、多くの場合、塩基によるプロトンの供与によって促進される。 |
|||
触媒反応の第二段階は、第二の基質の攻撃によるアシル酵素中間体の分解である。この基質が水であれば加水分解が行われ、有機分子であればその分子が第一の基質に移動する。この第二の基質による攻撃は、新しい四面体中間体を形成し、酵素の求核剤を放出し、第二の生成物を放出し、遊離酵素を再生することによって分解する。<ref>{{Cite thesis|last=Shafee|first=Thomas|name-list-style=vanc|title=Evolvability of a viral protease: experimental evolution of catalysis, robustness and specificity|date=2014|degree=PhD|url=https://www.repository.cam.ac.uk/handle/1810/245207|doi=10.17863/CAM.16528|publisher=University of Cambridge}}</ref>。 |
|||
{{wide image|Triad mechanism.svg|1000px|触媒三残基 (黒) によって触媒される一般的な反応機構:第二の基質 (青) による[[カルボニル基|カルボニル]]基質 (赤) での[[求核置換反応|求核置換]]。まず酵素の求核剤 (X) がカルボニル基質を攻撃して、共有結合したアシル酵素中間体を形成する。次にこの中間体は、第二の基質の求核剤 (X') によって攻撃される。第二の求核剤が水のヒドロキシルである場合、結果は加水分解であり、そうでない場合、結果はX'の[[:en:Group transfer reaction|基移動(英語版)]]である。}} |
|||
== 触媒三残基の例<!-- Examples of triads -->== |
|||
| ⚫ | |||
例えば[[キモトリプシン]]の場合は、195番目のセリン側鎖が57番目のヒスチジンの[[イミダゾール環]]に近接し、さらにイミダゾール環の-NH基が102番のアスパラギン酸の[[カルボキシ基]]に近接している。 |
例えば[[キモトリプシン]]の場合は、195番目のセリン側鎖が57番目のヒスチジンの[[イミダゾール環]]に近接し、さらにイミダゾール環の-NH基が102番のアスパラギン酸の[[カルボキシ基]]に近接している。 |
||
{{節スタブ}} |
|||
== 脚注<!-- References -->== |
|||
{{脚注ヘルプ}} |
|||
{{Reflist}} |
|||
== 関連項目 == |
|||
*[[活性部位]] |
|||
*[[収斂進化]] |
|||
*{{仮リンク|分岐進化|en|Divergent evolution|label=}} |
|||
*[[酵素反応]] |
|||
*{{仮リンク|酵素スーパーファミリー|en|Enzyme superfamily|label=}} |
|||
*[[基]] |
|||
*{{仮リンク|PA clan|en|PA clan|label=}} |
|||
*[[プロテアーゼ]] |
|||
*[[タンパク質分解]] |
|||
== 外部リンク == |
|||
{{節スタブ}} |
|||
{{Biosci-stub}} |
|||
{{DEFAULTSORT:しよくはいさんさんき}} |
{{DEFAULTSORT:しよくはいさんさんき}} |
||
2021年1月3日 (日) 10:47時点における版

触媒三残基(しょくばいさんざんき、英: Catalytic triad)とは、いくつかの酵素の活性部位に見られる3つの配位アミノ酸のセットである[1][2]。触媒三残基は、ヒドロラーゼおよびトランスフェラーゼ酵素 (例えば、プロテアーゼ、アミダーゼ、エステラーゼ、アシルラーゼ、リパーゼおよびβ-ラクタマーゼ) で最も一般的に見られる。酸-塩基-求核剤 三残基は、共有結合触媒のための求核性残基を生成するための一般的なモチーフである。この残基は電荷リレーネットワークを形成して求核剤を分極および活性化し、基質を攻撃して共有結合中間体を形成し、加水分解されて生成物を放出し、遊離酵素を再生する。求核剤は、最も一般的にはセリンやシステインアミノ酸であるが、時にはスレオニンまたはセレノシステインも含まれている。酵素の三次元構造は、配列上 (一次構造) では離れていても、三残基が正確な方向にまとまっていることを示している[3]。
機能 (および三残基の求核剤) の分岐進化と同様に、触媒三残基は収斂進化の最良の例のいくつかを示している。触媒作用に対する化学的制約により、同じ触媒解明が、少なくとも23の別々のスーパーファミリーで独立して展開している。その作用機序は、生化学的に最もよく研究されているものの一つである[4][5]。
歴史
トリプシンとキモトリプシンは、1930年代に初めて精製された酵素である[6]。1950年代には、トリプシンとキモトリプシンのそれぞれのセリンが触媒的求核剤として同定された (ジイソプロピルフルオロリン酸修飾)[7]。キモトリプシンの構造は、1960年代にX線結晶構造解析により解明され、活性部位に触媒三残基が配向していることが明らかになった[8]。他のプロテアーゼの配列が決定され、整理されて、現在はS1ファミリーと呼ばれる関連するプロテアーゼのファミリーが明らかにされた[9][10][11]。同時に、進化的に無関係なパパインおよびサブチリシンプロテアーゼの構造は類似の三残基を含んでいることが見いだされた。他の三残基メンバーによる求核剤の活性化のための「電荷リレー」メカニズムは、1960年代後半に提案された[12]。1970年代から80年代にかけて、X線結晶構造解析によってより多くのプロテアーゼの構造が解明され、相同な三残基 (TEVプロテアーゼのような) および類似した三残基 (パパインのような) が発見された[13][14][15]。1990年代から2000年代にかけて、プロテアーゼを構造的に関連した酵素スーパーファミリーに分類したMEROPS分類システムは、20以上のスーパーファミリーにおける三残基の収斂進化のデータベースとしての役割を果たしている[16][17]。進化に対する化学的制約が、どのようにして同じ三残基の幾何形状に非常に多くの酵素ファミリーの収斂のにつながったのか理解は、2010年代に発展した[2]。
それらの最初の発見以来、酵素の正確な触媒機構について詳細な研究が増えた。1990年代と2000年代には、低障壁水素結合が触媒作用に寄与しているのか[18][19][20]、それとも通常の水素結合で機構を説明するのに十分なのか[21]、ということが特に議論された[22]。触媒三残基が使用する電荷リレー型共有結合触媒に関する膨大な研究の結果、生化学の中で最も特徴的な機構が解明された[4][5][21]。
機能
触媒三残基を含む酵素は、基質を分割する (加水分解酵素) か、基質の一部分を第二の基質に移す (転移酵素) かの2つの反応タイプのいずれかに三残基を使用する。三残基は、酵素の活性部位にある相互に依存する残基の集合であり、他の残基 (例えば、結合部位やオキシアニオンホール) と協働して求核的触媒作用 (英語版) を達成するために作用する。これらの三残基が一緒に作用して求核剤メンバーの反応性を高め、基質との共有結合中間体を生成し、それが分解されて触媒反応が完了する。
機構
触媒三残基は、残基を求核剤として用いて共有結合触媒反応を行う。求核性残基の反応性は、他の三残基メンバーの官能基によって増加する。求核性残基は、それ自体が酸によって結合および安定化されている塩基により、分極および配向される。
触媒反応は2段階で行われる。まず、活性化された求核剤がカルボニル炭素を攻撃し、カルボニル酸素に電子を受け入れさせ、四面体中間体を形成する。この中間体への負電荷の蓄積は、通常、活性部位内のオキシアニオンホールによって安定化される。次に、中間体は崩壊してカルボニルに戻り、基質の前半を放出し、後半はアシル酵素中間体として酵素に共有結合したままになる。この最初の脱離基の放出は、多くの場合、塩基によるプロトンの供与によって促進される。
触媒反応の第二段階は、第二の基質の攻撃によるアシル酵素中間体の分解である。この基質が水であれば加水分解が行われ、有機分子であればその分子が第一の基質に移動する。この第二の基質による攻撃は、新しい四面体中間体を形成し、酵素の求核剤を放出し、第二の生成物を放出し、遊離酵素を再生することによって分解する。[23]。
触媒三残基の例

例えばキモトリプシンの場合は、195番目のセリン側鎖が57番目のヒスチジンのイミダゾール環に近接し、さらにイミダゾール環の-NH基が102番のアスパラギン酸のカルボキシ基に近接している。
この節の加筆が望まれています。 |
脚注
- ^ “Catalytic triads and their relatives”. Trends Biochem. Sci. 23 (9): 347–52. (1998). doi:10.1016/S0968-0004(98)01254-7. PMID 9787641.
- ^ a b “Intrinsic evolutionary constraints on protease structure, enzyme acylation, and the identity of the catalytic triad”. Proc. Natl. Acad. Sci. U.S.A. 110 (8): E653–61. (2013). Bibcode: 2013PNAS..110E.653B. doi:10.1073/pnas.1221050110. PMC 3581919. PMID 23382230.
- ^ “9 Catalytic Strategies”. Biochemistry (5th ed.). San Francisco: W.H. Freeman. (2002). ISBN 9780716749554
- ^ a b Perutz, Max (1992). Protein structure. New approaches to disease and therapy. New York: W.H. Freeman and Co. ISBN 9780716770213
- ^ a b “Proteolytic enzymes past and present: the second golden era. Recollections, special section in honor of Max Perutz”. Protein Sci. 3 (10): 1734–9. (1994). doi:10.1002/pro.5560031013. PMC 2142620. PMID 7849591.
- ^ “Endothelin-induced vasoconstriction and release of atrial natriuretic peptides in the rat”. Acta Physiol. Scand. 138 (4): 549–56. (1990). doi:10.1111/j.1748-1716.1990.tb08883.x. PMID 2141214.
- ^ “Amino Acid Sequence in the Region of Diisopropyl Phosphoryl Binding in Dip-Trypsin”. J. Am. Chem. Soc. 80 (5): 1260–1. (1958). doi:10.1021/ja01538a059.
- ^ “Three-dimensional structure of tosyl-α-chymotrypsin”. Nature 214 (5089): 652–656. (1967). Bibcode: 1967Natur.214..652M. doi:10.1038/214652a0. PMID 6049071.
- ^ “Trypsinogen and Chymotrypsinogen as Homologous Proteins”. Proc. Natl. Acad. Sci. U.S.A. 52 (4): 884–9. (1964). Bibcode: 1964PNAS...52..884W. doi:10.1073/pnas.52.4.884. PMC 300366. PMID 14224394.
- ^ “The phylogeny of trypsin-related serine proteases and their zymogens. New methods for the investigation of distant evolutionary relationships”. J. Mol. Biol. 92 (2): 225–59. (1975). doi:10.1016/0022-2836(75)90225-9. PMID 1142424.
- ^ “Conservation and variability in the structures of serine proteinases of the chymotrypsin family”. J. Mol. Biol. 258 (3): 501–37. (1996). doi:10.1006/jmbi.1996.0264. PMID 8642605.
- ^ “Role of a buried acid group in the mechanism of action of chymotrypsin”. Nature 221 (5178): 337–40. (1969). Bibcode: 1969Natur.221..337B. doi:10.1038/221337a0. PMID 5764436.
- ^ “Poliovirus-encoded proteinase 3C: a possible evolutionary link between cellular serine and cysteine proteinase families”. FEBS Lett. 194 (2): 253–7. (1986). doi:10.1016/0014-5793(86)80095-3. PMID 3000829.
- ^ “Viral cysteine proteases are homologous to the trypsin-like family of serine proteases: structural and functional implications”. Proc. Natl. Acad. Sci. U.S.A. 85 (21): 7872–6. (1988). Bibcode: 1988PNAS...85.7872B. doi:10.1073/pnas.85.21.7872. PMC 282299. PMID 3186696.
- ^ “Structural basis for the substrate specificity of tobacco etch virus protease”. J. Biol. Chem. 277 (52): 50564–72. (2002). doi:10.1074/jbc.M207224200. PMID 12377789.
- ^ “Evolutionary families of peptidases”. Biochem. J. 290 (1): 205–18. (1993). doi:10.1042/bj2900205. PMC 1132403. PMID 8439290.
- ^ “MEROPS: the peptidase database”. Nucleic Acids Res. 38 (supl_1): D227–33. (2010). doi:10.1093/nar/gkp971. PMC 2808883. PMID 19892822.
- ^ “A low-barrier hydrogen bond in the catalytic triad of serine proteases”. Science 264 (5167): 1927–30. (1994). Bibcode: 1994Sci...264.1927F. doi:10.1126/science.7661899. PMID 7661899.
- ^ “A low-barrier hydrogen bond in the catalytic triad of serine proteases? Theory versus experiment”. Science 278 (5340): 1128–32. (1997). Bibcode: 1997Sci...278.1128A. doi:10.1126/science.278.5340.1128. PMID 9353195.
- ^ “Direct evidence of a low barrier hydrogen bond in the catalytic triad of a Serine protease”. Sci. Rep. 8 (1): 10078. (2018). Bibcode: 2018NatSR...810078A. doi:10.1038/s41598-018-28441-7. PMC 6031666. PMID 29973622.
- ^ a b “Energy considerations show that low-barrier hydrogen bonds do not offer a catalytic advantage over ordinary hydrogen bonds”. Proc. Natl. Acad. Sci. U.S.A. 93 (24): 13665–70. (1996). Bibcode: 1996PNAS...9313665W. doi:10.1073/pnas.93.24.13665. PMC 19385. PMID 8942991.
- ^ “The low barrier hydrogen bond (LBHB) proposal revisited: the case of the Asp... His pair in serine proteases”. Proteins 55 (3): 711–23. (2004). doi:10.1002/prot.20096. PMID 15103633.
- ^ Shafee T (2014). Evolvability of a viral protease: experimental evolution of catalysis, robustness and specificity (PhD thesis). University of Cambridge. doi:10.17863/CAM.16528。
関連項目
外部リンク
この節の加筆が望まれています。 |