「酵素反応速度論」の版間の差分
翻訳 from en:Enzyme Kinetics 17:30, 4 August 2008 UTC, 後半の翻訳は近日中にやります。画像の移行をどなたかお願いできないでしょうか。 |
(相違点なし)
|
2008年8月13日 (水) 14:17時点における版

酵素反応速度論 とは、酵素によって触媒される化学反応を、反応速度の面から研究する学問である。酵素の反応速度論を研究することで、酵素反応の機構、代謝における役割、活性調節の仕組み、薬物や毒が酵素をどう阻害するかといったことを明らかにできる。
酵素は、通常、蛋白質分子であり、他の分子(酵素の基質という)に作用する。基質は、酵素の活性部位に結合し、段階的に生成物へと変化を遂げる。この過程は、反応機構と呼ばれる。反応機構は、単一基質機構と、複数基質機構に分類できる。
一つの基質としか結合しない酵素、例えばトリオースリン酸イソメラーゼの研究では、酵素が基質と結合する際の解離定数や、回転率の測定を目指す。
酵素が複数の基質と結合する場合、例えばジヒドロ葉酸還元酵素 (右図)では、基質が結合し、生成物が解離する順序を明らかにすることもできる。1つの基質と結合し、複数の生成物を放出する酵素の例として、プロテアーゼ が挙げられる。この酵素は、1つの基質蛋白質を切断して、2つのポリペプチドにする。2つの基質を1つに結合する酵素もある。DNAポリメラーゼは、ヌクレオチドをDNAに結合する。これらの酵素の反応機構は、複雑で何段階にも及ぶことが多いが、通常、律速段階があって、これが全体の反応速度を決定する。律速段階は、化学反応であったり、(生成物が酵素から離れる際など)酵素や基質のコンフォメーションの変化であったりする。
酵素の立体構造が分かると、速度論的な情報の解釈に有利である。例えば、構造から、触媒過程で基質や生成物がどのように結合しているか、反応中にどう変化するかが分かる。また、特定のアミノ酸残基が反応機構で果たす役割が分かることもある。酵素によっては、反応中に構造が大きく変化するものがあるが、このような場合は、酵素の構造を、触媒を受けない基質類似体が結合した場合と、結合していない場合それぞれで決定しておくとよい。
生体で触媒として働くのは、蛋白質でできた酵素だけではない。RNAによる触媒、つまりリボソームのようなリボザイムは、RNAスプライシングや翻訳といった多くの過程で不可欠である。リボザイムと酵素の主要な違いは、RNAのほうが触媒できる反応が限られているということだ。もっとも、RNAによる触媒の機構も、蛋白質酵素の場合と同じ方法で解析し、分類することができる。
総論

酵素によって触媒される反応でも、その反応物と生成物は、触媒を受けていない場合と変わらない。他の触媒と同じく、酵素も、反応物と生成物の間の化学平衡を動かすことはない。[1] ただし、触媒されていない反応と違って、酵素触媒下の反応は、反応速度に飽和点を持つ。ある酵素濃度のもとで、基質濃度が比較的低いとすると、反応速度は基質濃度に比例して上昇する。酵素分子は、自由に反応を触媒でき、基質濃度が上がれば、基質と酵素が出会う頻度も上昇するからだ。ところが、基質濃度がある程度高くなると、反応速度は、理論的上限値に漸近する。酵素の活性部位は、ほとんど全て基質と結合してしまい、反応速度は酵素固有の回転率によって決まるようになる。この両極端のちょうど中間にあたる基質濃度は、KMで表される。
酵素の速度論的特徴の中で特に重要なのは、特定の基質に対して酵素が飽和するのはいつかということと、その時の最大反応速度がどれだけかということである。これらの特徴から、酵素が細胞の中でどんな働きをしており、条件が変化したときに酵素がどう反応するかを推察できる。
酵素の試験

酵素の試験(アッセイ)とは、実験室で酵素反応の速度を測ることである。酵素は、反応を触媒する過程で消費されることはないため、実際に測るのは、基質か生成物の濃度変化である。測定方法にはいろいろある。紫外・可視・近赤外分光法では、生成物と反応物の吸光度の違いを測る。放射能分析では、生成物が徐々にできてくるのを、放射性元素の取り込みや放出によって測る。分光法は、反応速度を連続的に測定できるので便利である。放射能分析では、サンプルを取り出して測定する必要がある(非連続的分析である)が、多くの場合、非常に鋭敏で、ごくわずかな酵素活性でも測定できる。[2] よく似た方法として、質量分析器を使う手もあり、基質が反応物へ変化する過程で、安定同位体が取り込まれたり放出されるのを観測する。
最も敏感な酵素測定法は、レーザを使う方法である。顕微鏡下で、酵素1分子にレーザの焦点を合わせ、反応を触媒する過程での変化を観察する。反応中に補因子の蛍光が変化するのを測定したり、蛋白質の一部分を蛍光色素でラベルして、触媒中の動きを調べる。[3] こうした実験から、酵素単一分子の速度論やダイナミクス(反応中の動き)について、新しい知見が得られつつある。従来の方法は、数百万にも及ぶ多数の酵素分子の挙動を平均的したものを観察していた。[4][5]
酵素測定における典型的なグラフが左に示してある。反応開始時点では、初期速度にしたがって、線形に(時間に比例して)生成物ができる。時間が経つ(グラフの右)と、反応速度は低下する。基質が消費され、生成物が蓄積するからだ。初期速度で反応が進む時間の長さは、測定条件によって変わり、数ミリ秒のこともあれば、数時間に及ぶこともある。通常の測定では、この期間が約1分になるようにしておくと、実験が容易である。ただし、液体を急速に混ぜ合わせる装置を使えば、初期速度の段階が1秒に満たない反応でも測定できる。これを、ストップフロー法という。[6] これらの高速な測定技術は、後述するように、定常状態に達する前の速度を調べるのに不可欠である。
酵素反応速度論の実験では、反応のこの初期部分、つまり、生成物が時間に比例してできてくる線形部分に注目することが多い。しかし、反応全体を測定して、そのデータを非線形の速度方程式に当てはめることもできる。[7] このような測定を、progress-curve 解析という。この方法は、初期速度が速すぎて正確に測定できないときに有効である。
単一基質反応
単一基質機構を持つ酵素としては、トリオースリン酸イソメラーゼや二ホスホグリセリン酸ムターゼのようなイソメラーゼや、アデニル酸サイクラーゼやRNAリアーゼであるハンマーヘッドリボザイムのような、分子内リアーゼがある。ただし、基質が一つしかない酵素でも、反応機構は単一基質機構でないことがある。カタラーゼがその例で、まず基質である過酸化水素の1つ目の分子と反応して酵素自身が酸化され、続いて2つ目の分子によって還元される。確かに基質は1種類だが、修飾された酵素が中間体として存在している以上、カタラーゼの反応機構は、ピンポン機構というべきである。これについては、複数基質反応として、後述する。
ミカエリス-メンテン速度論

酵素によって触媒される反応は飽和を示すので、反応速度は、基質を増やしてもそれに比例して線形に増えるわけではない。反応の初期速度を、基質濃度([S]で表す)を変えながら測定すると、反応速度(v)は、右に示すように、[S] にあわせて上昇する。しかし、[S] がさらに増えると、酵素は基質で飽和し、反応速度は、酵素の最大反応速度 Vmaxに達する。
単一基質反応における、ミカエリス-メンテンのモデルを右に示した。まず、酵素 E と基質 S が反応して、酵素基質複合体 ES を作る二分子反応が起こる。単分子反応 で表される触媒反応の機構は、実際には複雑かもしれないが、通常、1つの律速段階があって、触媒作用を、速度定数k2を持つ1つの触媒反応で表現できる。
- (式 1).
k2は、kcatや回転数とも呼ばれ、酵素が1秒に行える反応回数の上限を示す。
基質濃度 [S] が小さい場合、酵素は、遊離型 E と基質酵素複合体 ES の平衡状態にある。[S] を増やすと、[E] が減って [ES] が増え、平衡が右に傾く。反応速度は [ES] によって決まるため、[S] のわずかな変化でも反応速度が大きく変わる。しかし、[S] が非常に大きくなると、酵素は完全に基質で飽和し、全て基質酵素複合体 ES となる。こうなると、反応速度(v≈k2[E]tot=Vmax)は、[S]が少し変化したくらいでは変わらない。ここで、, [E]totは、酵素の全濃度であり、
飽和条件での [ES] 濃度にほぼ等しい。
ミカエリス-メンテンの式 [8] は、反応速度 v が、酵素基質結合の平衡や速度定数 k2 とどう関係するかを示す式である。レオノール・ミカエリスとモード・レオノーラ・メンテン は、 k2 が k-1 よりもずっと小さいとき(平衡の仮定)、次の式を導いた。[9]
- (式 2)
この式は、単一基質機構をもつ酵素ほとんどの速度論の基礎となる。
ミカエリス定数 Km は、酵素の反応速度が Vmax の半分になるときの基質濃度として定義されている。このことは、ミカエリス-メンテン式で、[S] = Km を代入してみれば確認できる。反応の律速段階が、基質の解離よりもずっと遅い場合(k2 << k-1)、ミカエリス定数 Km は、複合体 ES の解離係数に概ね等しい。ただし、このような状況は比較的まれである。
多くの状況では、k2 > k-1 であり、Briggs-Haldane 状態と呼ばれる。[10] 定常状態近似から分かるように、ミカエリス-メンテン式は、これらの状況でも成立する。初期速度の段階では、反応速度はほとんど一定であり、[ES] も変化しないことが分かる(式1)。
よって、[ES] は、下の式であたえられる。
ここで、ミカエリス定数 Km の定義は、
([E] は遊離の酵素濃度)。まとめると、反応速度 v の一般式は、再びミカエリス-メンテン式に戻る。
特異度定数 は、酵素が基質を生成物に変換する際の効率を示す。ミカエリス定数の定義から、ミカエリス-メンテンの式は、次のような形にも書ける。
ここで、[E] は遊離の酵素濃度である。つまり、特異度定数とは、遊離の酵素が遊離の基質と結合して生成物をつくる際の、実質的な2次の速度式である。特異度定数は、溶液中で酵素と基質が出会う頻度によって制限されるが、その値は1010 M−1 s−1にも及ぶ。[11] 驚くべきことだが、この最大速度は、基質や酵素の大きさとはほとんど関係ない。[12] 2つの基質があったとき、その特異度係数の違いは、酵素がそれぞれの基質を変換する際の効率の違いを定量的に表す。基質濃度 [S] が小さいとき([S] << Km)、ミカエリス-メンテン式のグラフの傾きから、特異度定数を知ることもできる。
ミカエリス-メンテン式の線形プロット
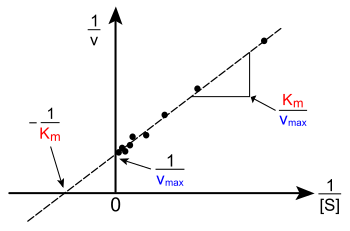
バージニア大学の、ミカエリス-メンテン速度論のチュートリアル(英語)で [α]、速度論的パラメータが変わったときの酵素の挙動を試してみることができる。
v と [S] を縦横に取ってグラフを描くと、直線にはならない。[S] が小さいうちは線形だが、[S] が大きい部分では飽和し、グラフが曲がってくるのだ。コンピュータで非線形回帰分析ができるようになる前は、この非線形性のせいで、Km と Vmax を正確に読み取ることは難しかった。そのため、Lineweaver-Burk プロット、Eadie-Hofstee 図、 Hanes-Woolf プロット といった線形化手法が編みだされた。これらの表現法は、データを表示するには有用だが、速度係数を求めるためにはもはや使われていない。非線形回帰によってより正確に係数を求めるソフトウェアがあるためだ。[13]
Lineweaver-Burk プロット、別名二重逆数プロットは、速度論的データを示す際によく使われる。これは、ミカエリス-メンテン式の両辺の逆数を取ると得られる。右に示したように、ミカエリス-メンテン式の線形化であり、y = mx + c 型の直線となる。y 切片は 1/Vmax に等しく、x切片は、 -1/Km を表す。
もちろん、 1/[S] が負となるような部分の数値を測定することはできない。1/[S] = 0 である下限(y切片)は、基質濃度無限大にあたり、右に示すように 1/v=1/Vmax である。また、x切片は、正の濃度における実験データから外挿したものである。しかも、Lineweaver-Burk プロットは、基質濃度が低い条件で測定したデータを過大評価しており、 Vmax と Km を不正確にする恐れがある。[14] より正確な線形プロットは、Eadie-Hofstee 図である。この図では、v を、v/[S] に対してプロットする。3つ目の線形プロットは、Hanes-Woolf プロットであり、[S]/v を [S] に対してプロットする。どの場合でも、データを正規化することで、実験の量を減らし、結果の信頼性を改善することができるし、視覚的または数値的な解析にも適している。[15]
速度定数の実際的な意義
酵素の速度論を研究することは、二つの基本的な理由から重要である。まず、酵素がどう働くかを考えるのに役立つ。また、生体内で酵素が示す挙動を予測するのにも役立つ。上のように定義された速度定数 Km と Vmax は、複数の酵素が共同して代謝を制御する仕組みを理解する上で不可欠である。
このような制御を理解することは、単純な系においてさえ、容易ではない。例えば、オキサロ酢酸は、ミトコンドリアの中でリンゴ酸デヒドロゲナーゼによって生成される。その後、リンゴ酸シンターゼ、ホスホエノールピルビン酸カルボキシラーゼ、アスパラギン酸アミノトランスフェラーゼなどによって消費され、それぞれ、クエン酸回路、糖新生、アスパラギン酸生合成へと進む。それぞれの経路にどれだけのオキサロ酢酸が進むかを予測するには、オキサロ酢酸の濃度だけでなく、各酵素の濃度と速度論的係数の情報が不可欠である。代謝経路の動態を予想することの究極の目的は、酵素の速度論と遺伝子発現の莫大なデータを、1個体全体の数学的モデルとしてまとめあげることにある。この目標は、真核生物に関しては、まだまだ先が長いが、バクテリアに関しては、既にEscherichia coliのモデルが作られて試験中である。[16][17]
Multi-substrate reactions
Multi-substrate reactions follow complex rate equations that describe how the substrates bind and in what sequence. The analysis of these reactions is much simpler if the concentration of substrate A is kept constant and substrate B varied. Under these conditions, the enzyme behaves just like a single-substrate enzyme and a plot of v by [S] gives apparent Km and Vmax constants for substrate B. If a set of these measurements is performed at different fixed concentrations of A, these data can be used to work out what the mechanism of the reaction is. For an enzyme that takes two substrates A and B and turns them into two products P and Q, there are two types of mechanism: ternary complex and ping–pong.
Ternary-complex mechanisms

In these enzymes, both substrates bind to the enzyme at the same time to produce an EAB ternary complex. The order of binding can either be random (in a random mechanism) or substrates have to bind in a particular sequence (in an ordered mechanism). When a set of v by [S] curves (fixed A, varying B) from an enzyme with a ternary-complex mechanism are plotted in a Lineweaver–Burk plot, the set of lines produced will intersect.
Enzymes with ternary-complex mechanisms include glutathione S-transferase,[18] dihydrofolate reductase[19] and DNA polymerase.[20] The following links show short animations of the ternary-complex mechanisms of the enzymes dihydrofolate reductase[β] and DNA polymerase[γ].
Ping–pong mechanisms

As shown on the right, enzymes with a ping-pong mechanism can exist in two states, E and a chemically modified form of the enzyme E*; this modified enzyme is known as an intermediate. In such mechanisms, substrate A binds, changes the enzyme to E* by, for example, transferring a chemical group to the active site, and is then released. Only after the first substrate is released can substrate B bind and react with the modified enzyme, regenerating the unmodified E form. When a set of v by [S] curves (fixed A, varying B) from an enzyme with a ping–pong mechanism are plotted in a Lineweaver–Burk plot, a set of parallel lines will be produced.
Enzymes with ping–pong mechanisms include some oxidoreductases such as thioredoxin peroxidase,[21] transferases such as acylneuraminate cytydilyltransferase[22] and serine proteases such as trypsin and chymotrypsin.[23] Serine proteases are a very common and diverse family of enzymes, including digestive enzymes (trypsin, chymotrypsin, and elastase), several enzymes of the blood clotting cascade and many others. In these serine proteases, the E* intermediate is an acyl-enzyme species formed by the attack of an active site serine residue on a peptide bond in a protein substrate. A short animation showing the mechanism of chymotrypsin is linked here.[δ]
Non-Michaelis–Menten kinetics

Some enzymes produce a sigmoid v by [S] plot, which often indicates cooperative binding of substrate to the active site. This means that the binding of one substrate molecule affects the binding of subsequent substrate molecules. This behaviour is most common in multimeric enzymes with several interacting active sites.[24] Here, the mechanism of cooperation is similar to that of haemoglobin, with binding of substrate to one active site altering the affinity of the other active sites for substrate molecules. Positive cooperativity occurs when binding of the first substrate molecule increases the affinity of the other active sites for substrate. Negative cooperativity occurs when binding of the first substrate decreases the affinity of the enzyme for other substrate molecules.
Allosteric enzymes include mammalian tyrosyl tRNA-synthetase, which shows negative cooperativity,[25] and bacterial aspartate transcarbamoylase[26] and phosphofructokinase[27], which show positive cooperativity.
Cooperativity is surprisingly common and can help regulate the responses of enzymes to changes in the concentrations of their substrates. Positive cooperativity makes enzymes much more sensitive to [S] and their activities can show large changes over a narrow range of substrate concentration. Conversely, negative cooperativity makes enzymes insensitive to small changes in [S].
The Hill equation[28] is often used to describe the degree of cooperativity quantitatively in non-Michaelis–Menten kinetics. The derived Hill coefficient n measures how much the binding of substrate to one active site affects the binding of substrate to the other active sites. A Hill coefficient of <1 indicates negative cooperativity and a coefficient of >1 indicates positive cooperativity.
Pre-steady-state kinetics
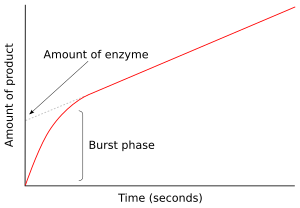
In the first moment after an enzyme is mixed with substrate, no product has been formed and no intermediates exist. The study of the next few milliseconds of the reaction is called pre-steady-state kinetics. Pre-steady-state kinetics is therefore concerned with the formation and consumption of enzyme–substrate intermediates (such as ES or E*) until their steady-state concentrations are reached.
This approach was first applied to the hydrolysis reaction catalysed by chymotrypsin.[29] Often, the detection of an intermediate is a vital piece of evidence in investigations of what mechanism an enzyme follows. For example, in the ping–pong mechanisms that are shown above, rapid kinetic measurements can follow the release of product P and measure the formation of the modified enzyme intermediate E*.[30] In the case of chymotrypsin, this intermediate is formed by the attack of the substrate by the nucleophilic serine in the active site and the formation of the acyl-enzyme intermediate.
In the figure to the right, the enzyme produces E* rapidly in the first few seconds of the reaction. The rate then slows as steady state is reached. This rapid burst phase of the reaction measures a single turnover of the enzyme. Consequently, the amount of product released in this burst, shown as the intercept on the y-axis of the graph, also gives the amount of functional enzyme which is present in the assay.[31]
Chemical mechanism
An important goal of measuring enzyme kinetics is to determine the chemical mechanism of an enzyme reaction, i.e., the sequence of chemical steps that transform substrate into product. The kinetic approaches discussed above will show at what rates intermediates are formed and inter-converted, but they cannot identify exactly what these intermediates are.
Kinetic measurements taken under various solution conditions or on slightly modified enzymes or substrates often shed light on this chemical mechanism, as they reveal the rate-determining step or intermediates in the reaction. For example, the breaking of a covalent bond to a hydrogen atom is a common rate-determining step. Which of the possible hydrogen transfers is rate determining can be shown by measuring the kinetic effects of substituting each hydrogen by deuterium, its stable isotope. The rate will change when the critical hydrogen is replaced, due to a primary kinetic isotope effect, which occurs because bonds to deuterium are harder to break then bonds to hydrogen.[32] It is also possible to measure similar effects with other isotope substitutions, such as 13C/12C and 18O/16O, but these effects are more subtle.[33]
Isotopes can also be used to reveal the fate of various parts of the substrate molecules in the final products. For example, it is sometimes difficult to discern the origin of an oxygen atom in the final product; since it may have come from water or from part of the substrate. This may be determined by systematically substituting oxygen's stable isotope 18O into the various molecules that participate in the reaction and checking for the isotope in the product.[34] The chemical mechanism can also be elucidated by examining the kinetics and isotope effects under different pH conditions,[35] by altering the metal ions or other bound cofactors,[36] by site-directed mutagenesis of conserved amino acid residues, or by studying the behaviour of the enzyme in the presence of analogues of the substrate(s).[37]
Enzyme inhibition

Enzyme inhibitors are molecules that reduce or abolish enzyme activity. These are either reversible (i.e., removal of the inhibitor restores enzyme activity) or irreversible (i.e., the inhibitor permanently inactivates the enzyme).
Reversible inhibitors
Reversible enzyme inhibitors can be classified as competitive, uncompetitive, non-competitive or mixed, according to their effects on Km and Vmax. These different effects result from the inhibitor binding to the enzyme E, to the enzyme–substrate complex ES, or to both, as shown in the figure to the right and the table below. The particular type of an inhibitor can be discerned by studying the enzyme kinetics as a function of the inhibitor concentration. The four types of inhibition produce Lineweaver–Burke and Eadie–Hofstee plots[38] that vary in distinctive ways with inhibitor concentration. For brevity, two symbols are used:
- and
where Ki and K'i are the dissociation constants for binding to the enzyme and to the enzyme–substrate complex, respectively. In the presence of the reversible inhibitor, the enzyme's apparent Km and Vmax become (α/α')Km and (1/α')Vmax, respectively, as shown below for common cases.
| Type of inhibition | Km apparent | Vmax apparent | ||
| Ki only | () | competitive | ||
| Ki' only | () | uncompetitive | ||
| Ki = Ki' | () | non-competitive | ||
| Ki ≠ Ki' | () | mixed |
Non-linear regression fits of the enzyme kinetics data to the rate equations above[39] can yield accurate estimates of the dissociation constants Ki and K'i.
Irreversible inhibitors
Enzyme inhibitors can also irreversibly inactivate enzymes, usually by covalently modifying active site residues. These reactions, which may be called suicide substrates, follow exponential decay functions and are usually saturable. Below saturation, they follow first order kinetics with respect to inhibitor.
Mechanisms of catalysis

The favoured model for the enzyme–substrate interaction is the induced fit model.[40] This model proposes that the initial interaction between enzyme and substrate is relatively weak, but that these weak interactions rapidly induce conformational changes in the enzyme that strengthen binding. These conformational changes also bring catalytic residues in the active site close to the chemical bonds in the substrate that will be altered in the reaction.[41] After binding takes place, one or more mechanisms of catalysis lower the energy of the reaction's transition state by providing an alternative chemical pathway for the reaction. Mechanisms of catalysis include catalysis by bond strain; by proximity and orientation; by active-site proton donors or acceptors; covalent catalysis and quantum tunnelling.[30][42]
Enzyme kinetics cannot prove which modes of catalysis are used by an enzyme. However, some kinetic data can suggest possibilities to be examined by other techniques. For example, a ping–pong mechanism with burst-phase pre-steady-state kinetics would suggest covalent catalysis might be important in this enzyme's mechanism. Alternatively, the observation of a strong pH effect on Vmax but not Km might indicate that a residue in the active site needs to be in a particular ionisation state for catalysis to occur.
Footnotes
α. ^ Link: Interactive Michaelis–Menten kinetics tutorial (Java required)
β. ^ Link: dihydrofolate reductase mechanism (Gif)
γ. ^ Link: DNA polymerase mechanism (Gif)
δ. ^ Link: Chymotrypsin mechanism (Flash required)
References
- ^ Ebbing, D.D. General chemistry (4th edition) Houghton Mifflin Co. 1993, ISBN 0-395-63696-5
- ^ Eisenthal R. Danson M.J. (Eds), Enzyme Assays: A Practical Approach. Oxford University Press (2002) ISBN 0-19-963820-9
- ^ Xie XS, Lu HP. Single-molecule enzymology. J Biol Chem. 1999 Jun 4;274(23):15967-70. PMID 10347141
- ^ Lu H (2004). “Single-molecule spectroscopy studies of conformational change dynamics in enzymatic reactions”. Current pharmaceutical biotechnology 5 (3): 261–9. doi:10.2174/1389201043376887. PMID 15180547.
- ^ Schnell J, Dyson H, Wright P (2004). “Structure, dynamics, and catalytic function of dihydrofolate reductase”. Annual review of biophysics and biomolecular structure 33: 119–40. doi:10.1146/annurev.biophys.33.110502.133613. PMID 15139807.
- ^ Gibson Q.H. Rapid mixing: Stopped flow Methods in Enzymology, (1969) 16:187–228
- ^ Duggleby, R.G. Analysis of enzyme progress curves by non-linear regression. Methods in Enzymology, (1995) 249:61–90.
- ^ Michaelis L. and Menten M.L. Kinetik der Invertinwirkung Biochem. Z. 1913; 49:333–369 English translation Accessed 6 April 2007
- ^ Cha S (1968). “A simple method for derivation of rate equations for enzyme-catalyzed reactions under the rapid equilibrium assumption or combined assumptions of equilibrium and steady state”. J. Biol. Chem. 243 (4): 820–5. PMID 5638598.
- ^ Briggs GE, Haldane JB. A Note on the Kinetics of Enzyme Action. Biochem J. 1925;19(2):338-9. PMID 16743508
- ^ Stroppolo ME, Falconi M, Caccuri AM, Desideri A (2001). “Superefficient enzymes”. Cell. Mol. Life Sci. 58 (10): 1451–60. doi:10.1007/PL00000788. PMID 11693526.
- ^ Davis ME, Madura JD, Sines J, Luty BA, Allison SA, McCammon JA (1991). “Diffusion-controlled enzymatic reactions”. Meth. Enzymol. 202: 473–97. PMID 1784185.
- ^ Jones ME (1992). “Analysis of algebraic weighted least-squares estimators for enzyme parameters”. Biochem. J. 288 ( Pt 2): 533–8. PMID 1463456.
- ^ Tseng SJ, Hsu JP. A comparison of the parameter estimating procedures for the Michaelis–Menten model. J Theor Biol. 1990 Aug 23;145(4):457–64. PMID 2246896
- ^ Bravo, I.G., Busto, F., De Arriaga, D., Ferrero, M. A., Rodríguez-Aparicio, L. B., Martínez-Blanco H., Reglero, A. A normalised plot as a novel and time-saving tool in complex enzyme kinetic analysis Biochem. J. (2001). 358, 573–583. PMID 11577687
- ^ Almaas E, Kovacs B, Vicsek T, Oltvai ZN, Barabasi AL. Global organization of metabolic fluxes in the bacterium Escherichia coli. Nature. 2004 Feb 26;427(6977):839-43. PMID 14985762
- ^ Reed JL, Vo TD, Schilling CH, Palsson BO. An expanded genome-scale model of Escherichia coli K-12 (iJR904 GSM/GPR). Genome Biol. 2003;4(9):R54. PMID 12952533
- ^ Dirr H, Reinemer P, Huber R. X-ray crystal structures of cytosolic glutathione S-transferases. Implications for protein architecture, substrate recognition and catalytic function. Eur J Biochem. 1994 Mar 15;220(3):645-61. PMID 8143720
- ^ Stone SR, Morrison JF. Dihydrofolate reductase from Escherichia coli: the kinetic mechanism with NADPH and reduced acetylpyridine adenine dinucleotide phosphate as substrates. Biochemistry. 1988 Jul 26;27(15):5493–9. PMID 3052577
- ^ Fisher PA. Enzymologic mechanism of replicative DNA polymerases in higher eukaryotes. Prog Nucleic Acid Res Mol Biol. 1994;47:371-97. PMID 8016325
- ^ Akerman SE, Muller S. 2-Cys peroxiredoxin PfTrx-Px1 is involved in the antioxidant defence of Plasmodium falciparum. Mol Biochem Parasitol. 2003 Aug 31;130(2):75-81. PMID 12946843
- ^ Bravo, I.G., Barrallo, S., Ferrero, M. A., Rodríguez-Aparicio, L. B., Martínez-Blanco H., Reglero, A. “Kinetic properties of the Acylneuraminate Cytidylytransferase from Pasteurella haemolytica A2”. Biochem. J. (2001) 358, 585-598. [1]
- ^ Kraut J. Serine proteases: structure and mechanism of catalysis. Annu Rev Biochem. 1977;46:331-58. PMID 332063
- ^ Ricard J, Cornish-Bowden A. Co-operative and allosteric enzymes: 20 years on. Eur J Biochem. 1987 Jul 15;166(2):255-72. PMID 3301336
- ^ Ward WH, Fersht AR., Tyrosyl-tRNA synthetase acts as an asymmetric dimer in charging tRNA. A rationale for half-of-the-sites activity. Biochemistry. 1988 Jul 26;27(15):5525–30. PMID 3179266
- ^ Helmstaedt K, Krappmann S, Braus GH., Allosteric regulation of catalytic activity: Escherichia coli aspartate transcarbamoylase versus yeast chorismate mutase. Microbiol. Mol. Biol. Rev. 2001 Sep;65(3):404-21 PMID 11528003
- ^ Schirmer T, Evans PR., Structural basis of the allosteric behaviour of phosphofructokinase. Nature. 1990 Jan 11;343(6254):140-5. PMID 2136935
- ^ Hill, A. V. The possible effects of the aggregation of the molecules of haemoglobin on its dissociation curves. J. Physiol. (Lond.), 1910 40, iv-vii.
- ^ Hartley B.S. and Kilby B.A. The reaction of p-nitrophenyl esters with chymotrypsin and insulin. Biochem J. 1954 Feb;56(2):288-97. PMID 13140189
- ^ a b Alan Fersht, Structure and Mechanism in Protein Science : A Guide to Enzyme Catalysis and Protein Folding. W. H. Freeman, 1998. ISBN 0-7167-3268-8
- ^ Bender ML, Begue-Canton ML, Blakeley RL, Brubacher LJ, Feder J, Gunter CR, Kezdy FJ, Killheffer JV Jr, Marshall TH, Miller CG, Roeske RW, Stoops JK. The Determination of the Concentration of Hydrolytic Enzyme Solutions : a-Chymotrypsin, Trypsin, Papain, Elastase, Subtilisin, and Acetylcholinesterase. J Am Chem Soc. 1966 Dec 20;88(24):5890-913. PMID 5980876
- ^ Cleland WW. The use of isotope effects to determine enzyme mechanisms. Arch Biochem Biophys. 2005 Jan 1;433(1):2–12. PMID 15581561
- ^ Northrop D (1981). “The expression of isotope effects on enzyme-catalyzed reactions”. Annu. Rev. Biochem. 50: 103–31. doi:10.1146/annurev.bi.50.070181.000535. PMID 7023356.
- ^ Baillie T, Rettenmeier A (1986). “Drug biotransformation: mechanistic studies with stable isotopes”. Journal of clinical pharmacology 26 (6): 448–51. PMID 3734135.
- ^ Cleland WW. Use of isotope effects to elucidate enzyme mechanisms. CRC Crit Rev Biochem. 1982;13(4):385–428. PMID 6759038
- ^ Christianson DW, Cox JD. Catalysis by metal-activated hydroxide in zinc and manganese metalloenzymes. Annu Rev Biochem. 1999;68:33–57. PMID 10872443
- ^ Kraut D, Carroll K, Herschlag D (2003). “Challenges in enzyme mechanism and energetics”. Annu. Rev. Biochem. 72: 517–71. doi:10.1146/annurev.biochem.72.121801.161617. PMID 12704087.
- ^ Tseng SJ, Hsu JP. A comparison of the parameter estimating procedures for the Michaelis–Menten model. J Theor Biol. 1990 Aug 23;145(4):457–64. PMID 2246896
- ^ Leatherbarrow RJ. Using linear and non-linear regression to fit biochemical data. Trends Biochem Sci. 1990 Dec;15(12):455–8. PMID 2077683
- ^ Koshland DE, Application of a Theory of Enzyme Specificity to Protein Synthesis. Proc. Natl. Acad. Sci. U.S.A. 1958 Feb;44(2):98–104. PMID 16590179
- ^ Hammes G (2002). “Multiple conformational changes in enzyme catalysis”. Biochemistry 41 (26): 8221–8. doi:10.1021/bi0260839. PMID 12081470.
- ^ Sutcliffe M, Scrutton N (2002). “A new conceptual framework for enzyme catalysis. Hydrogen tunnelling coupled to enzyme dynamics in flavoprotein and quinoprotein enzymes”. Eur. J. Biochem. 269 (13): 3096–102. doi:10.1046/j.1432-1033.2002.03020.x. PMID 12084049.
Further reading
Introductory
- Athel Cornish-Bowden, Fundamentals of Enzyme Kinetics. (3rd edition), Portland Press 2004, ISBN 1-85578-158-1.
- Nicholas Price, Lewis Stevens, Fundamentals of Enzymology, Oxford University Press, 1999. ISBN 0-19-850229-X
- Tim Bugg, An Introduction to Enzyme and Coenzyme Chemistry Blackwell Publishing, 2004 ISBN 1-4051-1452-5
Advanced
- Irwin H. Segel, Enzyme Kinetics : Behavior and Analysis of Rapid Equilibrium and Steady-State Enzyme Systems. Wiley-Interscience; New Ed edition 1993, ISBN 0-471-30309-7.
- Alan Fersht, Structure and Mechanism in Protein Science : A Guide to Enzyme Catalysis and Protein Folding. W. H. Freeman, 1998. ISBN 0-7167-3268-8
- Santiago Schnell, Philip K. Maini, A century of enzyme kinetics: Reliability of the KM and vmax estimates, Comments on Theoretical Biology 8, 169–187, 2004 DOI: 10.1080/08948550390206768
- Chris Walsh, Enzymatic Reaction Mechanisms. W. H. Freeman and Company. 1979. ISBN 0-7167-0070-0
- Paul F. Cook and W.W. Cleland Enzyme kinetics and mechanism Garland Science, 2007 ISBN 0-8153-4140-7
External links
- Animation of an enzyme assay — Shows effects of manipulating assay conditions
- MACiE — A database of enzyme reaction mechanisms
- ENZYME — Expasy enzyme nomenclature database
- ExCatDB — A database of enzyme catalytic mechanisms
- BRENDA — Comprehensive enzyme database, giving substrates, inhibitors and reaction diagrams
- SABIO-RK — A database of reaction kinetics
- Joseph Kraut's Research Group, University of California San Diego — Animations of several enzyme reaction mechanisms
- Symbolism and Terminology in Enzyme Kinetics — A comprehensive explanation of concepts and terminology in enzyme kinetics
- An introduction to enzyme kinetics — An accessible set of on-line tutorials on enzyme kinetics
- Enzyme kinetics animated tutorial — An animated tutorial with audio


![{\displaystyle {\begin{matrix}v=k_{2}[{\mbox{ES}}]\end{matrix}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/befe56463761168b47df5be5bfa796c6d3f6a7dd)
![{\displaystyle [{\mbox{E}}]_{\mathrm {tot} }\ {\stackrel {\mathrm {def} }{=}}\ [{\mbox{E}}]+[{\mbox{ES}}]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/d6be4bf3259d0b0e0cb5c7ca927bc7d35f6831ad)
![{\displaystyle v={\frac {V_{\max[}{\mbox{S}}]}{K_{m}+[{\mbox{S}}]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/b9fc1a556482d5787a07f8418562cf95ac8ffb37)
![{\displaystyle {\frac {d}{dt}}[{\mbox{ES}}]=k_{1}[{\mbox{E}}][{\mbox{S}}]-k_{2}[{\mbox{ES}}]-k_{-1}[{\mbox{ES}}]\approx 0.}](https://wikimedia.org/api/rest_v1/media/math/render/svg/3daed02cecf2893959a37ddb0ef331e2abd2deca)
![{\displaystyle [{\mbox{ES}}]\approx {\frac {[{\mbox{E}}]_{\mathrm {tot} }[{\mbox{S}}]}{[{\mbox{S}}]+K_{m}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/6af63680a6b832123f8fb44e0d4bb3b0e10adb9d)
![{\displaystyle K_{m}\ {\stackrel {\mathrm {def} }{=}}\ {\frac {k_{2}+k_{-1}}{k_{1}}}\approx {\frac {[{\mbox{E}}][{\mbox{S}}]}{[{\mbox{ES}}]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/428e7c1efa5baf314c725f64da8b0ef44c0034c3)
![{\displaystyle v=k_{2}[\mathrm {ES} ]={\frac {k_{2}[{\mbox{E}}]_{\mathrm {tot} }[{\mbox{S}}]}{[{\mbox{S}}]+K_{m}}}={\frac {V_{\max[}{\mbox{S}}]}{[{\mbox{S}}]+K_{m}}}.}](https://wikimedia.org/api/rest_v1/media/math/render/svg/daeeef493437fcf0ceedb52bf21f19064d63cecf)

![{\displaystyle v=k_{2}[\mathrm {ES} ]={\frac {k_{2}}{K_{m}}}[{\mbox{E}}][{\mbox{S}}]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/ce445b17dc8f27b81e07db99d3532af0be5c53ee)
![{\displaystyle {\frac {1}{v}}={\frac {K_{m}}{V_{\max[}{\mbox{S}}]}}+{\frac {1}{V_{\max }}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/3ace7013310ad596d2ba08978b8011292459dccc)
![{\displaystyle \alpha =1+{\frac {[{\mbox{I}}]}{K_{i}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/7d05bffbc19ef9047581c87887ae0eea5c11e7fe)
![{\displaystyle \alpha ^{\prime }=1+{\frac {[{\mbox{I}}]}{K_{i}^{\prime }}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/e8b2033b5725a8b85d7425d409192cf11885c199)









