相補性 (分子生物学)
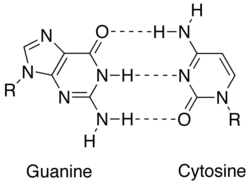

分子生物学において相補性(そうほせい、英: complementarity)とは、2つの構造が互いに鍵と鍵穴の関係にあるような関係性を意味している。自然界において、相補性はDNAの複製や転写の際の基本原則であり、相補的(complementary)な塩基対の形成によって、細胞は情報を次の世代へコピーすることが可能となり、配列中に保存されている情報の損傷を見つけて修復することも可能となる。
2つの核酸鎖の間でみられる相補性は完全な相補性から全く相補性がないものまでさまざまであり、相補性の程度によってこれらの配列が結合した際の安定性が決定される。さらに、DNA修復など、さまざまな調節機能が塩基対の相補性に基づいて行われている。塩基対の相補性の原則に基づいてDNA鎖とRNA鎖のハイブリッド二本鎖を形成することも可能であり、cDNAライブラリなどの現代的な生物工学のツールでこうした性質が利用されている。相補性の多くは2つの異なるDNA/RNA鎖の間で見られるものであるが、配列内部に相補性を有する配列では自分自身と相補的な結合を行って折りたたまれた構造をとることも可能である。
DNAとRNAの塩基対の相補性[編集]


相補性は、核酸塩基(アデニン、チミン(RNAではウラシル)、グアニン、シトシン)の間の相互作用によって達成されている。アデニンとグアニンはプリンであり、チミン、シトシンとウラシルはピリミジンである。プリンはピリミジンよりも大きい。どちらも、異なるタイプの核酸塩基とだけ塩基対を形成する。核酸では塩基は水素結合によって結びつくが、水素結合はアデニンとチミン(ウラシル)の間、グアニンとシトシンの間でのみ効果的に機能する。相補的な塩基対A=T(A=U)は2つの水素結合を持ち、G≡C塩基対は3つの水素結合を持つ。他の核酸塩基の配置は、すべて二重らせんの形成を阻害する。DNAの二本鎖は逆の方向を向いており、逆平行と呼ばれる[1]。
| 核酸 | 核酸塩基 | 相補的塩基対 |
| DNA | アデニン(A)、チミン(T)、グアニン(G)、シトシン(C) | A=T、G≡C |
| RNA | アデニン(A)、ウラシル(U)、グアニン(G)、シトシン(C) | A=U、G≡C |
DNAまたはRNAの相補鎖は、核酸塩基の相補性に基づいて構築される[2]。各塩基対、A=TとG≡Cはほぼ同じ位置を占めるため、立体的な歪みが生じることなくDNAの二重らせんが形成される。核酸塩基間の水素結合もDNA二重らせんを安定化する[3]。
二重らせんを形成するDNA鎖の相補性によって、一方の鎖を鋳型として利用して他方を構築することが可能となる。この原理はDNA複製に重要な役割を果たし、遺伝情報がどのように次の世代へ受け渡されるかについての基礎をなしている。相補性はDNAの転写でも利用され、DNAの鋳型からRNA鎖が生成される[4]。
校正などのDNA修復過程も相補性に基づいており、相補的でない核酸塩基を除去することでDNA複製の差異のエラーを修正することができる[1]。
一本鎖DNAは相補的なDNAまたはRNAと容易に結合し、ハイブリダイゼーションを行う。この原理は、ポリメラーゼ連鎖反応(PCR)などの一般的な実験技術の基礎をなしている[1]。
相補的な配列を持つ2本の鎖は、センス、アンチセンスと呼ばれる。一般的に、センス鎖は転写されるDNA配列または転写によって生成されたRNA鎖である。一方、アンチセンス鎖はセンス鎖に相補的な鎖である。
ヘアピンループの自己相補性[編集]

自己相補性(self-complementarity)とは、DNAまたはRNAの配列が折り返されて二本鎖に似た構造が形成されることを指している。配列のどの部分が自己相補性を示すかによって、鎖はヘアピンループ、ジャンクション、バルジ、インターナルループを形成する[1]。RNAは、グアニンとウラシルの間の結合などDNAではみられない塩基対が形成されるため、こうした構造を形成しやすい[1]。
調節機能[編集]
転写された遺伝子配列中に存在する相補的な核酸配列は塩基対を形成する。こうした構造は自然界でも一般的にみられ、遺伝子サイレンシングなどの調節機能を持っている[1]。
アンチセンス転写産物[編集]
アンチセンス転写産物は、コーディング配列に相補的なノンコーディングRNAである[5]。ゲノムワイド解析によって、アンチセンスRNAは自然界で一般的に生じていることが示されている。これらは、遺伝暗号のコーディング能力を増大させ、遺伝子調節に新たな複雑性を加えるものであると考えられている。これまで、ヒトゲノムの40%が双方向に転写されることが知られており、逆方向の転写の重要性が示唆されている[6]。センス転写産物とアンチセンス転写産物間の相補性領域は二本鎖RNAハイブリッドを形成し、遺伝子調節に重要な役割を果たす可能性がある。例えば、HIF1αとβ-セクレターゼのmRNAは双方向に転写されており、アンチセンス転写産物がセンス転写産物の安定化因子として機能していることが示されている[7]。
miRNAとsiRNA[編集]

miRNAは転写される遺伝子領域に相補的なRNA配列で、調節機能を持つ。現在の研究からは、体内を循環するmiRNAは新たなバイオマーカーとして利用できる可能性が示唆されており、疾患診断での利用の有望性が示されている[8]。miRNAは調節遺伝子に由来するより長いRNA配列から形成され、Dicerなどによって切断される。こうして形成された短い鎖はRNA誘導サイレンシング複合体(RISC)に結合する。そして、転写された遺伝子のmRNAへ相補性を利用して結合し、3つの方法で遺伝子のサイレンサーとして機能する。1つ目は、リボソームの結合を阻害し、翻訳の開始を防ぐことである。2つ目は、結合したmRNAを分解することである。3つ目は、Dicerが作用する2本鎖RNAを形成し、より多くのmiRNAを作り出すことでより多くのコピーを分解することである。siRNAはmiRNAと似た機能を持つ。siRNAは異なるRNAに由来するが、miRNAと同様な目的を果たす[1]。これらの配列の短さを考えると、相補性の規則は選択された標的を非常に特異的に認識できることを意味している。各塩基の位置ごとに4つの選択肢があるとすると、mi/siRNAの20–22塩基で1×1012通り以上の組み合わせを作り出すことが可能である。ヒトゲノムの長さが31億塩基対であるすると[9]、各miRNAがゲノム全体で偶然に一致するのは1ヶ所だけであることを意味している。
バイオインフォマティクス[編集]
相補性はDNAやRNAに存在する情報を一本の鎖に保存することを可能にする。鋳型鎖から相補鎖を決定することができ、逆にcDNAライブラリからは鋳型鎖を決定することができる。ミスマッチが存在する配列の簡略表記法(曖昧コード)、相補鎖を速く読み取るための表記法(アンビグラム)が開発されている。
cDNAライブラリ[編集]
cDNAライブラリは発現している遺伝子のコレクションであり、遺伝子の同定やクローニングの際に有用な参照ツールである。cDNAライブラリはRNA依存性DNAポリメラーゼ(逆転写酵素)を用いて、mRNAからDNAへの逆転写によって構築される。この過程はDNA/RNA間の相補性の原則に基づいている。ライブラリの最終産物は二本鎖DNAであり、プラスミドへ挿入される場合もある。cDNAライブラリは現代の研究において強力なツールである[1][10]。
曖昧コード[編集]
| シンボル[11] | 意味 | 塩基 | ||||
|---|---|---|---|---|---|---|
| A | アデニン(adenine) | A | 1 | |||
| C | シトシン(cytosine) | C | ||||
| G | グアニン(guanine) | G | ||||
| T | チミン(thymine) | T | ||||
| U | ウラシル(uracil) | U | ||||
| W | 弱い相互作用(weak) | A | T | 2 | ||
| S | 強い相互作用(strong) | C | G | |||
| M | アミノ(amino) | A | C | |||
| K | ケト(keto) | G | T | |||
| R | プリン(purine) | A | G | |||
| Y | ピリミジン(pyrimidine) | C | T | |||
| B | A以外(Aの後のB) | C | G | T | 3 | |
| D | C以外(Cの後のD) | A | G | T | ||
| H | G以外(Gの後のH) | A | C | T | ||
| V | T(U)以外(T、Uの後のV) | A | C | G | ||
| N | 任意(any) | A | C | G | T | 4 |
体系学において配列を表記する際、「2つのうちのいずれか」や「3つのうちのいずれか」を意味するIUPAC表記法を用いる必要がある場合がある。IUPAC表記法のR(AまたはG)はY(CまたはT)と相補的であり、M(AまたはC)はK(GまたはT)と相補的である。W(AまたはT)とS(CまたはG)は相補的ではない。WとSはヌクレオチドが相補的なパートナーと対合する際に用いる水素結合の数を示しており、パートナーは同数の結合を持っていなければならない[12]。
特定の1つのヌクレオチドを除去したコードは、そこで除去されたヌクレオチドと相補的なヌクレオチドを除去したコードと相補的となる。例えば、V(T以外)はB(A以外)と相補的となる。
アンビグラム[編集]
特定の文字(アンビグラム)を核酸の表記に用いることで、例えば、グアニンをb、シトシンをq、アデニンをn、チミンをuで表記すると、テキストをひっくり返すだけで相補的なDNA配列を作ることができる[13]。buqn(GTCA)はひっくり返すと、ubnq(TGAC)となる。
- qqubqnnquunbbqnbb
- bbnqbuubnnuqqbuqq
アンビグラムを用いた表記は、回文配列などの相補的な核酸配列の可視化を容易にする[14]。通常のASCIIやUnicode文字ではなくカスタムフォントを用いることで、この特徴はより明確になる[14]。
出典[編集]
- ^ a b c d e f g h Watson, James, Cold Spring Harbor Laboratory, Tania A. Baker, Massachusetts Institute of Technology, Stephen P. Bell, Massachusetts Institute of Technology, Alexander Gann, Cold Spring Harbor Laboratory, Michael Levine, University of California, Berkeley, Richard Losik, Harvard University ; with Stephen C. Harrison, Harvard Medical (2014). Molecular biology of the gene (Seventh ed.). Boston: Benjamin-Cummings Publishing Company. ISBN 978-0-32176243-6
- ^ Pray, Leslie (2008). “Discovery of DNA structure and function: Watson and Crick”. Nature Education 1 (1): 100 2013年11月27日閲覧。.
- ^ Shankar, A; Jagota, A; Mittal, J (Oct 11, 2012). “DNA base dimers are stabilized by hydrogen-bonding interactions including non-Watson-Crick pairing near graphite surfaces.”. The Journal of Physical Chemistry B 116 (40): 12088–94. doi:10.1021/jp304260t. PMID 22967176.
- ^ Hood, L; Galas, D (Jan 23, 2003). “The digital code of DNA.”. Nature 421 (6921): 444–8. Bibcode: 2003Natur.421..444H. doi:10.1038/nature01410. PMID 12540920.
- ^ He, Y; Vogelstein, B; Velculescu, VE; Papadopoulos, N; Kinzler, KW (Dec 19, 2008). “The antisense transcriptomes of human cells.”. Science 322 (5909): 1855–7. Bibcode: 2008Sci...322.1855H. doi:10.1126/science.1163853. PMC 2824178. PMID 19056939.
- ^ Katayama, S; Tomaru, Y; Kasukawa, T; Waki, K; Nakanishi, M; Nakamura, M; Nishida, H; Yap, CC et al. (Sep 2, 2005). “Antisense transcription in the mammalian transcriptome.”. Science 309 (5740): 1564–6. Bibcode: 2005Sci...309.1564R. doi:10.1126/science.1112009. PMID 16141073.
- ^ Faghihi, MA; Zhang, M; Huang, J; Modarresi, F; Van der Brug, MP; Nalls, MA; Cookson, MR; St-Laurent G, 3rd et al. (2010). “Evidence for natural antisense transcript-mediated inhibition of microRNA function.”. Genome Biology 11 (5): R56. doi:10.1186/gb-2010-11-5-r56. PMC 2898074. PMID 20507594.
- ^ Kosaka, N; Yoshioka, Y; Hagiwara, K; Tominaga, N; Katsuda, T; Ochiya, T (Sep 5, 2013). “Trash or Treasure: extracellular microRNAs and cell-to-cell communication.”. Frontiers in Genetics 4: 173. doi:10.3389/fgene.2013.00173. PMC 3763217. PMID 24046777.
- ^ “Ensembl genome browser 73: Homo sapiens - Assembly and Genebuild”. Ensembl.org. 2013年11月27日閲覧。
- ^ Wan, KH; Yu, C; George, RA; Carlson, JW; Hoskins, RA; Svirskas, R; Stapleton, M; Celniker, SE (2006). “High-throughput plasmid cDNA library screening.”. Nature Protocols 1 (2): 624–32. doi:10.1038/nprot.2006.90. PMID 17406289.
- ^ Cornish-Bowden, A. (1985-05-10). “Nomenclature for incompletely specified bases in nucleic acid sequences: recommendations 1984”. Nucleic Acids Research 13 (9): 3021–3030. doi:10.1093/nar/13.9.3021. ISSN 0305-1048. PMC 341218. PMID 2582368.
- ^ Reverse-complement tool page with documented IUPAC code conversion, source code available.
- ^ Rozak DA (2006). “The practical and pedagogical advantages of an ambigraphic nucleic acid notation”. Nucleosides Nucleotides Nucleic Acids 25 (7): 807–13. doi:10.1080/15257770600726109. PMID 16898419.
- ^ a b Rozak, DA; Rozak, AJ (May 2008). “Simplicity, function, and legibility in an enhanced ambigraphic nucleic acid notation.”. BioTechniques 44 (6): 811–3. doi:10.2144/000112727. PMID 18476835.
