「リプレッサー」の版間の差分
リンク違いを修正 |
m bot: 解消済み仮リンクコリプレッサーを内部リンクに置き換えます |
||
| (4人の利用者による、間の4版が非表示) | |||
| 1行目: | 1行目: | ||
[[File:Lac_Operon.svg|thumb|300x300px|ラクトースオペロンの模式図。'''1: RNAポリメラーゼ、2: lacリプレッサー、3: プロモーター、4: オペレーター、5: ラクトース、6: lacZ、7: lacY、8: lacA。上:''' 遺伝子がオフの状態。リプレッサーを阻害するラクトースが存在しないためリプレッサーはオペレーターに結合し、RNAポリメラーゼのプロモーターへの結合とラクターゼの合成を防いでいる。'''下:''' 遺伝子がオンの状態。ラクトースがリプレッサーを阻害し、RNAポリメラーゼはプロモーターに結合して遺伝子発現によってラクターゼが合成される。ラクターゼはリプレッサーに結合するラクトースが存在しなくなるまでラクトースを分解する。その後、ラクトースが解離したリプレッサーは再びオペレーターに結合し、ラクターゼの合成を止める。]] |
|||
'''リプレッサー''' ({{lang-en-short|Repressor}})と は、[[デオキシリボ核酸]] (DNA) の[[プロモーター]]近傍に位置する[[オペレーター]]に結合して[[遺伝子]]の発現を抑える[[タンパク質]]性の[[転写調節因子]]のことである。'''レプレッサー'''とも言われる。プロモーターやオペレーターが[[転写]]に関わる[[シス因子]]であるのに対し、リプレッサーは[[トランス因子]]である。 |
|||
[[分子遺伝学]]において'''リプレッサー'''({{Lang-en-short|repressor}})は、[[オペレーター]]または関連する{{仮リンク|サイレンサー (遺伝学)|en|Silencer (genetics)|label=サイレンサー}}に結合することで[[遺伝子発現|遺伝子の発現]]を阻害する、[[DNA結合タンパク質]]または[[RNA結合タンパク質]]である。DNAに結合するリプレッサーは[[プロモーター]]への[[RNAポリメラーゼ]]の結合をブロックし、[[遺伝子]]の[[伝令RNA|mRNA]]への[[転写 (生物学)|転写]]を防ぐ。RNAに結合するリプレッサーはmRNAに結合し、mRNAから[[タンパク質]]への[[翻訳 (生物学)|翻訳]]を防ぐ。こうしてもたらされる遺伝子発現の低下は、抑制(リプレッション、repression)と呼ばれる。 |
|||
普通、リプレッサーは結合を解除する低分子化合物と組になっており、基質と結合することでオペレーターとの結合が解除される。リプレッサーと結合してリプレッサーを不活化してオペレーターから遊離させ、転写できるようにする低分子化合物を[[インデューサー]](inducer)という。一方、リプレッサーと結合してリプレッサーを活性化してオペレーターに結合できるようにし、転写できなくなるようにする低分子化合物を[[コリプレッサー]](corepressor)という。[[原核生物]]においては、一つのリプレッサーとオペレーターの組み合わせが発現を抑える遺伝子は1つとは限らない(→[[オペロン]])。これは原核生物型の遺伝子群が、単一の[[メッセンジャーRNA]]上に複数の[[コーディング領域]]であるシストロンを持つポリシストロニック(polycistronic)・オペロンとして転写される場合が多いことと関連がある。 |
|||
== 機能 == |
|||
また、リプレッサーとは逆の役割を持つタンパク質もあり、こちらは[[アクチベーター]]と呼ばれる。 |
|||
遺伝子発現を開始する分子である[[インデューサー]]が存在する場合、インデューサーはリプレッサータンパク質と相互作用し、リプレッサーをオペレーターから除去する。その後、RNAポリメラーゼは遺伝子を転写する(遺伝子を発現する)ことができるようになる。この抑制機構はインデューサーの存在など特定の条件下でのみ転写を可能にし、一種のフィードバック機構を形成していることが多い。 |
|||
[[コリプレッサー]]はリプレッサーに結合する分子で、リプレッサーがオペレーターへより強固に結合できるようにすることで転写を低下させる。コリプレッサーと結合することでオペレーターに結合するタイプのリプレッサーは、アポリプレッサー(aporepressor)または不活性リプレッサー(inactive repressor)と呼ばれている。アポリプレッサーの例としては、細菌の重要な代謝タンパク質である{{仮リンク|トリプトファンリプレッサー|en|Tryptophan repressor}}が挙げられる。対照的に活性リプレッサー(active repressor)はオペレーターに直接結合し、転写を低下させる。 |
|||
== リプレッサーの例 == |
|||
=== ''lac''オペロンリプレッサー === |
|||
{{main|ラクトースオペロン}} |
|||
''lacZYA''オペロンはラクトースの分解に必要なタンパク質をコードする遺伝子を含んでいる。''lacI''遺伝子は一般に「''lac''リプレッサー」と呼ばれるタンパク質をコードし、''lac''オペロンのリプレッサーとして機能する。''lacI''遺伝子は''lacZYA''のすぐ上流に位置するが、転写は''lac''オペロンのプロモーターとは異なる''lacI''プロモーターから行われる。''lacI''遺伝子はLacIリプレッサータンパク質を合成し、LacIリプレッサータンパク質はオペレーター配列''lacO''に結合してlacZYAを抑制する<ref name=":1">{{Cite book|edition=1st ed|title=Microbiology : an evolving science|url=https://www.worldcat.org/oclc/185042615|publisher=W.W. Norton & Co|date=2009|location=New York|isbn=978-0-393-97857-5|oclc=185042615|others=John Watkins Foster|first=Joan|last=Slonczewski}}</ref>。 |
|||
''lac''リプレッサーは恒常的に発現しており、通常は''lacO''に結合してRNAポリメラーゼによる''lac''オペロンの転写の開始を阻害している。リプレッサーはインデューサーであるアロラクトースの存在下でコンフォメーション変化を起こし、DNA結合強度が低下して''lacO''配列から解離する。その後、RNAポリメラーゼは''lac''オペロンのプロモーターに結合して''lacZYA''遺伝子の転写を開始できるようになる<ref name=":1" />。 |
|||
=== ''met''オペロンリプレッサー === |
|||
リプレッサータンパク質の他の例としては、[[メチオニン]]リプレッサーMetJがある。MetJはリボン-ヘリックス-ヘリックス(RHH)モチーフを介してDNAと相互作用する<ref name=":2">{{cite journal|last1=Somers & Phillips|date=1992|title=Crystal structure of the met repressor-operator complex at 2.8 A resolution reveals DNA recognition by beta-strands.|journal=Nature|volume=359|issue=6394|pages=387–393|doi=10.1038/359387a0|pmid=1406951|s2cid=29799322}}</ref>。MetJは2つの単量体から構成されるホモ二量体である。各単量体の[[Βシート|βリボン]]は共に逆平行βシートを形成し、Met boxと呼ばれるオペレーター配列にDNAの主溝側から結合する。結合したMetJ二量体は、オペレーター配列の相補鎖に結合した他のMetJ二量体と[[αヘリックス]]を介して相互作用する。 |
|||
Met boxはAGACGTCTからなる[[回文配列]]で、DNAのどちらの鎖でも同じ配列が認識される。Met boxの中央に位置するCとGの間の接合部にはピリミジン-プリンステップが含まれており、[[ホスホジエステル結合|ホスホジエステル]]骨格が屈曲して正の[[DNA超らせん|超らせん]]が形成される。タンパク質は、DNA二重らせんの形状がタンパク質の形状に沿うことで、認識部位を確認する。言い換えると、タンパク質は特定の塩基配列を認識するのではなく、DNAの構造パラメータを間接的に読み取ることで認識が行われる。 |
|||
MetJ二量体には2つの[[補因子]][[S-アデノシルメチオニン|''S''-アデノシルメチオニン]](SAM)結合部位が存在する。SAMはメチオニン生合成の産物である。MetJのSAM結合ポケットはDNA結合部位とは重複していない。SAMはMetJに結合してMetJのオペレーター部位への親和性を高め、メチオニン合成に関係する遺伝子の転写を停止させる。SAM濃度が低くなると、リプレッサーはオペレーター部位から解離し、より多くのメチオニンが合成されるようになる。 |
|||
=== L-アラビノースオペロンリプレッサー === |
|||
{{main|L-アラビノースオペロン}} |
|||
L-アラビノースオペロンには[[アラビノース]]を分解する酵素が含まれている。これらの酵素は、グルコース濃度が低いか存在しないときに代替的なエネルギー源としてアラビノースを分解する役割を果たす<ref name="Voet, Donald, 2011">{{Cite book|last=Voet, Donald|url=https://www.worldcat.org/oclc/690489261|title=Biochemistry|date=2011|publisher=John Wiley & Sons|others=Voet, Judith G.|isbn=978-0-470-57095-1|edition=4th|location=Hoboken, NJ|oclc=690489261}}</ref>。オペロンはリプレッサー遺伝子(''araC'')、4つの制御部位(''araO<sub>2</sub>''、''araO<sub>1</sub>''、''araI<sub>1</sub>''、''araI<sub>2</sub>'')、2つのプロモーター(''Para<sub>c</sub>''/''Para<sub>BAD</sub>'')、3つの構造遺伝子(''araBAD'')から構成される。AraCは合成されると制御部位に結合してループを形成し、RNAポリメラーゼのプロモーター領域への結合と構造遺伝子の転写を防ぐリプレッサーとして機能する。 |
|||
アラビノースもAraCも存在しない場合、ループ形成は開始されないが構造遺伝子の発現は低い状態である。アラビノースは存在しないがAraCが存在する場合、AraCは二量体を形成して''araO<sub>2</sub>''と''araI<sub>1</sub>''に結合し、ループ形成によって''araO<sub>2</sub>''と''araI<sub>1</sub>''を近接させる<ref name=":3">{{Cite journal|last1=Harmer|first1=Tara|last2=Wu|first2=Martin|last3=Schleif|first3=Robert|date=2001-01-16|title=The role of rigidity in DNA looping-unlooping by AraC|url=https://www.pnas.org/content/98/2/427|journal=Proceedings of the National Academy of Sciences|volume=98|issue=2|pages=427–431|language=en|bibcode=2001PNAS...98..427H|doi=10.1073/pnas.98.2.427|issn=0027-8424|pmid=11209047|pmc=14602}}</ref>。アラビノースとAraCの双方が存在する場合、AraCはアラビノースと結合し、[[アクチベーター]]として機能する。AraCはアラビノースの結合に伴うコンフォメーション変化によってループを形成することができなくなり、線形となった遺伝子領域は''araBAD''領域へのRNAポリメラーゼのリクルートを促進する<ref name="Voet, Donald, 2011" />。 |
|||
[[File:Structure_of_L-arabinose_operon_of_E._coli.tif|center|thumb|600x600px|大腸菌のL-アラビノースオペロンの構造 ]] |
|||
=== ''Flowing Locus C''(エピジェネティックリプレッサー) === |
|||
{{main|{{仮リンク|Flowering Locus C|en|Flowering Locus C}}}} |
|||
[[シロイヌナズナ]]''Arabidopsis thaliana''の''Flowering Locus C''(''FLC'')オペロンは、[[開花]]のための[[メリステム]]の発達に必要な遺伝子を抑制することで、開花を負に制御している。''FLC''の発現は[https://www.uniprot.org/uniprot/P0DH90 FRIGIDA]の存在によって調節されていることが示されており、温度の低下と負に相関して[[春化]]を防いでいる<ref name=":4">{{Cite journal|last1=Shindo|first1=Chikako|last2=Aranzana|first2=Maria Jose|last3=Lister|first3=Clare|last4=Baxter|first4=Catherine|last5=Nicholls|first5=Colin|last6=Nordborg|first6=Magnus|last7=Dean|first7=Caroline|date=June 2005|title=Role of FRIGIDA and FLOWERING LOCUS C in Determining Variation in Flowering Time of Arabidopsis|journal=Plant Physiology|volume=138|issue=2|pages=1163–1173|doi=10.1104/pp.105.061309|issn=0032-0889|pmid=15908596|pmc=1150429}}</ref>。発現低下の程度は、季節の進行に伴う温度や露光時間の変化に依存している。''FLC''の発現がダウンレギュレーションされると、開花の可能性が出てくる。''FLC''の発現調節には、遺伝的な要因と{{仮リンク|ヒストンのメチル化|en|Histone methylation}}や[[DNAメチル化]]などの[[エピジェネティック]]な要因が関係している<ref name=":5">{{Cite journal|last1=Johanson|first1=U.|last2=West|first2=J.|last3=Lister|first3=C.|last4=Michaels|first4=S.|last5=Amasino|first5=R.|last6=Dean|first6=C.|date=2000-10-13|title=Molecular analysis of FRIGIDA, a major determinant of natural variation in Arabidopsis flowering time|url=https://pubmed.ncbi.nlm.nih.gov/11030654/|journal=Science|volume=290|issue=5490|pages=344–347|bibcode=2000Sci...290..344J|doi=10.1126/science.290.5490.344|issn=0036-8075|pmid=11030654}}</ref>。さらに、''FLC''遺伝子の負の転写因子として働くコファクターとなる遺伝子も多数存在する<ref name=":6">{{Cite journal|last1=Finnegan|first1=E. Jean|last2=Kovac|first2=Kathryn A.|last3=Jaligot|first3=Estelle|last4=Sheldon|first4=Candice C.|last5=Peacock|first5=W. James|last6=Dennis|first6=Elizabeth S.|date=2005|title=The downregulation of FLOWERING LOCUS C (FLC) expression in plants with low levels of DNA methylation and by vernalization occurs by distinct mechanisms|url=https://onlinelibrary.wiley.com/doi/abs/10.1111/j.1365-313X.2005.02541.x|journal=The Plant Journal|volume=44|issue=3|pages=420–432|language=en|doi=10.1111/j.1365-313X.2005.02541.x|issn=1365-313X|pmid=16236152|doi-access=free}}</ref>。また、''FLC''遺伝子には種を超えて多数の相同な遺伝子が存在し、さまざまな気候での適応を可能にしている<ref name=":7">{{Cite journal|last1=Sharma|first1=Neha|last2=Ruelens|first2=Philip|last3=D'hauw|first3=Mariëlla|last4=Maggen|first4=Thomas|last5=Dochy|first5=Niklas|last6=Torfs|first6=Sanne|last7=Kaufmann|first7=Kerstin|last8=Rohde|first8=Antje|last9=Geuten|first9=Koen|date=February 2017|title=A Flowering Locus C Homolog Is a Vernalization-Regulated Repressor in Brachypodium and Is Cold Regulated in Wheat1[OPEN]|journal=Plant Physiology|volume=173|issue=2|pages=1301–1315|doi=10.1104/pp.16.01161|issn=0032-0889|pmid=28034954|pmc=5291021}}</ref>。 |
|||
== 出典 == |
|||
{{Reflist}} |
|||
== 関連項目 == |
== 関連項目 == |
||
* [[遺伝子発現]] |
* [[遺伝子発現]] |
||
* [[プロモーター]] |
|||
* [[アクチベーター]] |
|||
* [[オペロン]] |
|||
* [[遺伝子発現の調節]] |
|||
* [[転写因子]] |
|||
* {{仮リンク|lacリプレッサー|en|Lac repressor|label=''lac''リプレッサー}} |
|||
* {{仮リンク|p300-CBPコアクチベーターファミリー|en|p300-CBP coactivator family}} |
|||
| ⚫ | |||
<!-- == 参考文献 == --> |
|||
* {{MeshName|Repressor+Proteins}} |
|||
| ⚫ | |||
{{Biosci-stub}} |
|||
{{DEFAULTSORT:りふれつさあ}} |
{{DEFAULTSORT:りふれつさあ}} |
||
2021年6月11日 (金) 20:33時点における版
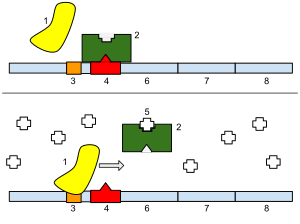
分子遺伝学においてリプレッサー(英: repressor)は、オペレーターまたは関連するサイレンサーに結合することで遺伝子の発現を阻害する、DNA結合タンパク質またはRNA結合タンパク質である。DNAに結合するリプレッサーはプロモーターへのRNAポリメラーゼの結合をブロックし、遺伝子のmRNAへの転写を防ぐ。RNAに結合するリプレッサーはmRNAに結合し、mRNAからタンパク質への翻訳を防ぐ。こうしてもたらされる遺伝子発現の低下は、抑制(リプレッション、repression)と呼ばれる。
機能
遺伝子発現を開始する分子であるインデューサーが存在する場合、インデューサーはリプレッサータンパク質と相互作用し、リプレッサーをオペレーターから除去する。その後、RNAポリメラーゼは遺伝子を転写する(遺伝子を発現する)ことができるようになる。この抑制機構はインデューサーの存在など特定の条件下でのみ転写を可能にし、一種のフィードバック機構を形成していることが多い。
コリプレッサーはリプレッサーに結合する分子で、リプレッサーがオペレーターへより強固に結合できるようにすることで転写を低下させる。コリプレッサーと結合することでオペレーターに結合するタイプのリプレッサーは、アポリプレッサー(aporepressor)または不活性リプレッサー(inactive repressor)と呼ばれている。アポリプレッサーの例としては、細菌の重要な代謝タンパク質であるトリプトファンリプレッサーが挙げられる。対照的に活性リプレッサー(active repressor)はオペレーターに直接結合し、転写を低下させる。
リプレッサーの例
lacオペロンリプレッサー
lacZYAオペロンはラクトースの分解に必要なタンパク質をコードする遺伝子を含んでいる。lacI遺伝子は一般に「lacリプレッサー」と呼ばれるタンパク質をコードし、lacオペロンのリプレッサーとして機能する。lacI遺伝子はlacZYAのすぐ上流に位置するが、転写はlacオペロンのプロモーターとは異なるlacIプロモーターから行われる。lacI遺伝子はLacIリプレッサータンパク質を合成し、LacIリプレッサータンパク質はオペレーター配列lacOに結合してlacZYAを抑制する[1]。
lacリプレッサーは恒常的に発現しており、通常はlacOに結合してRNAポリメラーゼによるlacオペロンの転写の開始を阻害している。リプレッサーはインデューサーであるアロラクトースの存在下でコンフォメーション変化を起こし、DNA結合強度が低下してlacO配列から解離する。その後、RNAポリメラーゼはlacオペロンのプロモーターに結合してlacZYA遺伝子の転写を開始できるようになる[1]。
metオペロンリプレッサー
リプレッサータンパク質の他の例としては、メチオニンリプレッサーMetJがある。MetJはリボン-ヘリックス-ヘリックス(RHH)モチーフを介してDNAと相互作用する[2]。MetJは2つの単量体から構成されるホモ二量体である。各単量体のβリボンは共に逆平行βシートを形成し、Met boxと呼ばれるオペレーター配列にDNAの主溝側から結合する。結合したMetJ二量体は、オペレーター配列の相補鎖に結合した他のMetJ二量体とαヘリックスを介して相互作用する。
Met boxはAGACGTCTからなる回文配列で、DNAのどちらの鎖でも同じ配列が認識される。Met boxの中央に位置するCとGの間の接合部にはピリミジン-プリンステップが含まれており、ホスホジエステル骨格が屈曲して正の超らせんが形成される。タンパク質は、DNA二重らせんの形状がタンパク質の形状に沿うことで、認識部位を確認する。言い換えると、タンパク質は特定の塩基配列を認識するのではなく、DNAの構造パラメータを間接的に読み取ることで認識が行われる。
MetJ二量体には2つの補因子S-アデノシルメチオニン(SAM)結合部位が存在する。SAMはメチオニン生合成の産物である。MetJのSAM結合ポケットはDNA結合部位とは重複していない。SAMはMetJに結合してMetJのオペレーター部位への親和性を高め、メチオニン合成に関係する遺伝子の転写を停止させる。SAM濃度が低くなると、リプレッサーはオペレーター部位から解離し、より多くのメチオニンが合成されるようになる。
L-アラビノースオペロンリプレッサー
L-アラビノースオペロンにはアラビノースを分解する酵素が含まれている。これらの酵素は、グルコース濃度が低いか存在しないときに代替的なエネルギー源としてアラビノースを分解する役割を果たす[3]。オペロンはリプレッサー遺伝子(araC)、4つの制御部位(araO2、araO1、araI1、araI2)、2つのプロモーター(Parac/ParaBAD)、3つの構造遺伝子(araBAD)から構成される。AraCは合成されると制御部位に結合してループを形成し、RNAポリメラーゼのプロモーター領域への結合と構造遺伝子の転写を防ぐリプレッサーとして機能する。
アラビノースもAraCも存在しない場合、ループ形成は開始されないが構造遺伝子の発現は低い状態である。アラビノースは存在しないがAraCが存在する場合、AraCは二量体を形成してaraO2とaraI1に結合し、ループ形成によってaraO2とaraI1を近接させる[4]。アラビノースとAraCの双方が存在する場合、AraCはアラビノースと結合し、アクチベーターとして機能する。AraCはアラビノースの結合に伴うコンフォメーション変化によってループを形成することができなくなり、線形となった遺伝子領域はaraBAD領域へのRNAポリメラーゼのリクルートを促進する[3]。

Flowing Locus C(エピジェネティックリプレッサー)
シロイヌナズナArabidopsis thalianaのFlowering Locus C(FLC)オペロンは、開花のためのメリステムの発達に必要な遺伝子を抑制することで、開花を負に制御している。FLCの発現はFRIGIDAの存在によって調節されていることが示されており、温度の低下と負に相関して春化を防いでいる[5]。発現低下の程度は、季節の進行に伴う温度や露光時間の変化に依存している。FLCの発現がダウンレギュレーションされると、開花の可能性が出てくる。FLCの発現調節には、遺伝的な要因とヒストンのメチル化やDNAメチル化などのエピジェネティックな要因が関係している[6]。さらに、FLC遺伝子の負の転写因子として働くコファクターとなる遺伝子も多数存在する[7]。また、FLC遺伝子には種を超えて多数の相同な遺伝子が存在し、さまざまな気候での適応を可能にしている[8]。
出典
- ^ a b Slonczewski, Joan (2009). Microbiology : an evolving science. John Watkins Foster (1st ed ed.). New York: W.W. Norton & Co. ISBN 978-0-393-97857-5. OCLC 185042615
- ^ Somers & Phillips (1992). “Crystal structure of the met repressor-operator complex at 2.8 A resolution reveals DNA recognition by beta-strands.”. Nature 359 (6394): 387–393. doi:10.1038/359387a0. PMID 1406951.
- ^ a b Voet, Donald (2011). Biochemistry. Voet, Judith G. (4th ed.). Hoboken, NJ: John Wiley & Sons. ISBN 978-0-470-57095-1. OCLC 690489261
- ^ Harmer, Tara; Wu, Martin; Schleif, Robert (2001-01-16). “The role of rigidity in DNA looping-unlooping by AraC” (英語). Proceedings of the National Academy of Sciences 98 (2): 427–431. Bibcode: 2001PNAS...98..427H. doi:10.1073/pnas.98.2.427. ISSN 0027-8424. PMC 14602. PMID 11209047.
- ^ Shindo, Chikako; Aranzana, Maria Jose; Lister, Clare; Baxter, Catherine; Nicholls, Colin; Nordborg, Magnus; Dean, Caroline (June 2005). “Role of FRIGIDA and FLOWERING LOCUS C in Determining Variation in Flowering Time of Arabidopsis”. Plant Physiology 138 (2): 1163–1173. doi:10.1104/pp.105.061309. ISSN 0032-0889. PMC 1150429. PMID 15908596.
- ^ Johanson, U.; West, J.; Lister, C.; Michaels, S.; Amasino, R.; Dean, C. (2000-10-13). “Molecular analysis of FRIGIDA, a major determinant of natural variation in Arabidopsis flowering time”. Science 290 (5490): 344–347. Bibcode: 2000Sci...290..344J. doi:10.1126/science.290.5490.344. ISSN 0036-8075. PMID 11030654.
- ^ Finnegan, E. Jean; Kovac, Kathryn A.; Jaligot, Estelle; Sheldon, Candice C.; Peacock, W. James; Dennis, Elizabeth S. (2005). “The downregulation of FLOWERING LOCUS C (FLC) expression in plants with low levels of DNA methylation and by vernalization occurs by distinct mechanisms” (英語). The Plant Journal 44 (3): 420–432. doi:10.1111/j.1365-313X.2005.02541.x. ISSN 1365-313X. PMID 16236152.
- ^ Sharma, Neha; Ruelens, Philip; D'hauw, Mariëlla; Maggen, Thomas; Dochy, Niklas; Torfs, Sanne; Kaufmann, Kerstin; Rohde, Antje et al. (February 2017). “A Flowering Locus C Homolog Is a Vernalization-Regulated Repressor in Brachypodium and Is Cold Regulated in Wheat1[OPEN”]. Plant Physiology 173 (2): 1301–1315. doi:10.1104/pp.16.01161. ISSN 0032-0889. PMC 5291021. PMID 28034954.
関連項目
外部リンク
- Repressor Proteins - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス(英語)
