ゲノムライブラリー
ゲノムライブラリー(英: genomic library)は、単一の生物から得られた全ゲノムDNAの集合体である。このDNAは、それぞれが異なるDNAインサートを持った同一ベクターからなる母集団に保存されている。ゲノムライブラリーを構築するためには、生物のDNAを細胞から抽出し、制限酵素で消化してDNAを特定のサイズのフラグメント(断片)に切断する。その後、このフラグメントをDNAリガーゼを用いてベクターに挿入する[1]。次に、ベクターDNAは宿主生物(一般的には大腸菌や酵母の集団)に取り込まれ、各細胞には1つのベクター分子のみ含まれる。宿主細胞を使用してベクターを運ぶことで、分析のためにライブラリーから特定のクローンを容易に増幅したり、検索することができる[2]。
さまざまなインサート容量を持った数種類のベクターが使用できる。一般的に、より大きなゲノムを持つ生物から作成されたライブラリーは、より大きなインサート容量を特徴とするベクターを必要とするため、ライブラリーを作成するために必要なベクター分子の選択肢は少なくなる。研究者は、完全なゲノムカバレッジに必要な所望のクローン数を見つけ、理想的なインサートサイズも考慮してベクターを選択することができる[3]。
ゲノムライブラリーは、一般的にシークエンシング・アプリケーションに使用され、ヒトゲノムやいくつかのモデル生物を含む、いくつかの生物の全ゲノム・シークエンシング(ゲノム配列決定)の実現において重要な役割を果たしてきた[4][5]。
ゲノムそのものを直接取り扱うことは非効率かつ困難なため、このようなライブラリー化が行われる。
歴史
[編集]これまでにない完全に配列決定された最初のDNAベースのゲノムは、1977年、2度のノーベル賞受賞者であるフレデリック・サンガーによって達成された。サンガーと彼の科学者チームは、DNAシークエンシング(DNA配列決定)で使用するためバクテリオファージ Phi X 174のライブラリーを作成した[6]。この成功の重要性は、遺伝子治療を研究するためにゲノム配列決定に対する需要の高まりへの貢献である。チームは現在、ゲノム中の多型をカタログ化して、パーキンソン病、アルツハイマー病、多発性硬化症、関節リウマチ、1型糖尿病などの疾患の原因となる候補遺伝子を調査することができるようになった[7]。これらは、ゲノムライブラリーの作成と配列決定が可能になったことから、ゲノムワイド関連研究の進歩によるものである。以前は、リンケージや候補遺伝子の研究が、いくつかの唯一のアプローチであった[8]。
ゲノムライブラリーの構築
[編集]ゲノムライブラリーの構築には、多数の組換えDNA分子を作成する必要がある。生物のゲノムDNAを抽出し、制限酵素で消化する。非常に小さなゲノム(〜10 kb)を持つ生物の場合、消化されたフラグメントは、ゲル電気泳動で分離でき、分離されたフラグメントを切り出し、別々にベクターにクローニングできる。しかし、大きなゲノムを制限酵素で消化すると、個々に切除するにはあまりにも多くのフラグメントが生じる。フラグメントの集合全体をベクターと一緒にクローニングしなければならず、その後クローンを分離する必要性が生じる。いずれの場合も、フラグメントは、同じ制限酵素で消化されたベクターに連結される。その後、挿入されたゲノムDNAフラグメントを持つベクターを宿主生物に導入することができる[1]。
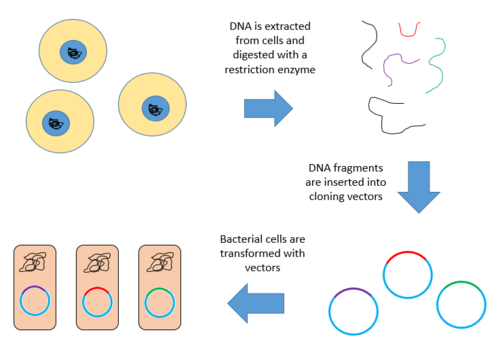
次は、大きなゲノムからゲノムライブラリーを作成するための手順である。
- DNAを抽出し、精製する。
- 制限酵素でDNAを消化する。これにより、大きさが似ており、それぞれが1つ以上の遺伝子を含むフラグメントが作成される。
- このDNAフラグメントを、同じ制限酵素で切断したベクターに挿入する。酵素DNAリガーゼを使用して、DNAフラグメントをベクターに封入する。これにより、組換え分子の大きなプール(貯蔵所)ができる。
- これらの組換え分子は、形質転換によって宿主細菌に取り込まれ、DNAライブラリーを作成する[9][10]。
以下に、上記の手順の概略を示す。
ライブラリーの力価の決定
[編集]ラムダファージなどのウイルスベクターを用いてゲノムライブラリーを構築した後、ライブラリーの力価を決定することができる。力価を計算することで、研究者はライブラリー内で正常に作成された感染性ウイルス粒子の数を概算できる。これを行うには、ライブラリーの希釈液を使用して、既知の濃度の大腸菌の培養物を形質転換する。次に、培養物を寒天プレート上に散布し、一晩培養する。ウイルスプラークの数をカウントして、ライブラリー内の感染性ウイルス粒子の総数を計算することができる。ほとんどのウイルスベクターは、インサートを含むクローンをインサートを持たないクローンと区別できるマーカーも備えている。これにより、研究者は、ライブラリーのフラグメントを実際に担持している感染性ウイルス粒子の割合を決定することもできる[11]。
同様の方法を使用して、プラスミドや細菌人工染色体(BAC)などの非ウイルス性ベクターで作成されたゲノムライブラリーの力価測定できる。ライブラリーの試験ライゲーション(連結)は、大腸菌の形質転換で使用することができる。次に、形質転換体を寒天プレート上に散布し、一晩培養する。形質転換の力価は、プレート上に存在するコロニーの数を数えることによって決定される。これらのベクターは一般的に、インサートを含むクローンと含まないクローンを区別できるようにする選択マーカーを持っている。このテストを行うことで、研究者は連結の効率を判断し、必要に応じて調整を行って、ライブラリーに必要な数のクローンを確実に取得することができる[12]。
ライブラリーのスクリーニング
[編集]
ライブラリーから関心のある領域を含むクローンを単離するためには、最初にライブラリーをスクリーニングする必要がある。スクリーニングの一つの方法は、ハイブリダイゼーションである。ライブラリーの各形質転換された宿主細胞は、1個のDNAインサートを持った1個のベクターのみが含まれることになる。ライブラリー全体を、培地上のフィルター上にプレーティング(塗布)することができる。フィルターとコロニーはハイブリダイゼーション用に準備され、次いでプローブで標識される[13]。下図のようにプローブとのハイブリダイゼーションにより、オートラジオグラフィーなどの検出法によって、目的とする標的DNAインサートを同定することができる。
スクリーニングの別の方法は、ポリメラーゼ連鎖反応(PCR)を用いるものである。ライブラリーの中にはクローンのプールとして保存されているものもあり、PCRによるスクリーニングは、特定のクローンを含むプールを特定するための効率的な方法である[2]。
ベクターの種類
[編集]ゲノムサイズは生物によって異なり、それに応じてクローニングベクターを選択する必要がある。大きなゲノムの場合は、比較的少数のクローンでゲノム全体をカバーできるように、高容量のベクターを選択する必要がある。しかし、より高容量のベクターに含まれるインサートの特徴付けは、しばしばより困難になる[3]。
次の表は、ゲノムライブラリーに一般的に使用される数種類のベクターと、各ベクターが一般的に保持するインサートサイズを示す。
| ベクター種類 | インサートサイズ (1,000塩基対単位) |
|---|---|
| プラスミド | 10まで |
| ラムダファージ (λ) | 25まで |
| コスミド | 45まで |
| バクテリオファージ (P1) | 70~100 |
| P1ファージ由来人工染色体(PAC) | 130~150 |
| 細菌人工染色体(BAC) | 120~300 |
| 酵母人工染色体(YAC) | 250~2000 |
プラスミド
[編集]プラスミドは、分子クローニングで一般的に使用される二本鎖環状DNA分子である。プラスミドの長さは一般的に2~4 k塩基対(kb)で、最大15 kbまでのインサートを運ぶことができる。プラスミドには複製起点が含まれており、宿主の染色体とは無関係に細菌内で複製することができる。プラスミドは一般に、プラスミドを含む細菌細胞の選択を可能にする抗生物質耐性の遺伝子を持っている。多くのプラスミドは、研究者がインサートを含むクローンと含まないクローンを区別することを可能にするレポーター遺伝子も持っている[3]。
ファージラムダ(λ)
[編集]ファージλは、大腸菌に感染する二本鎖DNAウイルスである。λ染色体の長さは48.5 kbで、最大25 kbのインサートを運ぶことができる。これらのインサートは、λ染色体の必須ではないウイルス配列を置き換えるが、ウイルス粒子の形成や感染に必要な遺伝子がそのまま残る。インサートDNAは、ウイルスDNAとともに複製される。このようにして、それらは一緒にウイルス粒子にパッケージングされる。これらの粒子は、感染と増殖において非常に効率的であり、組換えλ染色体の産生量の増加をもたらす[3]。ただし、インサートサイズが小さいため、λファージで作成されたライブラリーでは、完全なゲノムカバレッジのために多くのクローンが必要となる場合がある[14]。
コスミド
[編集]コスミドベクターは、cos配列と呼ばれるバクテリオファージλ DNAの小さな領域を含むプラスミドである。この配列により、コスミドをバクテリオファージλ粒子にパッケージ化することができる。線形化されたコスミドを含むこれらの粒子は、形質導入によって宿主細胞に導入される。宿主内に入ると、コスミドは、宿主のDNAリガーゼの助けを借りて環状化し、プラスミドとして機能する。コスミドは最大40 kbのサイズのインサートを運ぶことができる[2]。
バクテリオファージP1ベクター
[編集]バクテリオファージ (P1)ベクターは、70~100 kbのサイズのインサートを保持することができる。それらは、バクテリオファージP1粒子にパッケージングされた線状DNA分子として始まる。これらの粒子は、Cre組換え酵素を発現する大腸菌株に注入される。線形P1ベクターは、ベクター内の2つのloxP部位間の組換えによって環状化される。P1ベクターには一般に、抗生物質耐性遺伝子と、インサートを含むクローンと含まないクローンとを区別するためのポジティブ選択マーカーが含まれる。P1ベクターには、P1プラスミドレプリコンも含まれているため、細胞内にベクターのコピーが1つのみ存在することを確実にする。しかし、誘導性プロモーターによって制御される、第二のP1レプリコン(P1溶解性レプリコンと呼ばれる)が存在する。このプロモーターにより、DNA抽出の前に、細胞ごとにベクターの複数のコピーを増幅することができる[2]。
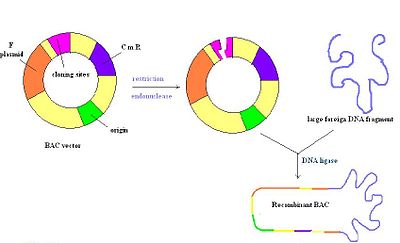
P1人工染色体
[編集]P1人工染色体(PAC)は、P1ベクターと細菌人工染色体(BAC)の両方の特徴を持っている。P1ベクターと同様に、上記のようにプラスミドと溶解レプリコンが含まれている。P1ベクターとは異なり、形質導入のためにバクテリオファージ粒子にパッケージ化する必要はない。代わりに、BACと同様に、エレクトロポレーションを介して環状DNA分子として大腸菌に導入される[2]。また、BACに似ているが、これらは単一複製起点のため、準備が比較的困難である[14]。
細菌人工染色体
[編集]細菌人工染色体(BAC)は、通常約7 kbの長さの環状DNA分子であり、最大300 kbのサイズのインサートを保持することができる。BACベクターには大腸菌F因子に由来するレプリコンが含まれているため、細胞ごとに1コピーが維持される[4]。インサートがBACに連結されると、BACはエレクトロポレーションによって大腸菌の組換え欠損株に導入される。ほとんどのBACベクターには、抗生物質耐性遺伝子とポジティブ選択マーカーが含まれている[2]。右の図は、制限酵素で切断されたBACベクターと、それに続くリガーゼによって再アニーリングされた外来DNAの挿入を示している。全体として、これは非常に安定したベクターであるが、PACのように複製起点が単一であるため、調整が難しい場合がある[14]。
酵母人工染色体
[編集]酵母人工染色体(YAC)は、テロメア、セントロメア、複製起点など、本物の酵母染色体に必要な機能を含む線状DNA分子である。DNAの大きなインサートをYACの中央に連結して、インサートの両側にYACの「腕」を配置できる。組換えYACは形質転換によって酵母に導入され、YAC中に存在する選択マーカーによって、成功した形質転換体の同定が可能になる。YACは2000 kbまでのインサートを保持できるが、ほとんどのYACライブラリーは250〜400 kbのサイズのインサートを保持している。理論的には、YACが保持できるインサートのサイズに上限はない。サイズの上限を決定するのは、インサートに使用するDNAの調製における品質である[2]。YACを使用する上で最も難しいのは、YACが再配列されやすいという事実である[14]。
ベクターの選び方
[編集]ベクター選択では、作成されたライブラリーがゲノム全体を代表するものであることを確認する必要がある。制限酵素に由来するゲノムの任意のインサートは、他のインサートと比較して、ライブラリーに含まれる確率が等しくなければならない。さらに、組換え分子は、ライブラリーのサイズを便利に取り扱えように、十分な大きさのインサートを含むべきである[14]。これは特に、ライブラリーに必要なクローンの数によって決定される。すべての遺伝子のサンプリングを得るために必要なクローンの数は、生物のゲノムのサイズと平均インサートサイズによって決定される。これは、次の式で表される(Carbon and Clarke式とも呼ばれる)[15]。
ここに、
は組換え体の必要数[16]、
は、作成されたライブラリーの中で、ゲノム中の任意のフラグメントが少なくとも一度は発生する確率、
は、単一の組替え体中のゲノムの割合を表す。
は、さらに次のように表すことができる。
ここに、
は挿入サイズ、
はゲノムサイズである。
したがって、(ベクターの選択によって)インサートサイズを大きくすると、ゲノムを表すために必要なクローン数が少なくなる。インサートサイズとゲノムの大きさの比率は、単一クローン内のそれぞれのゲノムの比率を表す[14]。以下に、すべての部分を考慮した式を示す。
ベクター選択の例
[編集]上記の式を用いて、2万塩基対のインサートサイズを有するベクター(例:ファージラムダベクター)を用いて、ゲノム中の全ての配列が表現される99%信頼水準を決定することができる。この例では、生物のゲノムサイズは30億塩基対である。
clones
したがって、この30億塩基対のゲノムからの所定のDNA配列が、2万塩基対のインサートサイズのベクターを用いて、99%の確率でライブラリーに存在することを保証するためには、約688,060個のクローンが必要である。
応用
[編集]ライブラリーを作成した後、生物のゲノムを配列決定し、遺伝子が生物にどのような影響を与えるかを解明したり、ゲノムレベルで類似の生物を比較したりすることができる。前述のゲノムワイド関連研究により、多くの機能的形質に由来する候補遺伝子を同定することができる。ゲノムライブラリーを介して遺伝子を単離し、ヒト細胞株や動物モデルを用いてさらなる研究を行うことができる[17]。さらに、安定性の問題がなく、正確なゲノム表現が可能な高忠実度クローンを作成することは、ショットガン・シークエンシング法や機能解析における完全遺伝子の研究のための中間体として十分に貢献できると考えられる[10]。
階層的シークエンシング法
[編集]
ゲノムライブラリーの主要な用途の一つは、階層的ショットガン・シークエンシングである。これはトップダウン、マップベース、またはクローン・バイ・クローン・シークエンシングとも呼ばれている。この手法は、ハイスループット技術が利用可能になる前の1980年代に、全ゲノムの配列決定のために開発された。ゲノムライブラリーからの個々のクローンは、通常500 bpから1000 bpの小さなフラグメントにせん断することができ、シークエンシング処理がより管理しやすくなる[4]。ゲノムライブラリーからのクローンが配列決定されると、その配列を使用して、配列決定されたクローンと重畳するインサートをもつ他のクローンのためにライブラリーをスクリーニングすることができる。重なり合ったクローンの配列決定をして、コンティグ(配列断片群)を形成することができる。染色体ウォーキングと呼ばれるこの技術を利用して、染色体全体を配列決定することができる[2]。
全ゲノム・ショットガン・シークエンシングは、高容量ベクターのライブラリーを必要としない、もう一つのゲノムシークエンシング方法である。この方法では、コンピュータアルゴリズムを使用して、ゲノム全体をカバーするように短鎖シークエンスリードを組み立てる。このような理由から、ゲノムライブラリーは、全ゲノム・ショットガン・シークエンシングと組み合わせて使用されることがよくある。高分解能マップは、ゲノムライブラリー内の複数のクローンからのインサートの両端を配列決定することにより作成することができる。このマップは、既知の距離を隔てた配列を提供し、ショットガン・シークエンシングで取得したシークエンスリードの組み立てに役立つ[4]。2003年に完全であると宣言されたヒトゲノム配列は、BACライブラリーとショットガン・シークエンシングの両方を用いて組み立てられた[18][19]。
ゲノム全域にわたる関連研究
[編集]ゲノムワイド関連研究は、人類内の特定の遺伝子標的や多型を見つけるための一般的な応用である。実際、国際HapMap計画は、このデータをカタログ化して利用するために、数カ国の科学者と機関のパートナーシップによって作成された[20]。このプロジェクトの目的は、異なる個人の遺伝子配列を比較して、染色体領域内の類似点と相違点を明らかにすることである[20]。参加国すべての科学者が、アフリカ系、アジア系、ヨーロッパ系の祖先を持つ集団のデータを用いて、これらの属性をカタログ化している。このようなゲノムワイドな評価は、さらなる診断や薬物治療につながる可能性があり、将来のチームが遺伝的特徴を考慮した治療法の調整に注力するのにも役立つだろう。これらの概念は、すでに遺伝子工学の分野で活用されている[20]。たとえば、ある研究チームは実際にPACシャトルベクターを構築し、ヒトゲノムの2倍のカバレッジを持つライブラリーを作成した[17]。これは、病気の原因となる遺伝子やその集合を特定するための驚異的なリソースとして役立つ可能性がある。さらに、これらの研究は、バキュロウイルスの研究で見られるように、転写制御を調査するための強力な方法として役立つ可能性がある[21]。全体的に、ゲノムライブラリー構築とDNA配列決定の進歩により、さまざまな分子標的の効率的な発見が可能になった[5]。これらの効率的な方法によって特徴を同化することで、新薬候補の採用を早めることができる。
脚注
[編集]- ^ a b Losick, Richard; Watson, James D.; Tania A. Baker; Bell, Stephen; Gann, Alexander; Levine, Michael W. (2008). Molecular biology of the gene. San Francisco: Pearson/Benjamin Cummings. ISBN 978-0-8053-9592-1
- ^ a b c d e f g h Russell, David W.; Sambrook, Joseph (2001). Molecular cloning: a laboratory manual. Cold Spring Harbor, N.Y: Cold Spring Harbor Laboratory. ISBN 978-0-87969-577-4
- ^ a b c d Hartwell, Leland (2008). Genetics: from genes to genomes. Boston: McGraw-Hill Higher Education. ISBN 978-0-07-284846-5
- ^ a b c d Muse, Spencer V.; Gibson, Greg (2004). A primer of genome science. Sunderland, Mass: Sinauer Associates. ISBN 978-0-87893-232-0
- ^ a b Henry RJ, Edwards M, Waters DL, etal (November 2012). “Application of large-scale sequencing to marker discovery in plants”. J. Biosci. 37 (5): 829–41. doi:10.1007/s12038-012-9253-z. PMID 23107919.
- ^ Sanger F, Air GM, Barrell BG, etal (February 1977). “Nucleotide sequence of bacteriophage phi X174 DNA”. Nature 265 (5596): 687–95. Bibcode: 1977Natur.265..687S. doi:10.1038/265687a0. PMID 870828.
- ^ Menon R, Farina C (2011). “Shared molecular and functional frameworks among five complex human disorders: a comparative study on interactomes linked to susceptibility genes”. PLOS ONE 6 (4): e18660. Bibcode: 2011PLoSO...618660M. doi:10.1371/journal.pone.0018660. PMC 3080867. PMID 21533026.
- ^ Cichon S, Mühleisen TW, Degenhardt FA, etal (March 2011). “Genome-wide association study identifies genetic variation in neurocan as a susceptibility factor for bipolar disorder”. Am. J. Hum. Genet. 88 (3): 372–81. doi:10.1016/j.ajhg.2011.01.017. PMC 3059436. PMID 21353194.
- ^ Yoo EY, Kim S, Kim JY, Kim BD (August 2001). “Construction and characterization of a bacterial artificial chromosome library from chili pepper”. Mol. Cells 12 (1): 117–20. PMID 11561720.
- ^ a b Osoegawa K, de Jong PJ, Frengen E, Ioannou PA (May 2001). “Construction of bacterial artificial chromosome (BAC/PAC) libraries”. Curr Protoc Hum Genet Chapter 5: 5.15.1–5.15.33. doi:10.1002/0471142905.hg0515s21. PMID 18428289.
- ^ John R. McCarrey; Williams, Steven J.; Barton E. Slatko (2006). Laboratory investigations in molecular biology. Boston: Jones and Bartlett Publishers. ISBN 978-0-7637-3329-2
- ^ Peterson, Daniel; Jeffrey Tomkins; David Frisch (2000). “Construction of Plant Bacterial Artificial Chromosome (BAC) Libraries: An Illustrated Guide”. Journal of Agricultural Genomics 5.
- ^ Kim UJ, Birren BW, Slepak T, etal (June 1996). “Construction and characterization of a human bacterial artificial chromosome library”. Genomics 34 (2): 213–8. doi:10.1006/geno.1996.0268. PMID 8661051.
- ^ a b c d e f “Cloning Genomic DNA”. University College London. 13 March 2013閲覧。[リンク切れ]
- ^ “Archived copy”. 2013年3月31日時点のオリジナルよりアーカイブ。2013年6月5日閲覧。
- ^ Blaber, Michael. “Genomic Libraries”. 1 April 2013閲覧。
- ^ a b Fuesler J, Nagahama Y, Szulewski J, Mundorff J, Bireley S, Coren JS (April 2012). “An arrayed human genomic library constructed in the PAC shuttle vector pJCPAC-Mam2 for genome-wide association studies and gene therapy”. Gene 496 (2): 103–9. doi:10.1016/j.gene.2012.01.011. PMC 3488463. PMID 22285925.
- ^ Pareek CS, Smoczynski R, Tretyn A (November 2011). “Sequencing technologies and genome sequencing”. J. Appl. Genet. 52 (4): 413–35. doi:10.1007/s13353-011-0057-x. PMC 3189340. PMID 21698376.
- ^ Pennisi E (April 2003). “Human genome. Reaching their goal early, sequencing labs celebrate”. Science 300 (5618): 409. doi:10.1126/science.300.5618.409. PMID 12702850.
- ^ a b c “HapMap Homepage”. 2021年2月13日閲覧。
- ^ Chen Y, Lin X, Yi Y, Lu Y, Zhang Z (2009). “Construction and application of a baculovirus genomic library”. Z. Naturforsch. C 64 (7–8): 574–80. doi:10.1515/znc-2009-7-817. PMID 19791511.
推薦文献
[編集]Klug, Cummings, Spencer, Palladino (2010). Essentials of Genetics. Pearson. pp. 355–264. ISBN 978-0-321-61869-6









![{\displaystyle N={\frac {ln(1-0.99)}{ln[1-{\frac {2.0\times 10^{4}basepairs}{3.0\times 10^{9}basepairs}}]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/053b596ba7adfc402824b91ceae3dd24973b2fec)

