HLA-A

HLA-Aは、ヒトの染色体6p21.3に位置するHLA-A遺伝子座にコードされる、ヒト白血球抗原(HLA)の1グループである[1]。HLAはヒト特異的な主要組織適合遺伝子複合体(MHC)抗原である。HLA-AはヒトのMHCクラスI分子の主要な3つのタイプのうちの1つ(他の2つはHLA-B、HLA-C)である[2]。MHCクラスI分子は大きなα鎖と小さなβ鎖からなるヘテロ二量体である。HLA-A遺伝子のさまざまな多型はα鎖をコードしており、β鎖は全て同じβ2-ミクログロブリン分子である[3]。β2-ミクログロブリンは、ヒトでは染色体15q21.1に位置するB2M遺伝子にコードされている[4][5]。
HLA-AなどのMHCクラスI分子は、免疫系に短いペプチドを提示する過程(抗原提示過程)の一部を構成している。こうしたペプチドは一般的には7–11アミノ酸長で、細胞で発現しているタンパク質に由来する(自己の)ものである場合も、外因性タンパク質に由来する(非自己の)ものである場合もある。正常な条件下では、血液循環を介して体内を巡回している細胞傷害性T細胞がこの複合体によって提示されているペプチドを「読み取り」、非自己ペプチドを提示しているものにのみ結合する。結合が生じた場合には、アポトーシスを介した細胞死に至る一連のイベントが開始される[6]。人体はこのようにして、ウイルスが感染した細胞や発現すべきでないタンパク質を発現している細胞(がん細胞など)を除去している。
大部分の哺乳類と同様、ヒトのMHCクラスI分子の一次構造は非常に多様であり、HLA-Aはヒトの遺伝子の中で最も早く進化しているコーディング配列のうちの1つとなっている。2022年3月時点で、HLA-Aのアレルは7452種類が知られており、4305種類が活性タンパク質をコードし、375種類は機能的なタンパク質をコードしない。こうしたMHCクラスI分子の多様性は移植拒絶反応の主要因となっており、ドナーとレシピエントをランダムに選んだ場合にHLA-A、B、C抗原がマッチする可能性はほとんどない。また、進化生物学者はHLAの高度な多様性は相反する病原体の圧力の間での均衡の結果であると考えている。HLAの多様性が高ければ、各病原体に対して高い抵抗性を持つ人物が存在する可能性が高くなり、1種類の病原体によって集団全体が消滅する確率は低下する[7]。
遺伝子[編集]
HLA-A遺伝子は6番染色体の短腕に位置し、HLA-Aの大きなα鎖をコードしている。HLA-Aのα鎖の多型はHLAの機能に重要であり、また集団の遺伝的多様性を促進している。各HLAはそれぞれ異なる構造を持つペプチドに対して異なる親和性を有するため、HLAの多様性が高ければ高いほど細胞表面に提示される抗原の多様性は高くなり、特定の侵入者に対して集団の一部が抵抗性を持つ可能性は高くなる。
各個人は、両親から受け継いだ、最大2種類のHLA-Aを発現している。一部の人物は両親から同じHLA-Aを受け継ぐため、その人のHLAの多様性は低下するが、大部分の人は2つの異なるコピーを受け取る。こうしたパターンは他の全てのHLA群にも共通している[12]。言い換えると、既知の数千種類のHLA-Aアレルのうち、各個人が発現するのは1種類もしくは2種類のみである。
アレル[編集]
全てのHLAは、 WHOの命名委員会(World Health Organization Naming Committee for Factors of the HLA System)によって名称が割り当てられている。この名称は、特定のアレルに関する最大限の情報を保持しつつ、できるだけ短くなるように構成されている。
各HLAの名称は「HLA-A*02:01:01:02L」のような形をしている。全てのアレルには少なくとも4桁での分類がなされている(例: HLA-A*02:12)。HLA-の後の「A」はそのアレルがどのHLA遺伝子に属しているのかを表している。HLA-Aには多くのアレルが存在するため血清型による分類がなされており、*に続く2桁の数字はこの割り当てを示している。例えば、HLA-A*02:02やHLA-A*02:04、HLA-A*02:324は全てA2血清型のメンバーである[2]。この血清型による分類がHLAの適合性と関係する主要な因子である。:で区切られた後の数字は血清型分類によっては決定することができないものであり、遺伝子のシークエンシングによって指定されている。この数字はどのHLAタンパク質が産生されているかを示しており、数字は発見の順序によって割り当てられている。2013年12月時点でHLA-A*02タンパク質には456種類が知られており、HLA-A*02:01からHLA-A*02:456までが割り当てられている。最も短いHLAの名称にも、こうした詳細が含まれている[1]。それ以降の数字や文字は、コーディング領域内の同義置換や領域外の変異などを表している。
タンパク質[編集]
HLA-A遺伝子には365アミノ酸からなる、約41 kDaのタンパク質がコードされている[13]。この遺伝子は8つのエクソンを含む[14]。
| エクソン | 領域 |
|---|---|
| 1 | シグナルペプチド |
| 2 | α1ドメイン |
| 3 | α2ドメイン |
| 4 | α3ドメイン |
| 5 | 膜貫通領域 |
| 6 | 細胞質テール |
| 7 | 細胞質テール |
| 8 | 不明 |
HLA-Aのシグナルペプチドは一連の疎水的アミノ酸からなり、N末端に位置してタンパク質を小胞体へ差し向ける。そして、残りの7ドメインは小胞体上で翻訳が行われる[13][14][15]。3つのαドメインは、CD8+T細胞へ提示されるペプチドを保持する溝を形成する。膜貫通領域は、小胞体内腔を囲むリン脂質二重層に埋め込まれる領域である[14]。HLA-Aは1回膜貫通タンパク質であり[13]、最初の4つのドメインが小胞体内腔に、最後の3つのドメインが小胞体外に位置することで、タンパク質の適切な機能に必要な配向が形成される。最後の3つのドメインは主にβシートからなるテールを形成し、細胞質基質にとどまる[14]。
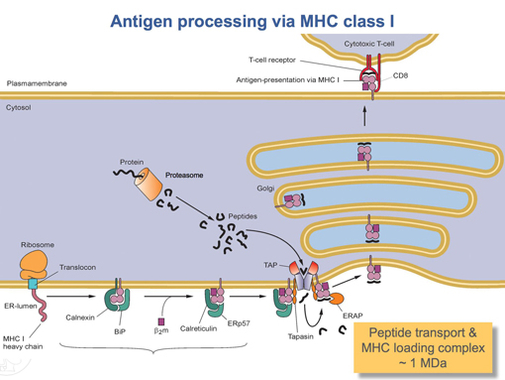
HLA-Aタンパク質の翻訳の完了後には、適切な形状へのフォールディングが必要である。カルネキシンと呼ばれるシャペロンタンパク質とERp57と呼ばれる酵素がこのフォールディング過程を補助する。カルネキシンはHLA-Aのα鎖を保持し、ERp57はα鎖とβ鎖(β2-ミクログロブリン)との間のジスルフィド結合の形成を触媒する。この結合によってα鎖のコンフォメーション変化が誘導され、ペプチドを保持するための溝が形成される。その後、カルネキシンは解離して別のシャペロンタンパク質カルレティキュリンに置き換わり、この複合体はペプチドローディング複合体(PLC)と呼ばれるようになる。短いペプチド断片は、TAPと呼ばれる特殊なトランスポーターによって、細胞周囲から小胞体内腔へ絶えず輸送されている。TAPはタパシンと呼ばれるタンパク質とともにPLCに結合する。この時点のPLCには、HLA-A(α鎖)、β2-ミクログロブリン(β鎖)、ERp57、カルレティキュリン、TAP(とそれに結合したペプチド断片)、タパシンが含まれている。タパシンはTAPの安定性を高めるとともに、PLC全体を安定化する。TAPは小胞体内腔へ輸送されたペプチドを放出するが、PLCの形成はHLA-Aの溝とTAPが近接して位置するよう保証しており、ペプチドが溝に結合する可能性を高めている。ペプチドのHLA-Aに対する親和性が十分に高い場合には、そのペプチドは溝に結合する[17]。タパシンは積極的にTAPからHLA-Aへペプチドをローデングし、また高親和性のペプチドが結合するまでMHCクラスI分子を小胞体内腔に保持する役割を果たしている可能性が研究からは示唆されている[18]。
十分に高い親和性のペプチドがMHCクラスI分子に結合すると、カルレティキュリン、ERp57、TAP、タパシンはMHCクラスI分子を放出する。この時点のMHCクラスI分子はHLA-Aタンパク質とそれに結合したβ2-ミクログロブリン、そして短いペプチドから構成されているが、HLA-Aの膜貫通ドメインによって小胞体膜に固定されている。何らかの時点で小胞体がシグナルを受け取ると、この複合体を保持している膜領域が出芽し、さらなるプロセシングのために輸送される。この複合体は小胞輸送によって、ゴルジ体を経て細胞膜へ輸送される。小胞の細胞膜への融合過程で、ペプチドを保持している部分を含む小胞体内腔側が細胞外側に露出する[15]。
機能[編集]
MHCクラスI分子は、一般的に7–10アミノ酸の長さからなる小さなペプチドを免疫系に提示する。CD8と呼ばれる糖タンパク質は、HLA-Aのα3ドメインの223–229番残基に結合し、細胞傷害性(CD8+)T細胞の上のT細胞受容体とMHCクラスI分子との相互作用を安定化する[19]。T細胞受容体はMHCによって提示されているペプチドに結合する能力を持つ。正しく機能している免疫系では自己ペプチドに結合しないT細胞のみが胸腺外に出ることができるため、T細胞がペプチドに結合する場合、そのペプチドは外来の因子、もしくは異常なタンパク質に由来するペプチドである。その後、T細胞はアポトーシス(プログラム細胞死)を開始させる。この過程は外来抗原の提示から早くも5分後には生じることがあるが、細胞死が顕著になるまでには数時間程度かかるのが一般的である[20]。この過程は獲得免疫の基礎をなしており、ウイルスやその他の細胞内病原体に対する主要な防御機構となっている。
移植された器官や組織上の因子によって、ホストの免疫系による移植組織の破壊が引き起こされていることが1960年代までに明らかとなり、MHCはこうした観察から発見された[7]。ペプチドを提示する複合体にはクラスIとクラスIIの2種類があり、それぞれ複数のHLA遺伝子にコードされている。HLA-Aはその1つである。ドナーとレシピエントの間で一致すべきHLAには、HLA-A、HLA-B(これらはクラスI)、HLA-DR(クラスII)の3つの主要なタイプがある。ドナーとレシピエントの組織でこれら3つのHLAをコードする遺伝子が同じである場合、拒絶反応は最も弱くなる[21]。
疾患における役割[編集]
| 疾患 | 血清型 | ||
| 強直性脊椎炎 | A24 | ||
| 1型糖尿病[22] | A1 | A24 | |
| ヘモクロマトーシス (CD8+細胞の減少) | A3 | ||
| 重症筋無力症 | A3 | A24 | A30 |
| 成人T細胞白血病 | A26 | A68 | |
| 多発性硬化症 | A3 | ||
| パピローマウイルス感受性 | A11 | ||
| 自然流産 | A2 | ||
HLAは、細胞内の現象と免疫系をつなぐ唯一の役割を果たしている。そのため、HLAの一部に変化が生じると、特定のペプチドとの結合が低下したり高まったりすることで、病気にかかりやすくなったりかかりにくくなったりといった表現型が生じることがある。特定のHLAは病原性タンパク質の分解によって生じる短いペプチドのいずれとも結合できない可能性がある。そうした場合には、免疫系は細胞が感染していることを知ることはできず、感染はほとんど抑制されることなく増大する。また、その逆が生じる場合もある。一部のHLAは病原性ペプチド断片と非常に高い親和性で結合する。このことはその特定の病原体に対して免疫系を実質的に強化することとなり、壊滅的な害を及ぼしかねない感染症への対処が行えるようになる[7]。
HIV/AIDS[編集]
病原体に対する免疫調節の差異について最もよく研究されている例の1つが、ヒト免疫不全ウイルス(HIV)である。HIVはRNAウイルスであるため、非常に迅速に変異が生じる。その結果、タンパク質分解によって産生されるペプチドが変化し、感染細胞のMHCによって免疫系に提示されるペプチドも変化する。特定のHLAに対して高い親和性で結合するペプチドが生成される変異を持つウイルスは免疫系によって迅速に死滅させられるために生き残ることができず、そうした高親和性ペプチドも産生されなくなる。しかしながら、HIVのゲノム中にも一部保存された領域が存在し、この保存領域から産生されるペプチドに対してHLAが結合できた場合、HIVが免疫系による検知と破壊を回避する術はほとんどない[7]。HLAの差異にによるHIVのウイルス量の差異の根底には、こうした原理が存在する。
HLA-AによってコードされるMHCには2000種類以上の多様性が存在し、全ての種類についてHIVのウイルス量に対する影響を明らかにすることは困難である。しかしながら、いくつかの種類に関してはウイルス量との関係が示唆されている。HLA-A*30は、ウイルス量を10,000コピー/mm3以下というかなり低い値に抑えることが示されている。一方HLA-A*02は、HLA-B*45を共に持つ場合に高ウイルス量(100,000コピー/mm3以上)と関係している。ザンビアの集団内ではハプロタイプHLA-A*23-C*07とHLA-A*02-C*16は一般的に高ウイルス量と関係している。最も効果的にHIVを阻害するハプロタイプはHLA-A*30-C*03であり、一方最も効果が低いのはHLA-A*23-B*14である。一般的に、サンプル集団内ではHLA-A*23は高ウイルス量との高い相関を示すが、民族の異なる集団間ではこの相関は大きく低下することに留意しておく必要がある[23]。
個々のHLA遺伝子やアレルのHIVに対する影響を分類することは困難であるが、いくつかの有力な結論は得られている。1つもしくは複数のクラスI HLA遺伝子がホモ接合型となっている人物は、ヘテロ接合型の人物よりもかなり急速にAIDSへと進行するのが一般的であり、一部のホモ接合型ではヘテロ接合型の進行速度の2倍である。こうした進行の差異は、ヘテロ接合性の程度とかなりの相関がみられる[24]。
特定のアレルがHIVに対する抵抗性の高さや低さを定めているだけでなく、HIVはHLAの発現を変化させることができ、それによってNK細胞によるウイルスの除去を抑制している。HIVは感染細胞でのMKCクラスI分子の発現をダウンレギュレーションするが、NK細胞はHLA-CやHLA-Eのダウンレギュレーションに応答するため、こうした変化はNK細胞による攻撃の機会を開くこととなる。この機構は明らかにHIVに対する選択圧として働いており、HIVはHLA-CやHLA-Eの発現を大きく変化させることなくHLA-AやHLA-Bをダウンレギュレーションする能力を進化させている[25]。HIVのゲノムにコードされるタンパク質Nefは、MHCクラスI分子がまだ小胞体に位置しているとき、そして時にはゴルジ体を介した輸送の初期段階にあるときにMHCクラスI分子の細胞質テールに結合することでこの変化を誘導する。このMHCとNefからなる複合体は、アダプタータンパク質複合体AP-1によってMHCを正常な機能を果たす細胞膜ではなく、分解が行われるリソソームへ差し向ける[26]。選択的なHLAのダウンレギュレーションに加えて、NefはCD4とCD8のダウンレギュレーションを可能にする。これらの糖タンパク質はそれぞれ、ヘルパーT細胞と細胞傷害性T細胞のMHCへの結合に必要不可欠である。これらのコファクターが存在しない場合には、たとえHLAがHIVに由来する(非自己の)ペプチドを提示していても、どちらの種類のT細胞もHLAに結合してアポトーシスを開始させることはできない。どちらのタンパク質も細胞質テールがNefの標的となる[26]。こうした作用の組み合わせによって、HIVは免疫系による検知を回避する能力を大きく高めている。
出典[編集]
- ^ a b “HLA Nomenclature @ hla.alleles.org”. Anthony Nolan Research Institute (2013年11月10日). 2013年12月8日閲覧。
- ^ a b “Statistics”. European Bioinformatics Institute (EBI) / European Molecular Biology Laboratory (EMBL). 2017年9月10日時点のオリジナルよりアーカイブ。2013年12月13日閲覧。
- ^ Delves PJ (2013年8月). “Human Leukocyte Antigen (HLA) System: Biology of the Immune System”. Merck Manual Professional. Merck Sharp & Dohme Corp. 2013年12月14日閲覧。
- ^ “B2M Gene”. GeneCards. Weizmann Institute of Science (2013年11月7日). 2013年12月14日閲覧。
- ^ “OMIM Entry - * 109700 - BETA-2-MICROGLOBULIN;B2M”. Online Mendelian Inheritance in Man. Johns Hopkins University (2016年8月5日). 2021年5月14日閲覧。
- ^ Accorsi D (2012年9月14日). “MHC class I assembly and presentation”. YouTube. 2013年12月8日閲覧。
- ^ a b c d Daniel M. Davis (2014). The Compatibility Gene. How Our Bodies Fight Disease, Attract Others, and Define Our Selves.. Oxford: Oxford University Press. ISBN 978-0-19-931641-0
- ^ a b c GRCh38: Ensembl release 89: ENSG00000206505、ENSG00000224320、ENSG00000206503、ENSG00000223980、ENSG00000229215、ENSG00000227715、ENSG00000235657、ENSG00000231834 - Ensembl, May 2017
- ^ a b c GRCm38: Ensembl release 89: ENSMUSG00000079507 - Ensembl, May 2017
- ^ Human PubMed Reference:
- ^ Mouse PubMed Reference:
- ^ Fix M (1998年). “HLA Matching, Antibodies, and You”. Kidney Transplantation: Past, Present, and Future. University of Michigan Medical Center/Stanford University. 2013年12月14日閲覧。
- ^ a b c “Major Histocompatibility Complex, Class I, A”. Gene Cards. Weizmann Institute of Science (2013年11月7日). 2013年12月16日閲覧。
- ^ a b c d “HLA-A major histocompatibility complex, class I, A [Homo sapiens (human)]”. National Center for Biotechnology Information. U.S. National Library of Medicine (2013年12月12日). 2013年12月16日閲覧。
- ^ a b Alberts, Bruce (2010). Essential Cell Biology (Third ed.). Garland Science. ISBN 9780815341291
- ^ Tampé, Robert. “P16 Translocation mechanism and viral inhibition of the MHC I peptide-loading complex”. Molecular Understanding of Transmembrane Processes. Institute of Biochemistry Biocenter. 2013年12月16日閲覧。
- ^ Accorsi, Diego (2012年9月14日). “MHC class I assembly and presentation”. Immunology Toronto. Department of Immunology and Biochemistry and Biomedical Communications at the University of Toronto. 2013年12月16日閲覧。
- ^ “Tapasin: an ER chaperone that controls MHC class I assembly with peptide”. Trends in Immunology 22 (4): 194–9. (April 2001). doi:10.1016/S1471-4906(01)01861-0. PMID 11274924.
- ^ “CD8”. T-cell Modulation Group. tcells.org (2009年). 2013年2月18日時点のオリジナルよりアーカイブ。2013年12月17日閲覧。
- ^ Janeway, Charles A. (2001). “8”. Immunobiology the immune system health & disease (5. ed.). New York: Garland. ISBN 978-0815336426 2013年12月17日閲覧。
- ^ “Banking on iPSC--is it doable and is it worthwhile”. Stem Cell Reviews 11 (1): 1–10. (February 2015). doi:10.1007/s12015-014-9574-4. PMC 4333229. PMID 25516409.
- ^ “The HLA class I A locus affects susceptibility to type 1 diabetes”. Human Immunology 63 (8): 657–64. (August 2002). doi:10.1016/S0198-8859(02)00421-4. PMC 4049513. PMID 12121673.
- ^ “Favorable and unfavorable HLA class I alleles and haplotypes in Zambians predominantly infected with clade C human immunodeficiency virus type 1”. Journal of Virology 76 (16): 8276–84. (August 2002). doi:10.1128/JVI.76.16.8276-8284.2002. PMC 155130. PMID 12134033.
- ^ “HLA and HIV-1: heterozygote advantage and B*35-Cw*04 disadvantage”. Science 283 (5408): 1748–52. (March 1999). Bibcode: 1999Sci...283.1748C. doi:10.1126/science.283.5408.1748. PMID 10073943.
- ^ “The selective downregulation of class I major histocompatibility complex proteins by HIV-1 protects HIV-infected cells from NK cells”. Immunity 10 (6): 661–71. (June 1999). doi:10.1016/S1074-7613(00)80065-5. PMID 10403641.
- ^ a b “HIV-1 Nef disrupts intracellular trafficking of major histocompatibility complex class I, CD4, CD8, and CD28 by distinct pathways that share common elements”. Journal of Virology 85 (14): 6867–81. (July 2011). doi:10.1128/JVI.00229-11. PMC 3126561. PMID 21543478.
外部リンク[編集]
- Molecular Anthropology Yahoo Group
- HLA Allele and Haplotype Frequency Database
- HLA Nomenclature Full List of Class I Proteins
- Overview of all the structural information available in the PDB for UniProt: P04439 (HLA class I histocompatibility antigen, A alpha chain) at the PDBe-KB.





