SNAREタンパク質

| SNARE-fusion membrane complex proteins | |
|---|---|
| 識別子 | |
| 略号 | SNARE |
| InterPro | IPR010989 |
| SCOP | 1kil |
| SUPERFAMILY | 1kil |
| TCDB | 1.F.1 |
| OPM superfamily | 197 |
| OPM protein | 3hd7 |
| Membranome | 198 |
SNAREタンパク質(スネアタンパクしつ、英: SNARE proteins)は、小胞が標的の膜結合性区画(リソソームなど)へ融合する過程(小胞融合)を媒介するタンパク質である。酵母では少なくとも24種類、哺乳類細胞では60種類以上のメンバーから構成され、SNARE複合体(soluble NSF attachment protein receptor complex)を形成する[2][3]。最もよく研究されているSNAREは、神経細胞においてシナプス小胞が細胞膜(シナプス前膜)へ融合する過程を媒介するものである。また、SNAREタンパク質はボツリヌス症や破傷風を引き起こす細菌の神経毒の標的となっている。
タイプ
[編集]SNAREタンパク質は2つのカテゴリに分類される。v-SNARE(vesicleの略)は出芽する輸送小胞の膜に取り込まれ、t-SNARE(targetの略)は神経終末の膜に結合している。t-SNARE間で安定なサブ複合体が形成され、それらがv-SNAREが結合してSNARE複合体の形成が完了する際のガイドとして機能することが示唆されている[4]。いくつかのSNAREタンパク質は小胞と標的膜の両方に存在しているため、より近年の分類では、SNAREの構造的特徴から、R-SNAREとQ-SNAREへの分類が行われている。多くの場合、R-SNAREはv-SNAREとして機能し、Q-SNAREはt-SNAREとして機能する。組み立てられたSNARE複合体のコア領域では4本のαヘリックスからなる4-ヘリックスバンドルが形成されるが、R-SNAREはコア領域でのzero ionic layerの形成に際し、アルギニン(R)残基を供するタンパク質である。R-SNAREの例はシナプス小胞に位置するシナプトブレビンである。Q-SNAREはzero ionic layerの形成に際しグルタミン(Q)残基を供するタンパク質である。Q-SNAREには、シンタキシンとSNAP-25が含まれる。Q-SNAREは、4-ヘリックスバンドル中の位置に応じてQa、Qb、Qcへとさらに分類される。
構造
[編集]SNAREは低分子量で存在量が多いタンパク質である。一部は尾部アンカー型タンパク質(tail-anchored protein)で、C末端の膜貫通ドメインが翻訳後に膜へ挿入されていることが多い。既知の38種類のSNAREのうち、SNAP-25を含めた7つは膜貫通ドメインを持たず、代わりにパルミトイル化のような脂質修飾を介して膜へ結合している[5]。尾部アンカー型タンパク質は、細胞膜、小胞体、ミトコンドリア、ペルオキシソームやその他の膜へと挿入されるが、各SNAREタンパク質はそれぞれ特定の膜を標的としている。C末端近傍のアミノ酸残基の組成を変えたり、膜貫通ドメインの長さを変えたりすることによって、SNAREタンパク質の標的化は影響を受ける。膜貫通ドメインを脂質アンカーへと置換すると、膜融合の際に2つの近接した層のみが融合し、離れた側の層は融合していない中間的な状態となる[6]。
SNAREの構造やサイズは多様であるが、その細胞質ドメインには60-70アミノ酸からなるSNAREモチーフが共通して存在し、そこにはコイルドコイル構造を形成するhepatad repeat(7アミノ酸からなるリピート構造)が含まれている。v-SNAREとt-SNAREは可逆的に集合し、トランスSNARE複合体(trans-SNARE complex)と呼ばれる強固な4-ヘリックスバンドルを形成する。シナプス小胞で容易に形成される準安定的状態であるトランスSNARE複合体は、細胞膜に位置するシンタキシンとSNAP-25、そして小胞の膜に固定されたシナプトブレビン(VAMP、vesicle-associated membrane proteinとも呼ばれる)から構成される。シンタキシンとシナプトブレビンはC末端ドメインでそれぞれの膜に固定されているが、SNAP-25はいくつかのシステイン残基に結合したパルミトイル鎖を介して細胞膜に結合している。シンタキシンとシナプトブレビンは1本ずつ、SNAP-25は2本のαヘリックスを提供し、4-ヘリックスバンドルが形成される。
細胞膜に位置するSNAREは微小ドメインまたはクラスターとして存在することが示されており、それらは細胞がエキソサイトーシスを完全に行うために必須である。
膜の融合
[編集]
膜融合の過程において、別々の膜に存在するv-SNAREとt-SNAREがトランスSNARE複合体を形成する。トランスSNARE複合体は「SNAREpin」という名称でも知られる。膜の融合の段階に応じて、複合体は異なる名前で呼ばれることがある。
膜の融合後はこの複合体はシスSNARE複合体(cis-SNARE complex)と呼ばれる。なぜなら、この段階ではSNAREタンパク質が同じ(cis)膜上に存在しているからである。融合後、シスSNARE複合体はアダプタータンパク質であるαSNAPによって解体される。その後、六量体AAA-ATPアーゼであるNSFがATP依存的にSNAREタンパク質を巻き戻し、リサイクルのために細胞質へ放出する。
SNAREは膜融合装置の核となる要素であると考えられており、細胞質の付加的な補助タンパク質とは独立に機能する。このことはSNAREドメインが細胞質ではなく細胞外領域に位置するよう「フリップした」SNAREを用いることで実証された。このようなフリップしたv-SNAREを持つ細胞とt-SNAREを持つ細胞が接触すると、トランスSNARE複合体が形成され、続いて細胞融合が起こった[7]。
構成要素
[編集]コアSNARE複合体は4-ヘリックスバンドルである[8]。シンタキシンとシナプトブレビンはαヘリックスを1本ずつ提供するが、SNAP-25からは2本のαヘリックスが加わる(Sn1、Sn2と略される)。SNARE複合体をジッパリング(zippering)するアミノ酸残基は層ごとにグループ分けされる。各層は、4本のαヘリックスそれぞれから1残基ずつの4つのアミノ酸残基で構成される。複合体の中央('0'層)は1つのアルギニン(R)と3つのグルタミン(Q)残基から構成されるzero ionic layerで、ロイシンジッパーが隣接している。複合体中央部の'-1'、'+1'、'+2'層は、ほぼ理想的なロイシンジッパー構造とアミノ酸構成をしている[9]。
zero ionic layerはVAMP-2のアルギニン56番残基、シンタキシン1Aのグルタミン226番残基、Sn1のグルタミン53番残基、Sn2のグルタミン174番残基から構成され、ロイシンジッパー層の内部に完全に埋め込まれている。アルギニン残基の正に帯電したグアニジノ基が、3つのグルタミン残基のカルボキシル基と相互作用する。
隣接するロイシンジッパー層は、イオン結合を周囲の溶媒から保護する耐水性シールとして機能している。zero ionic layerが水溶媒に曝露されると、隣接するロイシンジッパーが崩壊しSNARE複合体は不安定になる。これはシナプス小胞のエキソサイトーシスの完了後、α-SNAPとNSFがSNARE複合体をリサイクルする機構であると考えられている。
膜融合の機構
[編集]組み立て
[編集]
SNAREタンパク質が小胞融合に必要な力を提供するためには、トランスSNARE複合体へと組み立てられなければならない。4本のαヘリックス(シナプトブレビンとシンタキシンから1本ずつ、SNAP-25から2本)は集合してコイルドコイルモチーフを形成する。組み立て過程における律速段階はシンタキシンのSNAREドメインの結合である。このドメインは通常、他のSNAREタンパク質と相互作用できない「閉じた」状態となっている[10]。シンタキシンが「開いた」状態となると、各タンパク質のN末端のSNAREドメインが結合してトランスSNARE複合体の形成が開始される。SNAREドメインはC末端へ向かってコイルドコイルモチーフを形成していく。
SMタンパク質Munc18はSNARE複合体の集合に関与していると考えられているが、その正確な機構は未だ議論がある。Munc18はシンタキシンのSNAREドメインに結合して留め金のようにシンタキシンを閉じたコンフォメーションへ固定し、シンタキシンがSNARE複合体に参加するのを阻害する[10]。しかし、Munc18はトランスSNARE複合体の4-ヘリックスバンドルにも結合することができる。1つの仮説では、SNARE複合体の組み立ての間、Munc18の留め金はシンタキシンを閉じたコンフォメーションから解放する一方で、シンタキシンのN末端のペプチドへ結合したままであり(これによってシンタキシンのSNAREドメインが他のSNAREタンパク質と結合できるようになる)、その後新たに形成された4-ヘリックスSNARE複合体へと再結合するとされる[11]。この解離-再結合機構はカルシウム依存的である可能性がある[12]。この仮説はMunc18が小胞融合において重要な調節機能を果たすという考えを支持するもので、通常の状況ではSNARE複合体の形成はMunc18によって妨げられているが、いったん形成が開始されるとMunc18は融合の触媒としてSNARE複合体の組み立てを補助する[11]。
ジッパリングと融合孔開口
[編集]
膜の融合は、タンパク質の膜中の移動、脂質二重層の破壊、高度に湾曲した膜構造の再形成、というエネルギーを要する一連の過程からなる。2つの膜を近接させる過程は、膜間の静電的な反発力をの乗り越えるためにエネルギーの入力を必要とする。融合に先立って膜接触領域からの膜結合タンパク質の移動を調節する機構は不明であるが、膜の曲率の局所的な増大がこの過程に寄与していると考えられている。SNAREはタンパク質-脂質、タンパク質-タンパク質相互作用からエネルギーを産出し、それらが膜融合の駆動力となる。
1つのモデルでは、融合の間2つの膜を近接させておくために必要な力は、トランスSNARE複合体からシスSNARE複合体が形成される際のコンフォメーション変化からもたらされるとされる。現在の仮説では、この過程はSNAREジッパリング(zippering)と呼ばれている[13]。
トランスSNARE複合体が形成されたとき、SNAREタンパク質は未だ異なる膜上に存在する。SNAREタンパク質が自発的にコイル形成を続けるにつれて、より緊密で安定した4-ヘリックスバンドルが形成される。このSNARE複合体のジッパリングの過程で放出されるエネルギーの一部は、個々のSNAREモチーフが屈曲するストレスとして貯蔵されると考えられている。この機械的ストレスは、膜貫通ドメインとSNAREヘリックスバンドルの間の準剛体的な(フレキシブルではない)リンカー領域へ貯蔵されると想定されている[14][15]。このエネルギー的に不利な屈曲は、複合体が膜融合部位の周縁部へ移動したときに最小化となる。結果として、ストレスの解放が小胞-細胞膜間の反発力を上回り、2つの膜は互いに押し付けられることとなる[16]。
その後の段階のストークと融合孔の形成を説明するモデルもいくつか提唱されている。しかし、これらの過程の正確な性質については未だ議論がある。「ジッパー」仮説によると、SNARE複合体が形成されるにつれて、緊密化するヘリックスバンドルがシナプトブレビンとシンタキシンの膜貫通ドメインにねじり力を与える[17]。これによって膜貫通ドメインは各々の膜内で傾き、タンパク質のコイルはより緊密に巻き付くようになる。この膜貫通ドメインの不安定な配置は最終的に2つの膜の融合を引き起こし、SNAREタンパク質が同じ膜上に存在するシスSNARE複合体となる[18]。脂質の再配置の結果として、融合孔が開口し小胞の内容物が外部環境へ漏出する。
ストーク形成過程の連続的過程としての説明は、膜融合は最小限の半径から開始し、ストーク状の構造となるまで融合部の半径が放射状に広がっていくことを暗示する。しかし、このような説明は膜脂質の分子動力学を考慮に入れていないものである。近年の分子シミュレーションでは、膜が非常に近接すると、脂質の一部は疎水的な尾部を隣接する膜へ挿入し、いわば両脚を双方の膜へ伸ばした状態となることが示されている。この脚が広がった脂質状態を解消するために自発的にストーク構造が形成される。この分子的観点からは、ストークの形成ではなく、脚が広がった脂質を形成した中間状態が律速となるエネルギー障壁である。この広がった脂質コンフォメーションの形成のためのエネルギー障壁は、膜間の距離に比例する。SNARE複合体は2つの膜を互いに押し付け、この障壁を乗り越えるのに必要な自由エネルギーを提供する[19]。
解体
[編集]SNAREを介した膜融合が起こるために必要なエネルギーは、SNARE複合体の解体過程に由来するものである。想定されているエネルギー源は、膜融合に関与するATPアーゼNSFである。NSFのホモ六量体は、NSFの補助因子αSNAPとともに、ATPの加水分解と共役する形でSNARE複合体に結合して解体する[20]。この過程によってシナプトブレビンは再び小胞へ取り込まれるが、他のタンパク質は細胞膜へ結合したままである。
解離したSNAREタンパク質は、より安定なシスSNARE複合体よりも高エネルギー状態である。融合を駆動するエネルギーは、低エネルギー状態であるシスSNARE複合体への移行に由来するものであると考えられている。ATP加水分解と共役したSNARE複合体の解体は、「銃の撃鉄を起こす」のに似たエネルギー投資であり、いったん小胞融合の引き金が引かれれば、一連の過程は自発的に最適の速度で進行する。類似した過程は筋肉でも起こり、ミオシンの頭部はアクチンとの相互作用に必要なコンフォメーションを取るために最初にATPを加水分解する必要があり、その後パワーストロークが起こる。
エキソサイトーシスの調節
[編集]SNAP-25のパルミトイル化を介した調節
[編集]Q-SNAREタンパク質であるSNAP-25(synaptosomal-associated protein 25)は、ランダムコイルのリンカーで連結された2つのαヘリカルドメインから構成される。ランダムコイルリンカー領域は、4つのシステイン残基を持つことが知られている[21]。αヘリカルドメインはシンタキシンやシナプトブレビン(vesicle associated membrane protein、VAMPとしても知られる)とともに、4本のαヘリックスによるコイルドコイルからなるSNARE複合体を形成する。
シンタキシンとシナプトブレビンがそれぞれ標的部位と小胞の膜への結合を可能にする膜貫通ドメインを持っているのに対し、SNAP-25の標的膜への結合はランダムコイル領域のシステイン残基のパルミトイル化に依存している。いくつかの研究では、SNAREを介したシンタキシンとの結合によってこのような膜結合機構の必要がなくなることが示唆されている。しかし、シンタキシンをノックダウンしても膜に結合したSNAP-25の減少は見られず、代わりとなる結合機構が存在していると考えられる[22]。脂肪鎖はSNAP-25の1つまたはそれ以上のシステイン残基とチオエステル結合を介して共有結合しており、この段階で膜への結合や最終的にはSNAREを介したエキソサイトーシスが調節されている。この過程はDHHCパルミトイルトランスフェラーゼ(DHHC palmitoyl transferase)と呼ばれる特別な酵素によって媒介されている[23]。また、このSNAP-25のシステインに富むドメインは細胞膜へ弱く結合することが示されており、後のパルミトイル化のために酵素の近くへ局在する可能性がある。この過程の逆の過程はパルミトイルタンパク質チオエステラーゼと呼ばれる別の酵素によって行われる(図を参照)。

SNAP-25のSNARE複合体への取り込みは、標的膜の脂質マイクロドメインへのSNAP-25の局在によって空間的に調節されている可能性がある。パルミトイル化されたシステイン残基は、結合した脂肪酸に相補的で好ましい脂質環境(おそらくコレステロールに富んだ環境)をもつ標的膜上の特定の領域へと局在する可能性がある[24]。
神経軸索終末の電位依存性カルシウムチャネルによるSNAP-25の調節
[編集]活動電位が軸索終末に到達すると、脱分極によって電位依存性カルシウムチャネル(voltage-gated calcium channel、VGCC)の開口が刺激され、電気化学勾配に従ってカルシウムが急激に流入する。カルシウムはシナプトタグミン1との結合を介してエキスサイトーシスを促進する。しかし、SNAP-25はグルタミン酸作動性神経細胞においてVGCCの機能を負に制御することが示されている。SNAP-25はVGCCによる電流密度を減少させ、そのためシナプトタグミンに結合するカルシウムの量は減少し、エキソサイトーシスは減少する。逆に、SNAP-25の発現低下はVGCCの電流密度を増加させエキソサイトーシスを増大させる[25]。
さらなる調査によって、SNAP-25の過剰発現/発現低下とさまざまな脳疾患が関係している可能性が示唆されている。注意欠陥・多動性障害(ADHD)ではSNAP-25の遺伝子座の多型が疾患と関連付けられており、その症状における役割が示唆されている[26]。このことは、coloboma変異体マウスを用いて行われたSNAP-25のヘテロノックアウトによる研究でADHDに特徴的な表現型がみられたことからもさらに示唆される[27]。研究からは、SNAP-25の過剰発現/発現低下と統合失調症の発症との相関も示唆されている[28][29]。
シンタキシンとHabcドメイン
[編集]シンタキシンは、膜貫通ドメイン、αヘリックスからなるSNAREドメイン、短いリンカー領域、そして3つのαヘリカル領域からなるHabcドメインから構成される。シンタキシンのSNAREドメインは、SNARE複合体に必要な4-ヘリックスバンドルを形成しその後の膜融合を行うために、SNAP-25とシナプトブレビンが結合する標的部位である。しかし、Habcドメインはシンタキシンの自己阻害ドメインとして機能する。Habcドメインは折り返されてSNAREドメインと結合して「閉じた」状態を誘導し、SNAREの形成の物理的な障壁となる。逆に、HabcドメインがSNAREドメインから解離するとシンタキシンは自由にSNAP-25やシナプトブレビンと結合する[30]。
シンタキシン1Bと小胞の即時放出可能プール
[編集]シンタキシンのサブタイプには非常に大きな多様性があり、ヒトゲノムには15種類のサブタイプが存在する[31]。シンタキシン1Bは軸索終末において、小胞の即時放出可能プール(readily releasable pool、RRP)と呼ばれる、エキソサイトーシスに備えたシナプス小胞の数を調節していることが示唆されている。2014年に行われたノックアウトによる研究では、シンタキシン1Bが欠失するとRRPのサイズが大きく減少することが示された[32]。
毒素
[編集]多くの神経毒はSNARE複合体に直接的な影響を与える。このような毒素としてはボツリヌストキシンやテタヌストキシンがあり、SNAREの構成要素を標的とする。これらの毒素は小胞の適切なリサイクリングを防ぎ、結果として筋肉の制御不能、痙攣、麻痺が生じ、死に至ることさえもある。
ボツリヌス神経毒素
[編集]ボツリヌストキシンまたはボツリヌス神経毒素(botulinum neurotoxin、BoNT)は、これまでに発見されている毒素の中で最も毒性の高いものの1つである[33]。BoNTは、神経細胞でSNAREタンパク質を切断するタンパク質分解酵素である。そのタンパク質構造は、重鎖(100 kDa)と軽鎖(50 kDa)の2つのサブユニットから構成され、両者はジスルフィド結合によって連結されている。BoNTの作用機序は、神経細胞膜への結合、エンドサイトーシス、膜移行、そしてSNAREタンパク質の分解、という4段階からなる機構である[34]。
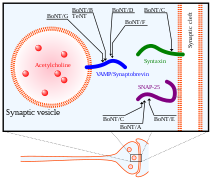
まず標的となる神経細胞を見つけるために、BoNTの重鎖が利用される。重鎖はシナプス前細胞のガングリオシドや膜タンパク質に結合する。続いて、毒素が細胞の膜の中へエンドサイトーシスされる。重鎖にはコンフォメーション変化が起こり、これは軽鎖が神経細胞の細胞質へ移行するために重要である。最後に、軽鎖が標的神経細胞の細胞質へ移動した後、軽鎖は重鎖から解離し、SNAREタンパク質の切断部位に到達できるようになる[34]。両鎖を結び付けているジスルフィド結合が還元されることで、軽鎖は重鎖から解離する。このジスルフィド結合の還元は、NADPH-チオレドキシンレダクターゼ-チオレドキシンシステムによって媒介される[36]。BoNTの軽鎖は、亜鉛(II)イオン依存的メタロプロテアーゼとしてSNAREタンパク質に作用し[37]、SNAREを切断してエキソサイトーシス機能を喪失させる。
BoNTには8つの既知のアイソタイプ(BoNT/AからBoNT/H)が存在し、それぞれSNAREタンパク質の特異的切断部位が異なる。シナプス膜に位置するSNAREタンパク質ファミリーのメンバーSNAP25は、アイソタイプA、C、Eによって切断される。これらのアイソタイプによるSNAP25の切断は、小胞のシナプス膜への融合のためのSNARE複合体形成を大きく阻害する。BoNT/Cは、シナプス膜に位置する別のタンパク質であるシンタキシン1をも標的とする。シンタキシンの切断もSNAP25と同様の結果となる。3つ目のSNAREタンパク質シナプトブレビン(VAMP)は小胞に位置する。VAMP2はアイソタイプB、D、Fの標的であり切断される[33]。BoNTのさまざまなアイソタイプとテタヌストキシン(TeNT)の標的が右の図で示されている。
BoNTはSNAREタンパク質の機能的損傷を引き起こすため、重大な生理的・医学的意義を有している。SNAREタンパク質を損傷させることで、毒素はシナプス小胞がシナプス膜へ融合して神経伝達物質をシナプス間隙へ放出するのを防ぐ。シナプス間隙への神経伝達物質の放出が阻害されることで、筋細胞の刺激のための活動電位が伝達されなくなる。その結果、感染者には麻痺が引き起こされ、深刻な場合には死が引き起こされることもある。BoNTの効果は致死的ともなりうるが、医療や美容目的の治療薬としても利用されている[38][39]。
テタヌス神経毒素
[編集]
テタヌストキシンまたはテタヌス神経毒素(Tetanus neurotoxin、TeNT)は、ジスルフィド結合で連結された重鎖(100 kDa)と軽鎖(50 kDa)から構成される。重鎖はTeNTの神経終末の膜への特異的結合、毒素のエンドサイトーシス、そして軽鎖の細胞質への移行を担う。軽鎖は亜鉛依存的エンドペプチダーゼ活性、より詳しくはマトリックスメタロプロテアーゼ活性を有し、シナプトブレビンやVAMPの切断を行う[40]。
TeNTの軽鎖が活性化するには、毒素1分子につき1つの亜鉛原子が結合しなければならない[41]。亜鉛結合時に、主にNADPH-チオレドキシンレダクターゼ-チオレドキシン酸化還元システムを介してジスルフィド結合が還元される[42]。その後、軽鎖はシナプトブレビンのGln76-Phe77間の結合を切断する[40]。シナプトブレビンの切断は、NSFの結合の標的となる低エネルギーコンフォメーションへの移行を制限し、SNAREのコアの安定性に影響を与える[43]。このシナプトブレビンの切断はTeNTの最終的な標的であり、毒素は低容量であっても神経伝達物質のエキソサイトーシスを阻害する。
神経伝達物質の放出における役割
[編集]神経伝達物質は、シナプス前終末内に限定された小胞の即時放出可能プールに貯蔵されている。神経分泌・エキソサイトーシスの過程を通じて、SNAREは小胞のドッキング、プライミング、融合、シナプス間隙への神経伝達物質の放出の同期に重要な役割を果たす。
シナプス小胞の融合の最初の段階はテザリング(tethering)であり、小胞は貯蔵プールから移動し膜と物理的に接触する。膜側では、Munc18がまずシンタキシン1Aの閉じた構造に結合する。複合体からMunc18が解離することで、シンタキシン1Aはv-SNAREタンパク質と結合できるようになると想定されている[44]。次の段階は小胞のドッキング(docking)であり、v-SNAREとt-SNAREタンパク質はカルシウム非依存的に一過的に結合する。その後はプライミング(priming)であり、SNAREモチーフが小胞と膜の間で安定な相互作用を形成する。コンプレキシンはプライミングされたSNARE複合体を安定化し、小胞を迅速なエンドサイトーシスが準備された状態とする。
プライミングされた小胞とSNAREタンパク質を高濃度で含むシナプス前膜の範囲は、アクティブゾーンと呼ばれる。電位依存性カルシウムチャネルはアクティブゾーンの周辺に高度に濃縮しており、シナプスの膜の脱分極に応答してチャネルが開く。カルシウムの流入はシナプトタグミン1によって検知され、シナプトタグミンはコンプレキシンを取り除き、神経伝達物質の放出のために小胞がシナプス前膜と融合できるようにする。また、電位依存性カルシウムチャネルは、t-SNAREであるシンタキシン1AとSNAP25、そしてシナプトタグミン1とも直接相互作用することが示されている。これらの相互作用はカルシウムチャネル活性を阻害するとともに、放出部位の周辺に分子を緊密に凝集させる[45]。
SNAREの遺伝子と神経疾患との関連は多くの臨床例で報告されている。SNAP25のmRNAの欠乏が一部の統合失調症患者の海馬組織で観察されており、SNAP25の一塩基多型は自閉症スペクトラムと、SNAP25Bの過剰発現は双極性障害の早期の発症と関連している[45]。
オートファジーにおける役割
[編集]マクロオートファジーはオートファゴソームと呼ばれる二重膜結合性の細胞小器官の形成を伴う異化過程であり、リソソームとの融合によって細胞内の構成要素を分解する。オートファジーの間、細胞質の一部は隔離膜(phagophore)と呼ばれるお椀型の二重膜構造に内包され、最終的には完全に組み立てられたオートファゴソームの内容物となる。オートファゴソームの生合成は、隔離膜の形成開始と成長とを必要とする。かつてはこの過程は脂質が新規に付加されて起こると考えられていたが、近年得られた証拠は隔離膜の成長に寄与する脂質が小胞体、ゴルジ体、細胞膜、ミトコンドリアを含むさまざまな膜要素に由来することを示唆している[46]。SNAREは隔離膜の形成開始と拡大、そしてオートファジーの後期の段階であるオートファゴソームとリソソームの融合の媒介に重要な役割を果たす。
哺乳類の隔離膜の形成開始機構は不明であるが、SNAREはAtg16L、v-SNAREタンパク質VAMP7、そしてそのパートナーであるシンタキシン7、シンタキシン8、VTI1Bなどのt-SNAREを含む、小型のクラスリン被覆小胞どうしの同型融合による隔離膜の形成への関与が示唆されている[47]。酵母では、t-SNAREであるSec9pとSso2pがエキソサイトーシスには必要であり、オートファゴソームの生合成に必要なAtg9陽性小胞の細管小胞性(tubulovesiclar)の出芽を促進する[48][49]。これらのSNAREのいずれかをノックアウトすると、融合しない小さなAtg9陽性小胞が蓄積し、オートファゴソーム前駆構造体の形成が妨げられる[50]。
隔離膜の組み立てに加えて、SNAREはオートファゴソームとリソソームの融合の媒介にも重要である、哺乳類では、VAMP7、VAMP8、VTI1Bがオートファゴソームとリソソームの融合には必要である。リソソーム蓄積症ではこの過程に異常がみられ、コレステロールがリソソームに蓄積し、SNAREは膜のコレステロールに富む領域に隔離されてリサイクリングが阻害される。近年、VAMP8とSNAP29と相互作用し、リソソームとの融合に必要なオートファゴソーム関連SNAREとしてシンタキシン17が同定された[51]。シンタキシン17はオートファゴソームの外膜に局在しているが、隔離膜や他のオートファゴソーム前駆体には見られず、これらの未成熟な構造体とリソソームとの融合が防がれている[51]。酵母では、オートファゴソームと液胞(酵母ではリソソームに相当する)の融合には、シンタキシンのホモログVam3、SNAP25のホモログVam7、Ras様GTPアーゼYpt7、そしてNSFのオルソログSec18といった、SNAREとその関連タンパク質が必要とされる[46]。
出典
[編集]- ^ Georgiev, Danko D; James F . Glazebrook (2007). “Subneuronal processing of information by solitary waves and stochastic processes”. In Lyshevski, Sergey Edward. Nano and Molecular Electronics Handbook. Nano and Microengineering Series. CRC Press. pp. 17–1–17–41. doi:10.1201/9781420008142.ch17. ISBN 978-0-8493-8528-5
- ^ Burri, Lena; Lithgow, Trevor (2004-01-01). “A complete set of SNAREs in yeast”. Traffic (Copenhagen, Denmark) 5 (1): 45–52. doi:10.1046/j.1600-0854.2003.00151.x. ISSN 1398-9219. PMID 14675424.
- ^ Cell and Molecular Biology (4th ed.). John Wiley & Sons. (2002)
- ^ “Organization of SNAREs within the Golgi stack.”. Cold Spring Harbor Perspectives in Biology 3 (10): a005249. (1 October 2011). doi:10.1101/cshperspect.a005249. PMC 3179334. PMID 21768609.
- ^ “Tethering the assembly of SNARE complexes.”. Trends in Cell Biology 24 (1): 35–43. (January 2014). doi:10.1016/j.tcb.2013.09.006. PMID 24119662.
- ^ “Mechanisms of membrane fusion: disparate players and common principles”. Nature Reviews Molecular Cell Biology 9 (7): 543–556. (21 May 2008). doi:10.1038/nrm2417. PMID 18496517.
- ^ “Fusion of Cells by Flipped SNAREs”. Science 300 (5626): 1745–1749. (13 June 2003). doi:10.1126/science.1084909. PMID 12805548.
- ^ “Crystal structure of a SNARE complex involved in synaptic exocytosis at 2.4 Å resolution”. Nature 395 (6700): 347–353. (1998). doi:10.1038/26412. PMID 9759724.
- ^ “Conserved structural features of the synaptic fusion complex: SNARE proteins reclassified as Q- and R-SNAREs”. Proceedings of the National Academy of Sciences 95 (26): 15781–15786. (1998). doi:10.1073/pnas.95.26.15781. PMC 28121. PMID 9861047.
- ^ a b “Munc18a controls SNARE assembly through its interaction with the syntaxin N-peptide”. EMBO J. 27 (7): 923–33. (2008). doi:10.1038/emboj.2008.37. PMC 2323264. PMID 18337752.
- ^ a b “Membrane fusion: grappling with SNARE and SM proteins”. Science 323 (5913): 474–7. (January 2009). doi:10.1126/science.1161748. PMC 3736821. PMID 19164740.
- ^ “Molecular machines governing exocytosis of synaptic vesicles”. Nature 490 (7419): 201–7. (2012). doi:10.1038/nature11320. PMC 4461657. PMID 23060190.
- ^ “SNARE-mediated membrane fusion”. Nat. Rev. Mol. Cell Biol. 2 (2): 98–106. (2001). doi:10.1038/35052017. PMID 11252968.
- ^ “Functional analysis of conserved structural elements in yeast syntaxin Vam3p”. J. Biol. Chem. 276 (30): 28598–605. (2001). doi:10.1074/jbc.M101644200. PMID 11349128.
- ^ “Measuring distances in supported bilayers by fluorescence interference-contrast microscopy: polymer supports and SNARE proteins.”. Biophysical Journal 84 (1): 408–18. (January 2003). doi:10.1016/s0006-3495(03)74861-9. PMC 1302622. PMID 12524294.
- ^ “Caught in the act: visualization of SNARE-mediated fusion events in molecular detail.”. ChemBioChem: A European Journal of Chemical Biology 12 (7): 1049–55. (2 May 2011). doi:10.1002/cbic.201100020. hdl:11858/00-001M-0000-0027-C8EA-9. PMID 21433241.
- ^ “How could SNARE proteins open a fusion pore?”. Physiology 29 (4): 278–85. (2014). doi:10.1152/physiol.00026.2013. PMC 4103061. PMID 24985331.
- ^ Zucker, Robert S.; Kullmann, Dimitri M.; Kaeser, Pascal S. (August 2014). “Chapter 15: Release of Neurotransmitters”. In Byrne, John H.; Heidelberger, Ruth; Waxham, M. Neal. From Molecules to Networks: An Introduction to Cellular and Molecular Neuroscience. Academic Press. pp. 443–488. ISBN 9780123982674
- ^ “How SNARE molecules mediate membrane fusion: recent insights from molecular simulations.”. Current Opinion in Structural Biology 22 (2): 187–96. (April 2012). doi:10.1016/j.sbi.2012.01.007. hdl:11858/00-001M-0000-000F-9AF7-9. PMID 22365575.
- ^ “A protein assembly-disassembly pathway in vitro that may correspond to sequential steps of synaptic vesicle docking, activation, and fusion”. Cell 75 (3): 409–18. (1993). doi:10.1016/0092-8674(93)90376-2. PMID 8221884.
- ^ Bock, LV; Woodbury, DJ (9 August 2010). “Chemomechanical regulation of SNARE proteins studied with molecular dynamics simulations”. Biophysical Journal 99 (4): 1221–1230. doi:10.1016/j.bpj.2010.06.019. PMC 2920728. PMID 20713006.
- ^ Greaves, Jennifer (5 April 2009). “Regulation of SNAP-25 Trafficking and Function by Palmitoylation”. Biochemical Society Transactions 38 (part 1): 163–166. doi:10.1042/BST0380163. PMID 20074052.
- ^ Greaves, Jennifer (11 May 2010). “Palmitoylation of the SNAP-25 Protein Family: Specificity and Regulation by DHHC Palmitoyl Transferases”. The Journal of Biological Chemistry 285 (32): 24629–24638. doi:10.1074/jbc.M110.119289. PMC 2915699. PMID 20519516.
- ^ Greaves, Jennifer (5 April 2009). “Regulation of SNAP-25 Trafficking and Function by Palmitoylation”. Biochemical Society Transactions 38 (part 1): 163–166. doi:10.1042/bst0380163. PMID 20074052.
- ^ Condliffe, Steven B (3 June 2010). “Endogenous SNAP-25 Regulates Native Voltage-gated Calcium Channels in Glutamatergic Neurons”. The Journal of Biological Chemistry 285 (32): 24968–24976. doi:10.1074/jbc.M110.145813. PMC 2915732. PMID 20522554.
- ^ Corradini, Irene (21 January 2009). “SNAP-25 in Neuropsychiatric Disorders”. Annals of the New York Academy of Sciences 1152: 93–99. doi:10.1111/j.1749-6632.2008.03995.x. PMC 2706123. PMID 19161380.
- ^ Hess, EJ (1992). “Spontaneous locomotor hyperactivity in a mouse mutant with a deletion including the Snap gene on chromosome 2”. Journal of Neuroscience 12 (7): 2865–2874. doi:10.1523/JNEUROSCI.12-07-02865.1992. PMID 1613559.
- ^ Thompson, PM (1998). “Altered levels of the synaptosomal associated protein SNAP-25 in schizophrenia”. Biological Psychiatry 43 (4): 239–243. doi:10.1016/s0006-3223(97)00204-7. PMID 9513732.
- ^ Gabriel, SM (1997). “Increased concentrations of presynaptic proteins in the cingulate cortex of subjects with schizophrenia”. Archives of General Psychiatry 54 (6): 559–566. doi:10.1001/archpsyc.1997.01830180077010. PMID 9193197.
- ^ MacDonald, Chris (3 April 2009). “Autoinhibition of SNARE complex assembly by a conformational switch represents a conserved feature of syntaxins”. Biochemical Society Transactions 38 (Pt 1): 209–212. doi:10.1042/BST0380209. PMC 5242387. PMID 20074061.
- ^ Teng, Felicia Yu Hsuan (24 October 2001). “The Syntaxin”. Genome Biology 2 (11): reviews 3012.1–7. doi:10.1186/gb-2001-2-11-reviews3012. PMC 138984. PMID 11737951.
- ^ Mishima, Tatsuya (28 February 2014). “Syntaxin 1B, but Not Syntaxin 1A, Is Necessary for the Regulation of Synaptic Vesicle Exocytosis and of the Readily Releasable Pool at Central Synapses”. PLoS ONE 9 (2): e90004. doi:10.1371/journal.pone.0090004. PMC 3938564. PMID 24587181.
- ^ a b “Cytotoxicity of botulinum neurotoxins reveals a direct role of syntaxin 1 and SNAP-25 in neuron survival”. Nature Communications 4: 1472. (12 February 2013). doi:10.1038/ncomms2462. PMC 4052923. PMID 23403573.
- ^ a b “An update on the mechanism of action of tetanus and botulinum neurotoxins”. Acta Chim Slov 58 (4): 702–7. (December 2011). PMID 24061118.
- ^ “Botulinum neurotoxin detection and differentiation by mass spectrometry”. Emerging Infect. Dis. 11 (10): 1578–83. (2005). doi:10.3201/eid1110.041279. PMC 3366733. PMID 16318699.
- ^ Pirazzini, Marco; Bordin, Fulvio; Rossetto, Ornella; Shone, Clifford C.; Binz, Thomas; Montecucco, Cesare (2013-01-16). “The thioredoxin reductase-thioredoxin system is involved in the entry of tetanus and botulinum neurotoxins in the cytosol of nerve terminals”. FEBS letters 587 (2): 150–155. doi:10.1016/j.febslet.2012.11.007. ISSN 1873-3468. PMID 23178719.
- ^ “Catalytic Features of the Botulinum Neurotoxin A light chain Revealed by High Resolution Structure of an Inhibitory Peptide Complex”. Biochemistry 47 (21): 5736–5745. (May 2008). doi:10.1021/bi8001067. PMID 18457419.
- ^ “Botulinum toxin A, adjunctive therapy for refractory headaches associated with pericranial muscle tension”. Headache 38 (6): 468–71. (1998). doi:10.1046/j.1526-4610.1998.3806468.x. PMID 9664753.
- ^ “Cosmetic denervation of the muscles of facial expression with botulinum toxin. A dose-response study”. Dermatol Surg 22 (1): 39–43. (1996). doi:10.1111/j.1524-4725.1996.tb00569.x. PMID 8556256.
- ^ a b “Tetanus and botulinum-B neurotoxins block neurotransmitter release by proteolytic cleavage of synaptobrevin.”. Nature 359 (6398): 832–5. (29 October 1992). doi:10.1038/359832a0. PMID 1331807.
- ^ “Tetanus toxin is a zinc protein and its inhibition of neurotransmitter release and protease activity depend on zinc.”. The EMBO Journal 11 (10): 3577–83. (October 1992). doi:10.1002/j.1460-2075.1992.tb05441.x. PMC 556816. PMID 1396558.
- ^ “The thioredoxin reductase-thioredoxin system is involved in the entry of tetanus and botulinum neurotoxins in the cytosol of nerve terminals”. FEBS Lett. 587 (2): 150–5. (2013). doi:10.1016/j.febslet.2012.11.007. PMID 23178719.
- ^ “Clostridial neurotoxins compromise the stability of a low energy SNARE complex mediating NSF activation of synaptic vesicle fusion.”. The EMBO Journal 14 (19): 4705–13. (2 October 1995). doi:10.1002/j.1460-2075.1995.tb00152.x. PMC 394567. PMID 7588600.
- ^ “Dual roles of Munc18-1 rely on distinct binding modes of the central cavity with Stx1A and SNARE complex.”. Molecular Biology of the Cell 22 (21): 4150–60. (November 2011). doi:10.1091/mbc.e11-02-0150. PMC 3204075. PMID 21900493.
- ^ a b “The SNARE complex in neuronal and sensory cells.”. Molecular and Cellular Neurosciences 50 (1): 58–69. (May 2012). doi:10.1016/j.mcn.2012.03.009. PMC 3570063. PMID 22498053.
- ^ a b “Autophagosome Precursor Maturation Requires Homotypic Fusion”. Cell 146 (2): 303–317. (July 2011). doi:10.1016/j.cell.2011.06.023. PMC 3171170. PMID 21784250.
- ^ “Plasma membrane contributes to the formation of pre-autophagosomal structures”. Nature Cell Biology 12 (8): 747–757. (18 July 2010). doi:10.1038/ncb2078. PMC 2923063. PMID 20639872.
- ^ Abeliovich, H.; Darsow, T.; Emr, S. D. (1999-11-01). “Cytoplasm to vacuole trafficking of aminopeptidase I requires a t-SNARE-Sec1p complex composed of Tlg2p and Vps45p”. The EMBO journal 18 (21): 6005–6016. doi:10.1093/emboj/18.21.6005. ISSN 0261-4189. PMC 1171666. PMID 10545112.
- ^ Nair, Usha; Jotwani, Anjali; Geng, Jiefei; Gammoh, Noor; Richerson, Diana; Yen, Wei-Lien; Griffith, Janice; Nag, Shanta et al. (2011-07-22). “SNARE proteins are required for macroautophagy”. Cell 146 (2): 290–302. doi:10.1016/j.cell.2011.06.022. ISSN 1097-4172. PMC 3143362. PMID 21784249.
- ^ “SNARE Proteins Are Required for Macroautophagy”. Cell 146 (2): 290–302. (July 2011). doi:10.1016/j.cell.2011.06.022. PMC 3143362. PMID 21784249.
- ^ a b “The Hairpin-type Tail-Anchored SNARE Syntaxin 17 Targets to Autophagosomes for Fusion with Endosomes/Lysosomes”. Cell 151 (6): 1256–1269. (December 2012). doi:10.1016/j.cell.2012.11.001. PMID 23217709.
外部リンク
[編集]- SNARE Proteins - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス
- SNARE Complex - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス
