リボンダイアグラム

リボンダイアグラム(英: Ribbon diagram)は、リチャードソンダイアグラムとも呼ばれ、タンパク質の構造を三次元的に模式的に表現した、現在使用されている最も一般的なタンパク質の描写方法の1つである。リボンはタンパク質骨格の全体的な経路と構成を三次元で示していて、右上の画像にあるミオグロビンの活性部位に結合している酸素原子の球のように、完全な原子構造の詳細を取り付けるための視覚的な枠組みとしての役割を果たしている。リボンダイアグラムは、ポリペプチド骨格を貫く滑らかな曲線を補間して作成したもので、α-ヘリックスはコイル状のリボンまたは太いチューブで、β-ストランドは矢印で、非反復コイルまたはループは線または細いチューブで示している。ポリペプチド鎖の方向は、矢印によって局所的に示され、全体的にはリボンの長手方向に沿った色変化で示されることもある[1]。
リボンダイアグラムは、シンプルでありながら強力で、分子構造の視覚的な基部(ねじれ、折りたたみ、展開)を表現している。この方法は、タンパク質構造の全体的な構成をうまく描写することに成功し、その三次元的な性質を反映して、構造生物学の専門家だけでなく、他の科学者や学生、一般の人々も[2]、この複雑な物体の理解を深めることができた。
歴史
[編集]
1980年にジェーン S. リチャードソンによって手書きされた最初のリボンダイアグラム(それ以前の個人のイラストに影響された)は[3]、タンパク質の三次元構造を体系的に作成した最初の概略図であった[3][4]。これらは、Advances in Protein Chemistry誌に掲載された論文のために[5]、タンパク質構造の分類を説明するために作成された(現在、Anataxからオンラインで注釈付きの形で入手可能)。これらの図面は、原子座標のCαトレースを印刷したトレーシングペーパーの上にペンで輪郭を描き、色鉛筆やパステルで陰影をつけたもので[6]、位置を保持し、主鎖経路を滑らかにし、視覚的な外観を明確にするために小さな局所的偏位を取り入れている[4]。右のトリオースイソメラーゼのリボンダイアグラムの他にも、プレアルブミン、フラボドキシン、Cu,Znスーパーオキシドディスムターゼなどが手書きで描かれている。
1982年、Arthur M. Leskと共同研究者は、Protein Data Bankファイルを入力として使用する計算機的な実装により、リボンダイアグラムの自動生成を初めて可能にした[7]。この概念的にシンプルなアルゴリズムは、3次多項式Bスプライン曲線をペプチド平面にフィットさせる。最近のグラフィックシステムのほとんどは、基本的な描画プリミティブとして、Bスプラインまたはエルミートスプラインが用意されている。ある種類のスプライン実装では、各Cαガイドポイントを通過させることで、正確ではあるが途切れた曲線を生成する。手書きのリボンも、ほとんどのコンピュータのリボンも(ここに示すものなど)、約4つの連続したガイドポイント(通常はペプチドの中間点)の上で平滑化され、より視覚的に美しく、理解しやすい表現を作り出す。滑らかなβ-ストランドを維持しながら、らせん状のスパイラルに適切な半径を与えるために、スプラインは局所的な曲率に比例したオフセットで修正することができる。この方法は、Mike CarsonがRibbonsプログラム[8]で最初に開発したもので、その後、右上のリボン画像を作成したキネマージュグラフィックス[9]用のオープンソースのMageプログラムなど、他の分子グラフィックスソフトウェアでも採用された。(例: 1XK8トリマー、DNAポリメラーゼ)
リボンダイアグラムは、その登場から現在にいたるまで、タンパク質の構造を表す最も一般的な図であり、ジャーナルや教科書の表紙に使われる一般的な選択肢となっている。
現在のコンピュータプログラム
[編集]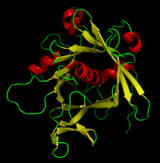
リボンダイアグラムの描画に使用される人気のあるプログラムの1つにMolscriptがある。Molscriptは、エルミートスプラインを利用して、コイル、ターン、ストランド、およびヘリックスの座標を作成する。その曲線は、方向ベクトルによって導かれるすべての制御点(Cα原子)を通過する。このプログラムは、Arthur M. Lesk、Karl Hardman、John Priestleによって伝統的な分子グラフィックスをベースに構築された[10]。Jmolは、ウェブ上で分子構造を閲覧するためのオープンソースのJavaベースのビューアで、リボンを簡略化した「漫画」バージョンも含まれている。他にも、DeepView(例:ウレアーゼ)やMolMol(例:SH2ドメイン)などのグラフィックプログラムでもリボンダイアグラムを作成する。KiNGは[11]、Mageの後継となるJavaベースのソフトウェアである(例:α溶血素の上面図と側面図)。
UCSF Chimeraは、リボンなどの可視化も含む強力な分子モデリングプログラムで、特に低温電子顕微鏡データの輪郭形状と組み合わせる機能が特徴である[12]。Warren DeLanoによるPyMOLは[13]、人気の高い柔軟な分子グラフィックスプログラムで(Pythonベース)、対話的モードで動作し、リボンダイアグラムやその他の多くのプレゼンテーション品質の2D画像を作成する。
特徴
[編集]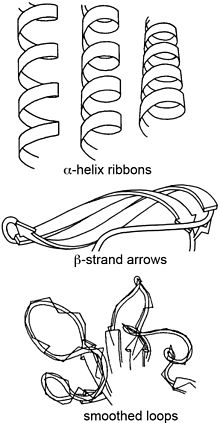
| 二次構造[14][15] | |
|---|---|
| α-ヘリックス | 円筒形のスパイラルリボンで、リボンの平面はペプチドの平面にほぼ沿っている。 |
| β-ストランド | 幅の約4分の1の厚みの矢印は、アミノ末端からカルボキシ末端までのストランドの方向とねじれを示している。隣り合うストランドが一体となってねじれているため、βシートは一体化して見える。 |
| ループとその他 | |
| 非反復ループ | 手前が太く、奥に向かって細くなっていく丸いロープは、Cαトレースの滑らかな経路に沿っている。 |
| ループとヘリックスの接合部 | 丸いロープが徐々に平らになり、細いらせん状のリボンになる。 |
| その他の機能 | |
| ポリペプチドの方向、NH2末端とCOOH末端 | 終端または文字の片方または両方に小さな矢印、または文字がある。β-ストランドの場合は、矢印の方向で十分である。今日、ポリペプチド鎖の方向は、色変化で示すことが多い。 |
| ジスルフィド結合 | 結合したSSシンボルや、様式化された稲妻のようなジグザグ。 |
| 補欠分子族または阻害剤 | 棒人形または球棒モデル。 |
| 金属 | 球。 |
| 陰影と色 | 陰影や色は、図に立体感を与える。一般的に、手前にあるものが最もコントラストが高く、奥にあるものが最も低い。 |
参照項目
[編集]脚注
[編集]
- ^ Smith, Thomas J. (October 27, 2005). “Displaying and Analyzing Atomic Structures on the Macintosh”. Danforth Plant Science Center. 28 March 2002時点のオリジナルよりアーカイブ。2021年4月16日閲覧。
- ^ Richardson, D. C.; Richardson, J. S. (January 2002). “Teaching Molecular 3-D Literacy”. Biochemistry and Molecular Biology Education 30 (1): 21–26. doi:10.1002/bmb.2002.494030010005.
- ^ a b Richardson, Jane S. (2000), “Early ribbon drawings of proteins”, Nature Structural Biology 7 (8): 624–625, doi:10.1038/77912, PMID 10932243.
- ^ a b Richardson, Jane S. (1985), “Schematic Drawings of Protein Structures”, Methods in Enzymology, Methods in Enzymology 115: 359–380, doi:10.1016/0076-6879(85)15026-3, ISBN 978-0-12-182015-2, PMID 3853075.
- ^ Richardson, Jane S. (1981), “Anatomy and Taxonomy of Protein Structures”, Advances in Protein Chemistry, Advances in Protein Chemistry 34: 167–339, doi:10.1016/S0065-3233(08)60520-3, ISBN 978-0-12-034234-1, PMID 7020376.
- ^ “Science’s ‘Mother of Ribbon Diagrams’ celebrates 50 years at Duke” (英語). Duke Stories (2018年10月19日). 2020年6月9日閲覧。
- ^ Lesk, Arthur M.; Hardman, Karl D. (1982), “Computer-Generated Schematic Diagrams of Protein Structures”, Science 216 (4545): 539–540, Bibcode: 1982Sci...216..539L, doi:10.1126/science.7071602, PMID 7071602.
- ^ Carson, M.; Bugg, C. E. (1986), “Algorithm for Ribbon Models of Proteins”, Journal of Molecular Graphics 4 (2): 121–122, doi:10.1016/0263-7855(86)80010-8.
- ^ Richardson, D. C.; Richardson, J. S. (January 1992), “The kinemage: a tool for scientific communication”, Protein Science 1 (1): 3–9, doi:10.1002/pro.5560010102, PMC 2142077, PMID 1304880
- ^ MolScript v2.1: About the program
- ^ Chen, V. B.; Davis, I. W.; Richardson, D. C. (2009), “KING (Kinemage, Next Generation): A versatile interactive molecular and scientific visualization program”, Protein Science 18 (11): 2403–2409, doi:10.1002/pro.250, PMC 2788294, PMID 19768809
- ^ Goddard, Thomas D.; Huang, Conrad C.; Ferrin, Thomas E. (2005), “Software Extensions to UCSF Chimera for Interactive Visualization of Large Molecular Assemblies”, Structure 13 (3): 473–482, doi:10.1016/j.str.2005.01.006, PMID 15766548.
- ^ Brunger, Axel T.; Wells, James A. (2009), “Warren L. DeLano, 21 June 1972-3 November 2009”, Nature Structural & Molecular Biology 16 (12): 1202–1203, doi:10.1038/nsmb1209-1202, PMID 19956203.
- ^ Richardson, Jane S. (1985), “Schematic Drawings of Protein Structures”, Methods in Enzymology, Methods in Enzymology 115: 359–380, doi:10.1016/0076-6879(85)15026-3, ISBN 978-0-12-182015-2, PMID 3853075.
- ^ Richardson, Jane S. (1981), “Anatomy and Taxonomy of Protein Structures”, Advances in Protein Chemistry, Advances in Protein Chemistry 34: 167–339, doi:10.1016/S0065-3233(08)60520-3, ISBN 978-0-12-034234-1, PMID 7020376.
