「ドッキング (分子)」の版間の差分
ページ「Docking (molecular)」の翻訳により作成 |
(相違点なし)
|
2020年7月23日 (木) 23:29時点における版
分子モデリングの分野では、ドッキング(docking)は、安定なタンパク質複合体を形成するために互いに結合したときに、ある分子の第2の分子に対する好ましい配向を予測する方法である[1]。好ましい配向の知識を使用すれば、例えばスコアリング関数 (分子)を使用して、2つの分子間の会合の強さや結合親和性を予測することができる。
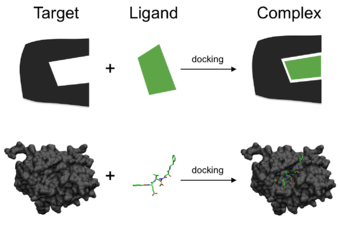
タンパク質、ペプチド、核酸、炭水化物、脂質などの生物学的に関連する分子間の関連付けは、シグナル伝達において中心的な役割を果たしている。さらに、相互作用する2つのパートナーの相対的な配向は、生成されるシグナルの種類 (例えば、アゴニスト(agonism)対アンタゴニスト(antagonism)) に影響を与える可能性がある。したがって、ドッキングは、生成されるシグナルの強度と種類の両方を予測するのに有用である。
分子ドッキングは、低分子リガンドの適切なターゲット結合部位への結合コンフォメーションを予測できるため、構造に基づいた医薬品設計(structure-based drug design; SBDD)において最も頻繁に使用される手法の一つである。結合挙動の特性評価は、基本的な生化学的プロセスを解明するだけでなく、薬剤の合理的な設計においても重要な役割を果たしている[2]。
問題の定義
分子ドッキングは、「ロック(lock)」を開ける「キー(key)」の正しい相対的な向き(ロックの表面のどこに鍵穴があるか、キーを挿入した後にキーをどの方向に回すかなど)を見つけたいという、「ロックアンドキー」(lock-and-key)の問題と考えることができる。ここでは、タンパク質を「ロック」、リガンドを「キー」と考えることができる。分子ドッキングは最適化問題として定義されることがあり、興味のある特定のタンパク質に結合するリガンドの「ベストフィット」(best-fit)な配向を記述することになる。しかし、リガンドとタンパク質の両方が柔軟であるため、「ロックとキー」よりも「手袋をはめた」(hand-in-glove)のアナロジーがより適切である[3]。ドッキングプロセスの間、リガンドとタンパク質はコンフォメーションを調整して全体的な「ベストフィット」を達成する。この種のコンフォメーション調整により、全体的な結合が生じることを「誘導フィット」(induced-fit)と呼ぶ[4]。
分子ドッキング研究では、分子認識プロセスを計算機的にシミュレートすることに焦点を当てている。それは、タンパク質とリガンドの両方の最適なコンフォメーション、およびタンパク質とリガンドの相対的な配向を達成し、システム全体の自由エネルギーを最小化することを目的としている。
ドッキングアプローチ
分子ドッキングのコミュニティでは、2つのアプローチが特に人気がある。一つは、タンパク質とリガンドを相補的な表面として記述するマッチング技術を使用している[5][6][7]。二つ目のアプローチは、実際のドッキングプロセスをシミュレートし、リガンドとタンパク質のペアワイズ相互作用エネルギーを計算する[8]。どちらのアプローチにも大きな利点があり、いくつかの制限もある。これらを以下に概説する。
形状の相補性
幾何学的マッチング(geometric matching) / 形状相補性法(shape complementarity methods)は、タンパク質とリガンドをドッキング可能にする特徴の集合として記述する[9]。これらの特徴にはは、分子表面 (英語版) / 相補的表面記述子(complementary surface descriptors)を含んでもよい。この場合、受容体の分子表面は、その溶媒にアクセス可能な表面積 (英語版) の観点から説明され、リガンドの分子表面は、その相補的な(一致する)表面記述の観点から記述される。2つの表面間の相補性は、ターゲット分子とリガンド分子をドッキングする相補的な姿勢を見つけるのに役立つ可能性のある形状マッチングの説明になる。別のアプローチは、主鎖原子のターンを使用してタンパク質の疎水性の特徴を記述することである。さらに別のアプローチは、フーリエ形状記述子の技術を使用することである[10][11][12]。形状相補性に基づくアプローチは一般的に高速でロバスト(堅牢)であり、最近の開発によりこれらの方法でリガンドの柔軟性を調べることができるようになったものの、通常はリガンド/タンパク質のコンフォメーションの動きや動的変化を正確にモデル化することはできない。形状相補性法は数千個のリガンドを数秒でスキャンし、実際にそれらがタンパク質の活性部位に結合できるかどうかを把握することができ、通常はタンパク質とタンパク質の相互作用にまで拡張可能である。それらはまた、最適な結合を見つけるためにリガンドの幾何学的記述を使用するため、ファーマコフォアに基づくアプローチにも適している。
シミュレーション
ドッキングプロセスのシミュレーションははるかに複雑である。このアプローチでは、タンパク質とリガンドは物理的な距離だけ隔てられており、リガンドはそのコンフォメーション空間内で一定数の「移動」を行った後、タンパク質の活性部位にその位置を見つけだす。この移動には、並進や回転などの剛体変換だけでなく、ねじれ角回転などのリガンドの構造への内部変化も含まれている。リガンドのコンフォメーション空間におけるこれらの移動のそれぞれは、システムの総エネルギーコストを誘発する。したがって、システムの総エネルギーは、すべての移動の後に計算される。
ドッキングシミュレーションの明らかな利点は、リガンドの柔軟性を簡単に取り入れることができることであるのに対し、形状相補性技術では、リガンドの柔軟性を取り入れるには独創的な方法を用いなければならない。また、形状補完技術がより抽象的であるのに対し、シミュレーションはより正確に現実をモデル化することができる。
明らかに、シミュレーションは計算コストが高く、大規模なエネルギー地形(energy landscape)を探索しなければならない。グリッドベースの技術、最適化手法、コンピュータの高速化により、ドッキングシミュレーションがより現実的なものになった。
ドッキングのメカニズム
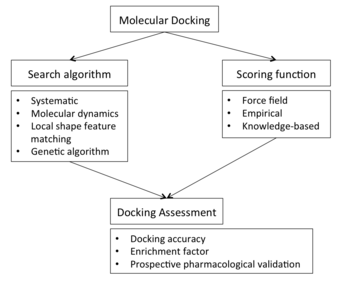
ドッキング・スクリーン(docking screen)を実行するために、最初の要件は、関心のあるタンパク質の構造である。通常、構造は、X線結晶構造解析、NMR分光法、低温電子顕微鏡法(クライオEM)などの生物物理学的手法を用いて決定されているが、ホモロジーモデル構築からも導き出すことができる。このタンパク質構造と潜在的なリガンドのデータベースは、ドッキングプログラムの入力として機能する。ドッキングプログラムが成功するかどうかは、検索アルゴリズムとスコアリング関数の2つのコンポーネントに依存する。
検索アルゴリズム
理論的には、探索空間は、リガンドと対になるタンパク質のすべての可能な配向(orientations)とコンフォメーション(conformations)から構成されている。しかし、実際には現在の計算リソースでは、探索空間を網羅的に探索することは不可能である。これには、各分子のすべての可能な歪み(分子は動的であり、コンフォーメーション状態のアンサンブル(ensemble; 調和)の中に存在している)と、与えられた粒度レベルでのタンパク質に対するリガンドのすべての可能な回転および並進の方向を列挙する必要がある。使用されているほとんどのドッキングプログラムは、リガンド (柔軟性のあるリガンド) のコンフォメーション空間全体を考慮しており、いくつかは柔軟性のあるタンパク質受容体をモデル化しようとしている。ペアの各「スナップショット」は、ポーズ(pose)と呼ばれている。
さまざまな立体配座の探索戦略が、リガンドと受容体に適用されてきた。これらには以下が含まれる:
- 回転可能な結合に関する系統的または確率的 (英語版) なねじれ角 (英語版) 探索
- 分子動力学シミュレーション
- 遺伝的アルゴリズムは、新しい低エネルギーコンフォメーションを「進化」させ、各ポーズのスコアが次の反復のための個体を選択するために使用されるフィットネス関数として機能する。
リガンドの柔軟性
リガンドのコンフォメーションは、受容体の不在時に生成され、その後ドッキングされてもよいし[13]、受容体結合空洞(receptor binding cavity)の存在時にオン・ザ・フライ(on-the-fly)で生成されてもよいし[14]、フラグメントベースのドッキングを使用して、すべての二面体角の完全な回転の柔軟性を持つコンフォメーションが生成されてもよい[15]。力場エネルギー評価(Force field energy evaluation)は、エネルギー的に合理的なコンフォメーションを選択するために最も頻繁に使用されるが[16]、知識ベースの方法も使用されている[17]。
ペプチドは非常に柔軟性が高く、比較的大きな分子であるため、その柔軟性をモデル化することは困難なタスクとなっている。タンパク質-ペプチドのドッキング時にペプチドの柔軟性を効率的にモデル化するために、多くの手法が開発されてきた[18]。
受容体の柔軟性
計算能力は過去10年間で飛躍的に向上し、コンピュータ支援薬物設計におけるより洗練された計算集約的な手法を使用できるようになった。しかし、ドッキング方法論における受容体の柔軟性を扱うことはまだ厄介な問題である[19]。この困難さの背後にある主な理由は、この種の計算で考慮しなければならない多数の自由度である。しかし、それを無視すると、いくつかのケースでは、結合ポーズの予測面でドッキング結果が乏しくなる可能性がある[20]。
異なるコンフォーメーションの同じタンパク質について実験的に決定された複数の静的構造は、しばしば、受容体の柔軟性をエミュレートするために使用される[21]。あるいは、結合空洞を取り囲むアミノ酸側鎖の回転異性体ライブラリを検索して、代替的ではあるがエネルギー的に合理的なタンパク質のコンフォメーションを生成してもよい[22][23]。
スコアリング関数
ドッキングプログラムは多数の潜在的なリガンドのポーズを生成するが、その中にはタンパク質との衝突のために即座に拒否されるものもある。残りはスコアリング関数を使って評価される。この関数は入力としてポーズを取り、そのポーズが好ましい結合相互作用を表す可能性(likelihood; 尤度; もっともらしさ)を示す数値を返し、あるリガンドを別のリガンドに対して相対的なランク付けを行う。
ほとんどのスコアリング関数は、物理学に基づいた分子力学的な力場であり、結合部位内のポーズのエネルギーを推定する。結合への様々な寄与は、加法方程式として書くことができる:
その構成要素は、溶媒効果(solvent effects)、タンパク質とリガンドの立体構造的変化(conformational changes)、タンパク質とリガンドの相互作用(interactions)による自由エネルギー、内部回転(internal rotations)、単一複合体を形成するためのリガンドと受容体の会合エネルギー(association energy)、振動モード(vibrational modes)の変化による自由エネルギーで構成されている[24]。低い(負の)エネルギーの場合は、システムが安定していることを示し、結合が相互作用している可能性がある。
別のアプローチとしては、蛋白質構造データバンク(Protein Data Bank; PDB)のようなタンパク質-リガンド複合体の大規模なデータベースから相互作用の知識ベースの統計的ポテンシャルを導出し、この推定されたポテンシャルに従ってポーズの適合性を評価する方法がある。
タンパク質と高親和性リガンドとの複合体については、X線結晶構造解析による構造が多数存在しますが、低親和性リガンドについては比較的少ないため、安定性が低く結晶化が難しい傾向にある。このデータを用いてトレーニングされたスコアリング関数は、高親和性リガンドを正しくドッキングさせることができるが、結合していないリガンドに対しても、もっともらしいドッキング・コンフォメーションを与える。これにより、多くの偽陽性ヒット、すなわち、タンパク質に結合すると予測されたリガンドが、試験管内に一緒に置かれたときに実際には結合しないと予測される。
偽陽性の数を減らす一つの方法は、一般化ボルン法(Generalized Born)やポアソン-ボルツマン法(Poisson-Boltzmann methods)のような、(潜在的に)より正確ではあるが計算量の多い手法を用いて、上位スコアのポーズのエネルギーを再計算することである[25]。
ドッキング評価
サンプリングとスコアリング機能の間の相互依存性は、新規化合物のための説得力のあるポーズや結合親和性を予測する際のドッキング能力に影響する。したがって、その予測能力を決定するためには、ドッキングプロトコルの評価が一般的に必要とされている(実験データが利用可能な場合)。ドッキング評価は、次のようなさまざまな戦略を使用して実行できる:
- ドッキング精度(DA)計算。
- ドッキングスコアと実験的応答または濃縮係数(EF)の決定との相関関係[26]。
- イオン結合部位と活性部位内のイオンとの間の距離。
- 誘導適合モデルの存在(induce-fit models)
ドッキング精度
ドッキング精度(docking accuracy; DA)は[27][28]、リガンドの正しいポーズを実験的に観測されたものと比較して予測する能力を合理化することによって、ドッキングプログラムの適合性を定量化する一つの指標となる[29]。
濃縮係数(Enrichment factor; EF)
ドッキング・スクリーンはまた、結合していないと推定される「デコイ (生化学)」分子の大規模なデータベースの中から、既知のバインダー(結合剤)のアノテーションされたリガンドを濃縮することによって評価することができる[30]。このようにして、ドッキング・スクリーンの成功は、データベース内の非常に多数のデコイ分子の中から、スクリーンの最上位にある少数の既知の活性化合物を濃縮する能力によって評価される。その性能を評価するために、受信者操作特性(ROC)曲線の下の面積が広く使われている。
見込み
ドッキング・スクリーン(docking screens)から得られた結果のヒットは、薬理学的検証 (例:IC50、親和性または力価 (薬理学)の測定) の対象となる。前向き研究(prospective studies)のみが、特定のターゲットに対する技術の適合性に対し、決定的な証拠を構成する[31]。
ベンチマーク
X線結晶構造解析によって決定された結合モードを再現するドッキングプログラムの可能性は、さまざまなドッキング・ベンチマーク・セットによって評価できる。
低分子については、ドッキングとバーチャル・スクリーニング(virtual screening)のためのいくつかのベンチマーク・データセットが存在する。例えば、高品質のタンパク質-リガンドのX線結晶構造[32]で構成される Astex Diverse Set やバーチャル・スクリーニングの性能を評価するための Directory of Useful Decoys (DUD)などがある[33]。
ペプチド結合モードを再現する可能性のあるドッキングプログラムの評価は、「ドッキングとスコアリングの効率評価」(Lessons for Efficiency Assessment of Docking and Scoring (LEADS-PEP))によって評価することができる[34]。
アプリケーション
低分子リガンドと酵素タンパク質との間の結合相互作用は、酵素の活性化または阻害をもたらし得る。タンパク質が受容体である場合、リガンドの結合は、アゴニズムまたはアンタゴニズムをもたらす可能性がある。ドッキングは医薬品設計の分野で最も一般的に使用されている - ほとんどの薬物は小さな有機分子であり、ドッキングは次のような場合に適用される:
- ヒット同定(hit identification) - スコアリング関数と組み合わせたドッキングを使用して、潜在的な薬物の大規模なデータベースを「イン・シリコ」(in silico)ですばやくスクリーニングし、目的のタンパク質ターゲットに結合する可能性のある分子を同定できる(バーチャル・スクリーニングを参照)。
- リード最適化(lead optimization) - ドッキングは、リガンドがタンパク質に結合する場所と相対的な配向 (結合モードやポーズとも呼ばれる) を予測するために使用できる。この情報を使用して、より強力で選択的なアナログを設計できる。
- バイオレメディエーション(bioremediation) - タンパク質リガンドドッキングは、酵素によって分解される可能性のある汚染物質を予測するためにも使用できる[35][36]。
参照項目
- 医薬品設計 (英語版)
- Katchalski-Katzir algorithm (英語版)
- 分子グラフィックスシステムのリスト (英語版)
- 高分子ドッキング (英語版)
- 分子力学法
- タンパク質構造
- タンパク質設計 (英語版)
- 分子力学モデリング用ソフトの比較
- タンパク質-リガンドドッキングソフトウェアの比較 (英語版)
- 分子設計ソフトウェア (英語版)
- Docking@Home
- Ibercivis (英語版)
- ZINCデータベース (英語版)
- Lead Finder (英語版)
- バーチャル・スクリーニング
- スコアリング関数 (分子)
参考文献
- ^ “Computational methods for biomolecular docking”. Current Opinion in Structural Biology 6 (3): 402–6. (Jun 1996). doi:10.1016/S0959-440X(96)80061-3. PMID 8804827.
- ^ “Docking and scoring in virtual screening for drug discovery: methods and applications”. Nature Reviews. Drug Discovery 3 (11): 935–49. (Nov 2004). doi:10.1038/nrd1549. PMID 15520816.
- ^ “Rusting of the lock and key model for protein-ligand binding”. Science 254 (5034): 954–5. (Nov 1991). doi:10.1126/science.1719636. PMID 1719636.
- ^ “Testing a flexible-receptor docking algorithm in a model binding site”. Journal of Molecular Biology 337 (5): 1161–82. (Apr 2004). doi:10.1016/j.jmb.2004.02.015. PMID 15046985.
- ^ “QSD quadratic shape descriptors. 2. Molecular docking using quadratic shape descriptors (QSDock)”. Proteins 38 (1): 79–94. (2000). doi:10.1002/(SICI)1097-0134(20000101)38:1<79::AID-PROT9>3.0.CO;2-U. PMID 10651041.
- ^ “Automated docking with grid-based energy evaluation”. Journal of Computational Chemistry 13 (4): 505–524. (1992). doi:10.1002/jcc.540130412.
- ^ “Automated docking using a Lamarckian genetic algorithm and an empirical binding free energy function”. Journal of Computational Chemistry 19 (14): 1639–1662. (1998). doi:10.1002/(SICI)1096-987X(19981115)19:14<1639::AID-JCC10>3.0.CO;2-B.
- ^ “Performance comparison of generalized born and Poisson methods in the calculation of electrostatic solvation energies for protein structures”. Journal of Computational Chemistry 25 (2): 265–84. (Jan 2004). doi:10.1002/jcc.10378. PMID 14648625.
- ^ “Molecular docking using shape descriptors”. Journal of Computational Chemistry 13 (3): 380–397. (2004). doi:10.1002/jcc.540130311.
- ^ “Protein-ligand recognition using spherical harmonic molecular surfaces: towards a fast and efficient filter for large virtual throughput screening”. Journal of Molecular Graphics & Modelling 20 (4): 313–28. (Jan 2002). doi:10.1016/S1093-3263(01)00134-6. PMID 11858640.
- ^ “Real spherical harmonic expansion coefficients as 3D shape descriptors for protein binding pocket and ligand comparisons”. Bioinformatics 21 (10): 2347–55. (May 2005). doi:10.1093/bioinformatics/bti337. PMID 15728116.
- ^ “Shape variation in protein binding pockets and their ligands”. Journal of Molecular Biology 368 (1): 283–301. (Apr 2007). doi:10.1016/j.jmb.2007.01.086. PMID 17337005.
- ^ “Flexibases: a way to enhance the use of molecular docking methods”. Journal of Computer-Aided Molecular Design 8 (5): 565–82. (Oct 1994). doi:10.1007/BF00123666. PMID 7876901.
- ^ “Glide: a new approach for rapid, accurate docking and scoring. 1. Method and assessment of docking accuracy”. Journal of Medicinal Chemistry 47 (7): 1739–49. (Mar 2004). doi:10.1021/jm0306430. PMID 15027865.
- ^ “eHiTS: a new fast, exhaustive flexible ligand docking system”. Journal of Molecular Graphics & Modelling 26 (1): 198–212. (Jul 2007). doi:10.1016/j.jmgm.2006.06.002. PMID 16860582.
- ^ Romesberg, Floyd, ed (September 2007). “Preference of small molecules for local minimum conformations when binding to proteins”. PLOS ONE 2 (9): e820. doi:10.1371/journal.pone.0000820. PMC 1959118. PMID 17786192.
- ^ “A fast and efficient method to generate biologically relevant conformations”. Journal of Computer-Aided Molecular Design 8 (5): 583–606. (October 1994). doi:10.1007/BF00123667. PMID 7876902.
- ^ “Protein-peptide docking: opportunities and challenges”. Drug Discovery Today 23 (8): 1530–1537. (May 2018). doi:10.1016/j.drudis.2018.05.006. PMID 29733895.
- ^ “Understanding the challenges of protein flexibility in drug design”. Expert Opinion on Drug Discovery 10 (12): 1301–13. (December 2015). doi:10.1517/17460441.2015.1094458. hdl:1911/88215. PMID 26414598.
- ^ “MADAMM: a multistaged docking with an automated molecular modeling protocol”. Proteins 74 (1): 192–206. (January 2009). doi:10.1002/prot.22146. PMID 18618708.
- ^ “Flexible ligand docking to multiple receptor conformations: a practical alternative”. Current Opinion in Structural Biology 18 (2): 178–84. (Apr 2008). doi:10.1016/j.sbi.2008.01.004. PMC 2396190. PMID 18302984.
- ^ “Docking and scoring with alternative side-chain conformations”. Proteins 74 (3): 712–26. (Feb 2009). doi:10.1002/prot.22189. PMID 18704939.
- ^ “FDS: flexible ligand and receptor docking with a continuum solvent model and soft-core energy function”. Journal of Computational Chemistry 24 (13): 1637–56. (Oct 2003). doi:10.1002/jcc.10295. PMID 12926007.
- ^ “Computational Methods to Predict Binding Free Energy in Ligand-Receptor Complexes”. Journal of Medicinal Chemistry 38 (26): 4953–67. (Dec 1995). doi:10.1021/jm00026a001. PMID 8544170.
- ^ “Performance comparison of generalized born and Poisson methods in the calculation of electrostatic solvation energies for protein structures”. Journal of Computational Chemistry 25 (2): 265–84. (Jan 2004). doi:10.1002/jcc.10378. PMID 14648625.
- ^ “Benchmarking sets for molecular docking”. Journal of Medicinal Chemistry 49 (23): 6789–801. (Nov 2006). doi:10.1021/jm0608356. PMC 3383317. PMID 17154509.
- ^ “An Automated Strategy for Binding-Pose Selection and Docking Assessment in Structure-Based Drug Design”. Journal of Chemical Information and Modeling 56 (1): 54–72. (Jan 2016). doi:10.1021/acs.jcim.5b00603. PMID 26682916.
- ^ “Comparative study of several algorithms for flexible ligand docking”. Journal of Computer-Aided Molecular Design 17 (11): 755–63. (Nov 2003). doi:10.1023/B:JCAM.0000017496.76572.6f. PMID 15072435.
- ^ Ballante, Flavio (2018). Protein-Ligand Docking in Drug Design: Performance Assessment and Binding-Pose Selection. 1824. 67–88. doi:10.1007/978-1-4939-8630-9_5. ISBN 978-1-4939-8629-3. ISSN 1940-6029. PMID 30039402
- ^ “Benchmarking sets for molecular docking”. Journal of Medicinal Chemistry 49 (23): 6789–801. (Nov 2006). doi:10.1021/jm0608356. PMC 3383317. PMID 17154509.
- ^ “Community benchmarks for virtual screening”. Journal of Computer-Aided Molecular Design 22 (3–4): 193–9. (2008-02-14). doi:10.1007/s10822-008-9189-4. PMID 18273555.
- ^ “Diverse, high-quality test set for the validation of protein-ligand docking performance”. Journal of Medicinal Chemistry 50 (4): 726–41. (Feb 2007). doi:10.1021/jm061277y. PMID 17300160.
- ^ “Benchmarking sets for molecular docking”. Journal of Medicinal Chemistry 49 (23): 6789–801. (Nov 2006). doi:10.1021/jm0608356. PMC 3383317. PMID 17154509.
- ^ “A Benchmark Data Set for Assessment of Peptide Docking Performance”. Journal of Chemical Information and Modeling 56 (1): 188–200. (Dec 2015). doi:10.1021/acs.jcim.5b00234. PMID 26651532.
- ^ “An in silico [correction of insilico] approach to bioremediation: laccase as a case study”. Journal of Molecular Graphics & Modelling 26 (5): 845–9. (Jan 2008). doi:10.1016/j.jmgm.2007.05.005. PMID 17606396.
- ^ “Implications of Molecular Docking Assay for Bioremediation”. Data Analytics in Medicine: Concepts, Methodologies, Tools, and Applications. IGI Global. (2020). pp. 1556-1577. ISBN 1799812049
外部リンク
- “Molecular Docking Server - Ligand Protein Docking & Molecular Modeling”. Virtua Drug Ltd. 2008年7月15日閲覧。 “Internet service that calculates the site, geometry and energy of small molecules interacting with proteins”
- Malinauskas T. “Step by step installation of MGLTools 1.5.2 (AutoDockTools, Python Molecular Viewer and Visual Programming Environment) on Ubuntu Linux 8.04”. 2009年2月26日時点のオリジナルよりアーカイブ。2008年7月15日閲覧。
- Docking@GRID - グリッド上のコンフォメーションサンプリングおよびドッキングのプロジェクト。1つの目的は、計算グリッドにいくつかの固有の分散ドッキングアルゴリズムを展開し、 Docking@GRIDオープンソースLinuxバージョンをダウンロードすることでである
- Click2Drug.org - 計算薬物設計ツールのディレクトリ。
- Ligand:Receptor Docking - MOE (Molecular Operating Environment)

