非相同組換え
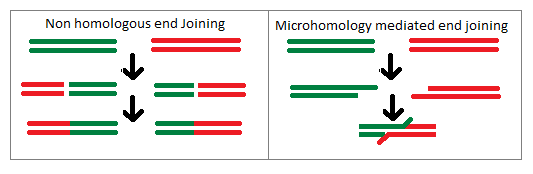
非相同組換え(ひそうどうくみかえ、英: nonhomologous recombination, illegitimate recombination)は、2つの無関係なDNA二本鎖断片が連結される過程である。こうした過程で本来隣接していない遺伝物質が挿入されることで、遺伝子が破壊され、その遺伝子がコードするタンパク質が正しく発現しなくなることがある。その主要な経路の1つが、非相同末端結合(NHEJ)と呼ばれる修復機構である[1]。
発見[編集]
非相同組換えは、大腸菌Escherichia coliで最初に発見された、自然に存在する過程である。700から1400塩基対のDNA断片がgalやlacオペロンに挿入され、強力な極性変異を引き起こすことが発見された[2]。その後、この機構は他の短い遺伝子配列も細菌のゲノム内の他の部位へ挿入し、それによってしばしば隣接する遺伝子の発現の変化が引き起こされることが発見された。多くの場合、隣接する遺伝子は単純にオフになるだけであるが、こうした断片の一部には強いスタートシグナルとストップシグナルを持ち、隣接する遺伝子の調節を変化させ、転写量の変化を引き起こすものがある。この遺伝子組換え機構は相同なDNA断片を用いる必要がない点で、遺伝的相同性に依存した機構とは異なる。その当時はまだ完全には理解されていなかったが、この機構は染色体の進化に変化をもたらす可能性があることが知られるようになった[3]。
機構[編集]
原核生物[編集]
原核生物における非相同組換えによる遺伝子配列の変異は真核生物とは異なる形で行われるが、その1つが欠失である。欠失変異では、非相同組換えによって遺伝子配列の連続した断片が除去される。こうした変異はファージの誘発後に高頻度で生じる。原核生物における非相同組換えの他の形態は、ゲノムの重複変異である。この場合、親ゲノムの一部がゲノムへ複数回挿入される。この重複変異は相同性に基づかずに行われるため、元の断片と同じ向きで挿入される場合も反対向きに挿入される場合もある[3]。
真核生物[編集]
真核生物における非相同組換えの主要な機構は非末端相同結合(NHEJ)であり、相同性を持たない2つのDNA鎖が遺伝子修復機構によって連結される。二本鎖切断の認識に伴って、鎖の修復が可能となるようタンパク質複合体が2つの鎖を十分に近接させた状態に維持する。続いて、DNA末端の正しくないヌクレオチドや損傷したヌクレオチドが除去され、修復される。その後DNA鎖どうしがライゲーションされ、1本のDNA鎖となる。この過程は真核生物細胞で広くみられ、修復機構として作用する傾向があるが、非相同組換えが生じた場合には変異が引き起こされる場合がある。真核生物は原核生物よりもはるかに大きなDNA断片を持つため、非相同組換えは大きな染色体異常の形をとることが多い。非相同末端結合は、染色体への挿入変異や欠失変異、ある染色体断片から別の染色体断片への転座を生み出す非相同組換えを引き起こす可能性がある。こうした真核生物の染色体の大規模な変化は、遺伝的な有利さよりもむしろ個体に有害な影響を与える傾向がある[4]。
有害な影響[編集]
非相同組換えは個体の遺伝子配列に大規模な変化を引き起こすため、有害な影響を及ぼすことが多い。こうした変化は、相同性に基づかないDNAの連結によって遺伝子エレメントがそれまで位置していた場所ではない場所に配置されることによるものである。その結果、生物の機能に必要不可欠な遺伝子の機能が破壊される可能性がある。がんの場合には、非相同組換えによるヘアピン構造の形成によって遺伝子の機能の変化が引き起こされ、腫瘍が形成されることがあることが知られている[5]。
応用[編集]
非相同組換えは実験室的に利用されるツールであり、有用な研究ツールである。非相同組換えはランダム突然変異を誘発し、個体の遺伝子配列にランダムな変化を引き起こす[6]。こうしたランダム変異導入による遺伝子断片の機能の変化は、遺伝子配列の研究へ利用することができる。変異体と野生型の差異を解析することでその遺伝子の機能を研究し、その遺伝子がどの過程に関連しているのかを解釈することでができる。
出典[編集]
- ^ “Nonhomologous end-joining: mechanisms, conservation and relationship to illegitimate recombination.”. Molecular Genetics of Recombination. Topics in Current Genetics. 17. Berlin, Heidelberg: Springer. (2006). pp. 487–513. doi:10.1007/978-3-540-71021-9_17. ISBN 978-3-540-71020-2
- ^ “Illegitimate recombination legitimised”. Nature 274 (5668): 213–4. (July 1978). Bibcode: 1978Natur.274..213S. doi:10.1038/274213a0. PMID 355886.
- ^ a b “Illegitimate recombination in bacteria and bacteriophage”. Annual Review of Genetics 11 (1): 451–73. (December 1977). doi:10.1146/annurev.ge.11.120177.002315. PMID 339822.
- ^ “Mechanisms of illegitimate recombination”. Gene 135 (1–2): 161–6. (December 1993). doi:10.1016/0378-1119(93)90061-7. PMID 8276254.
- ^ “Chromosome translocation based on illegitimate recombination in human tumors”. Proceedings of the National Academy of Sciences of the United States of America 95 (20): 11786–91. (September 1998). Bibcode: 1998PNAS...9511786Z. doi:10.1073/pnas.95.20.11786. PMC 21718. PMID 9751743.
- ^ “Illegitimate recombination: an efficient method for random mutagenesis in Mycobacterium avium subsp. hominissuis”. BMC Microbiology 12: 204. (September 2012). doi:10.1186/1471-2180-12-204. PMC 3511198. PMID 22966811.
