重鎖抗体
重鎖抗体(じゅうさこうたい、英: heavy-chain antibody)は、2本の重鎖のみで構成され、通常の抗体に含まれる2本の軽鎖を持たない抗体である。
一般的な抗体では、抗原結合領域は重鎖および軽鎖の可変ドメイン(VHとVL)から構成されている。重鎖抗体はVHドメインしか持たないにもかかわらず、抗原に結合することができる。この観察結果から、医薬品としての利用が期待できる新しいタイプの抗体フラグメント、いわゆるシングルドメイン抗体が開発された[1]。
発見[編集]
1989年、ブリュッセル自由大学のレイモンド・ハマーズ率いる生物学者のグループは、ヒトコブラクダの免疫系を調査した。予想されていた4本鎖の抗体に加えて、彼らは2本の重鎖のみからなるより単純な抗体を同定した。この発見は1993年に Nature 誌に掲載された[2]。1995年、マイアミ大学の研究チームは、サメから別の種類の重鎖抗体を発見した[3]。
軟骨魚類[編集]
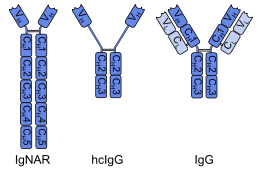
軟骨魚類(サメなど)の免疫グロブリン新抗原受容体(IgNAR)は、重鎖抗体である。IgNARは、他の抗体とは構造面で大きな相違がある。これは、1本の鎖に通常の3つではなく5つの定常ドメイン(CH)があり、珍しい位置にいくつかのジスルフィド結合があり、相補性決定領域3(CDR3)は他の抗体の軽鎖と結合する部位を覆う延長ループを形成している。これらの違いは、軟骨魚類の系統発生的な年代とあいまって、IgNARは哺乳類の免疫グロブリンよりも原始的な抗原結合タンパク質に近い可能性があるという仮説につながった。この仮説を検証するためには、無顎魚類のヤツメウナギやヌタウナギのように、系統発生的にさらに古い脊椎動物からIgNARまたは同様の抗体を発見する必要がある[4]。無脊椎動物には抗体は全くない。
サメ(およびおそらく他の軟骨魚類も)には、免疫グロブリンM(IgM)と免疫グロブリンW(IgW)があり、どちらも2本の重鎖と2本の軽鎖を持つ種類である[5]。
ラクダ科[編集]
重鎖抗体(IgGのような)を持つ哺乳類は、ヒトコブラクダ、ラクダ、ラマ、アルパカなどのラクダ科動物のみである[6]。これは二次的な発達である。これらの抗体の重鎖は、定常ドメインの1つ(CH1)を失い、可変ドメイン(VH)が修飾を受けており、いずれも軽鎖との結合に必要な構造要素である。あるサブグループでは、画像のように、欠落したCH1が拡張ヒンジ領域に置き換えられているようである[1][2]。全体的な構造が異なるにもかかわらず、ラクダ科の重鎖抗体は、拡張されたCDR3ループやCDR1のコンフォメーションなど、IgNARといくつかの特性を共有している。このような類似性は、純粋な関係ではなく、機能的な要求や収斂進化によるものだと考えられている[4]。
ラクダ科動物の抗体の約50%は、通常の哺乳類の重鎖/軽鎖型である。どのような種類の動物が重鎖抗体のみを持ち、2本の重鎖と2本の軽鎖を持つ一般的な種類を完全に欠いているかどうかは不明である。
ラクダ科動物の重鎖抗体は、通常の抗体と同じように特異的であることがわかっており、場合によってはより頑健である。また、従来の抗体と同じファージパニング法を使用して簡単に分離できるため、生体外(ex vivo)で高濃度で培養することができる。サイズが小さく、ドメインが単一であるため、細菌細胞に形質転換して大量生産することが容易であり、研究目的に最適な抗体となる[7]。
脚注[編集]
- ^ a b Harmsen, M. M.; Haard, H. J. (2007). “Properties, production, and applications of camelid single-domain antibody fragments”. Applied Microbiology and Biotechnology 77 (1): 13–22. doi:10.1007/s00253-007-1142-2. PMC 2039825. PMID 17704915.
- ^ a b Hamers-Casterman, C; Atarhouch, T; Muyldermans, S; Robinson, G; Hamers, C; Songa, EB; Bendahman, N; Hamers, R (3 June 1993). “Naturally occurring antibodies devoid of light chains”. Nature 363 (6428): 446–8. Bibcode: 1993Natur.363..446H. doi:10.1038/363446a0. PMID 8502296.
- ^ Greenberg, A.S.; Avila, D.; Hughes, M.; Hughes, A.; McKinney, E.C.; Flajnik, M.F. (1995). “A new antigen receptor gene family that undergoes rearrangement and extensive somatic diversification in sharks”. Nature 374 (6518): 168–173. Bibcode: 1995Natur.374..168G. doi:10.1038/374168a0. PMID 7877689.
- ^ a b Stanfield, R.; Dooley, H.; Flajnik, M.; Wilson, I. (2004). “Crystal structure of a shark single-domain antibody V region in complex with lysozyme”. Science 305 (5691): 1770–1773. Bibcode: 2004Sci...305.1770S. doi:10.1126/science.1101148. PMID 15319492.
- ^ Flajnik, M. F.; Dooley, H. (2009). The Generation and Selection of Single-Domain, V Region Libraries from Nurse Sharks. Methods in Molecular Biology. 562. pp. 71–82. doi:10.1007/978-1-60327-302-2_6. ISBN 978-1-60327-301-5. PMID 19554288
- ^ Conrath, K. E.; Wernery, U.; Muyldermans, S.; Nguyen, V. K. (2003). “Emergence and evolution of functional heavy-chain antibodies in Camelidae”. Developmental and Comparative Immunology 27 (2): 87–103. doi:10.1016/S0145-305X(02)00071-X. PMID 12543123.
- ^ Ghannam, A., Kumari, S., Muyldermans, S., & Abbady, A. Q. (2015). Camelid nanobodies with high affinity for broad bean mottle virus: a possible promising tool to immunomodulate plant resistance against viruses. Plant Molecular Biology, 1-15.
外部リンク[編集]
- Wikilite:免疫グロブリン軽鎖の生物学
