Hsp90
| Histidine kinase-, DNA gyrase B-, and HSP90-like ATPase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 ATPを結合した酵母Hsp90二量体のリボンモデル(αヘリックス=赤、βシート=シアン、ループ=灰色、ATP=スティックモデル)[1] | |||||||||
| 識別子 | |||||||||
| 略号 | HATPase_c | ||||||||
| Pfam | PF02518 | ||||||||
| Pfam clan | CL0025 | ||||||||
| InterPro | IPR003594 | ||||||||
| SMART | SM00387 | ||||||||
| SCOP | 1ei1 | ||||||||
| SUPERFAMILY | 1ei1 | ||||||||
| |||||||||
| Hsp90 protein | |||||||||
|---|---|---|---|---|---|---|---|---|---|
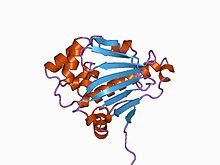 酵母Hsp90のN末端ドメインの構造[2] | |||||||||
| 識別子 | |||||||||
| 略号 | Hsp90 | ||||||||
| Pfam | PF00183 | ||||||||
| InterPro | IPR020576 | ||||||||
| PROSITE | PDOC00270 | ||||||||
| SCOP | 1ah6 | ||||||||
| SUPERFAMILY | 1ah6 | ||||||||
| |||||||||




Hsp90(heat shock protein 90 kDa)は、他のタンパク質が正しくフォールディングするよう補助し、熱ストレスに対する安定化をもたらし、また分解を助けるシャペロンタンパク質である。腫瘍成長に必要ないくつかのタンパク質に対しても安定化作用を示すため、Hsp90阻害剤は抗がん剤としての研究が行われている。
熱ショックタンパク質に分類されるタンパク質群は、全ての生物種を通じて最も高度に発現しているものの1つである[3]。その名称が示す通り、熱ショックタンパク質は温度上昇によるストレス時に細胞を保護する役割を果たす。こうしたタンパク質はストレスを受けていない細胞でも全タンパク質の1–2%を占めているが、細胞が加熱された場合には4–6%にまで増加する[4]。
Hsp90は、こうした熱関連タンパク質の中でも最も広く存在するものの1つである。90という数字はそのサイズが約90 kDaであることに由来している。Hsp90は細菌に存在し、真核生物でも全ての分類群に存在しているが、古細菌には存在しないようである[5]。真核生物では、細胞質のHsp90はあらゆる状況下で生存に必須であるが、細菌ホモログであるHtpGは熱ストレス条件下以外では必須ではない[6]。
Hsp90は、熱、脱水やその他、タンパク質の変性が生じるさまざまな方法でストレス処理された細胞からタンパク質を抽出することで単離され[7]、後に非ストレス下の細胞においても必須の機能を果たしていることが発見された。
アイソフォーム[編集]
哺乳類細胞には、細胞質基質型のHsp90ホモログをコードする遺伝子が2つ以上存在し[5]、ヒトのHsp90αとHsp90βは85%の配列同一性を示す[8]。Hsp90αとHsp90βは、数百万年前に生じた遺伝子重複によって分岐したものであると考えられている[5]。
Hsp90アイソフォームをコードする5種類のヒト遺伝子を下に示す[8]。
| ファミリー | 細胞内局在 | サブファミリー | 遺伝子 | タンパク質 |
|---|---|---|---|---|
| HSP90A | 細胞質基質 | HSP90AA (誘導性) |
HSP90AA1 | Hsp90α1 |
| HSP90AA2 | Hsp90α2 | |||
| HSP90AB (恒常的発現) |
HSP90AB1 | Hsp90β | ||
| HSP90B | 小胞体 | HSP90B1 | Endoplasmin/ GRP-94 | |
| TRAP | ミトコンドリア | TRAP1 | TRAP1 (TNF Receptor- Associated Protein 1) |
Hsp90Nと呼ばれる、ATP結合部位を欠いた、膜結合型の細胞質基質Hsp90が報告されている[9]。Hsp90Nをコードする転写産物はキメラであり、コーディング配列の最初の105 bpはCD47遺伝子に由来し、残りのコーディング配列はHSP90AA1に由来するものである[8]。しかしながら、Hsp90Nをコードする遺伝子はヒトゲノムには存在せず、クローニング時のアーティファクトか単一の細胞株にのみ生じた染色体組換えによる産物である可能性が高いと考えられている[10]。
構造[編集]
ドメイン構造[編集]
Hsp90は4つの構造ドメインから構成される[11][12][13]。
- 高度に保存された約25 kDaのN末端ドメイン(NTD)
- N末端ドメインとミドルドメインを連結する荷電したリンカー領域
- 約40 kDaのミドルドメイン(MD)
- 約12 kDaのC末端ドメイン(CTD)
阻害剤やヌクレオチドを結合した酵母やヒトHsp90のN末端ドメイン[14][15][16]、酵母Hsp90のミドルドメインの結晶構造が解かれている[17]。また、大腸菌(2IOP、2IOQ)[18]、酵母(2CG9、2CGE)[19]、イヌ小胞体(2O1U、2O1V)[20]由来のHsp90の全長構造も明らかにされている[21]。
Hsp90はホモ二量体を形成し、開いたコンフォメーションでは主にC末端ドメインが単量体間の接触を担っている。閉じたコンフォメーションでは、N末端ドメインも互いに接近して接触を行う[17]。
N末端ドメイン[編集]
N末端ドメインはHsp90ファミリー内で相同性を示すだけでなく、GHKLドメイン(ジャイレース(Gyrase)、Hsp90、ヒスチジンキナーゼ(Histidine Kinase)、MutL)スーパーファミリーのATPアーゼ/キナーゼの間でも相同である[12]。
ATPと阻害剤ゲルダナマイシンの結合ポケットは共通しており、N末端ドメインに位置している。酵母Hsp90(Hsc82)においてATPとの相互作用に直接関与するのは、Leu34、Asn37、Asp79、Asn92、Lys98、Gly121、Phe124である。さらに、Mg2+と水分子がHsp90とATPの間の静電的相互作用と水素結合をそれぞれ媒介している。ATPの加水分解にはGlu33が必要である。
ミドルドメイン[編集]
ミドルドメインは、N末端側からα-β-αサンドイッチ、ヘリカルコイル、そしてもう1つのα-β-αサンドイッチという3つの領域に分けられる[12]。
MDはクライアントタンパク質の結合に関与している。MDと相互作用することが知られているタンパク質には、PKB/Akt1、eNOSなどがある[22][23]。さらに、Aha1やHch1といったコシャペロンもMDに結合し、ATPアーゼ活性を高めることが知られている[17][24]。
C末端ドメイン[編集]
C末端ドメインには副次的ATP結合部位が存在し、N末端ドメインのBergerat型ポケットにATPが結合している際にアクセス可能となる[25][26]。
最もC末端の領域にはTPRモチーフによる認識部位である保存されたMEEVD配列が存在し、イムノフィリン(FKBP51やFKBP52)、STIP1(Hop)、シクロフィリンD、PP5、Tom70などのコファクターとの相互作用を担っている[27]。
機構[編集]
Hsp90には、ATPの結合、タンパク質の結合、そして二量体化を担うドメインが存在し、それぞれがタンパク質の機能に重要な役割を果たしている。
ATPの結合[編集]
N末端ドメインには、高親和性のATP結合部位が存在する。ATPはタンパク質の側面に存在する溝に結合し、そのポケットの深さは約15 Åである。このポケットはATPに対して高い親和性を有し、Hsp90は適切なタンパク質基質の存在下でATPをADPと無機リン酸へ分解する。ATP結合の直接阻害剤、もしくはATP結合やATPアーゼ活性のアロステリック阻害剤によって、Hsp90の機能は遮断される[28]。Hsp90のATP結合領域について興味深い他の特徴としては、ADP結合時には開き、ATP結合時には閉じる「ふた」が存在することである[29]。このふたは、開いた状態ではタンパク質内相互作用を行わないが、閉じた際にはいくつかの残基と接触する[30]。このふたがHsp90の活性に与える影響に関しては部位特異的変異導入による解析が行われており、閉じた状態を安定化するAla107Asn変異体は新たな水素結合を形成することでATPアーゼ活性を大きく高める一方、AMP-PNP結合型コンフォメーションには影響しない[30]。
Hsp90のATP結合領域は、このタンパク質を標的とする薬剤の主要な結合部位であるため、精力的に研究が行われている[31]。Hsp90のこの部位を標的とする抗腫瘍活性物質には、ゲルダナマイシン[28][32]、ハービマイシン、ラジシコール、デグエリン[33]、デルボン[34]、マクベシン[35]やβ-ラクタム系化合物[36]がある。
タンパク質の結合[編集]
Hsp90は、開いたコンフォメーションと閉じたコンフォメーションの大きく2つのコンフォメーションを取る。ATPの加水分解はいわゆる"pincer"型と呼ばれるコンフォメーション変化を引き起こす[37]。
開いたコンフォメーションのHsp90では一部の疎水性残基が露出しており、フォールディングしていない、または誤ったフォールディングをしたタンパク質の疎水性領域がこの部位へ高い親和性でリクルートされる[38]。基質が正しい位置に結合すると、N末端ドメインのATPアーゼ機能によってコンフォメーション変化が引き起こされ、Hsp90は閉じて基質を締め付ける[30]。GyrBやMutLなどのクランプタンパク質と同様の反応によって、Hsp90はタンパク質フォールディング機能を発揮する。GyrBやMutLは正に帯電した側鎖に富むクランプによってDNAの負に帯電した骨格を結合し、トポイソメラーゼとして機能するが[39]、Hsp90は主に疎水的結合を利用して機能する。こうしたHsp90のクランプとしての機能は、フォールディングの補助、凝集の防止、輸送の促進などいくつかの機能を果たしている。
機能[編集]
正常細胞[編集]
ストレスを受けていない細胞では、Hsp90はタンパク質のフォールディング、細胞内輸送、維持、分解、シグナル伝達の促進など、いくつかの重要な役割を果たしている。
タンパク質のフォールディングとシャペロンとしての役割[編集]
Hsp90は多くのタンパク質の非ネイティブ構造と結合していることが知られており、Hsp90はタンパク質のフォールディングに対して全般的な関与を行っていると考えられている[40]。さらに、Hsp90は広範囲のクライアント(基質)タンパク質の凝集を抑制することが示されており、全般的な保護機能を示すシャペロンとして機能している[41][42][43]。しかしながら、Hsp90は他のシャペロンよりも若干の基質選択性がみられる[44]。
タンパク質の分解[編集]
真核生物では、不要となったタンパク質や誤ったフォールディングをしたタンパク質、損傷したタンパク質は、通常ポリユビキチン化経路によって分解のための標識が付加される。こうしたユビキチン化タンパク質は26Sプロテアソームによって認識されて分解される[45][46]。このように、26Sプロテアソームは細胞のタンパク質分解機構の重要な部分をなしているが、その三次構造の維持には機能的なHsp90の継続的供給が必要である[47]。また、熱感受性Hsp90変異体と26Sプロテアソームを用いて行われた実験では、プロテアソームのATPアーゼ活性は、全てではないもののその大部分をHsp90が担っていることが示唆されている[45]。
ステロイド受容体との相互作用[編集]


グルココルチコイド受容体(GR)は、その機能がHsp90との相互作用に大きく依存しているステロイドホルモン受容体として、最も詳細な研究がなされている[49][50]。ステロイドホルモンであるコルチゾールが存在しない場合にはGRは細胞質基質に位置し、Hsp90を含むいくつかのシャペロンタンパク質と複合体を形成している。こうしたシャペロンはGRをホルモンの結合が可能な状態に維持している。Hsp90のもう1つの役割は、GR複合体をダイニンによるタンパク質輸送経路へ結びつけるイムノフィリン(FKBP52など)を結合することである。その結果、活性化された受容体は細胞質から核内へ移行する[51]。核内ではGRは二量体化し、DNA上の特定の配列に結合してGR応答性遺伝子の発現をアップレギュレーションする。ミネラロコルチコイド受容体(アルドステロンを結合)[52]、アンドロゲン受容体(アンドロゲン)[53]、エストロゲン受容体(エストロゲン)[54]、プロゲステロン受容体(プロゲステロン)[55]など、他のいくつかのステロイド受容体の適切な機能にもHsp90は必要である。
がん細胞[編集]
がん細胞は、EGFRなどの成長因子受容体[56]、PI3KやAKTなどのシグナル伝達タンパク質(これらの阻害はアポトーシスを引き起こす)といったいくつかのタンパク質を過剰発現している。Hsp90はさまざまな成長因子受容体[57]、PI3KやAKTなど一部のシグナル伝達タンパク質を安定化しているため、Hsp90の阻害によってPI3K/AKT経路はダウンレギュレーションされ、抗アポトーシスタンパク質Bcl-wのダウンレギュレーションによってがん細胞や老化細胞のアポトーシスが引き起こされる[14][58][59]。
ナノ治療によるHsp90の阻害は、乳がんの薬剤抵抗性を低下させ、NK細胞の抑制を緩和することが示されている[60]。がんにおけるHsp90の他の重要な役割は、v-Src、Bcr/Abl融合タンパク質、形質転換時に出現する変異型p53といった変異体タンパク質の安定化である。Hsp90は、変異によって生まれた安定性の低いタンパク質を保護する役割を果たしているようである[61]。
Hsp90は血管内皮細胞増殖因子(VEGF)や一酸化窒素合成酵素(NOS)の誘導にも必要である[22]。これらの因子は、腫瘍が組織内での酸素の拡散距離の限界以上に成長するために必要とされるde novo血管新生に重要な役割を果たしている[61]。また、Hsp90はマトリックスメタロプロテアーゼMMP2を補助することで、転移の浸潤段階を促進する[62]。Hsp90はコシャペロンとともに、AKT[23]、TNF受容体、NK-κBの機能を介して腫瘍細胞のアポトーシスを調節する[63]。Hsp90は、成長シグナルの自己充足、変異タンパク質の安定化、血管新生、転移といった発がんの重要過程の多くに関与している。
臨床的意義[編集]
Hsp90は、タンパク質の形成や維持だけでなく、分解にも必要不可欠であるという、一見矛盾する役割を果たしている。Hsp90の正常な機能は健康な細胞の維持に重要であり、その調節不全は発がんに寄与している可能性がある。このシャペロンが26Sプロテアソーム(不要もしくは有害なタンパク質を分解する)を安定化しており、またキナーゼをプロテアソームによる分解から保護し安定化する役割を果たしていることは、その機能的多様性を端的に示している。Hsp90阻害剤はがん治療に用いられ、治療標的としても重要である[64]。
薬剤によるHsp90の標的化は、臨床試験で有望な結果が得られている。一例として、Hsp90阻害剤ゲルダナマイシンは抗腫瘍活性物質として使用されている[28]。この薬剤は当初はキナーゼ阻害剤として機能すると考えられていたが、後にHsp90阻害剤であることが示され、コンパクトなコンフォメーションをとることでATP結合部位に挿入されることが明らかにされている[28]。
Hsp90βは、ヒトの卵巣の機能不全そして不妊をもたらす自己免疫疾患に関与する自己抗原バイオマーカーの1つとして同定されている[65]。抗Hsp90自己抗体を有する不妊女性の血清を用いて、Hsp90βタンパク質の免疫優性エピトープの予測と検証が行われており、10アミノ酸からなるペプチドEP6(380–389)、次いでEP1(1–12)、EP8(488–498)が主要な免疫原性エピトープとなっていることが示されている。自己抗原エピトープに関する知識は、その後の病理を理解する上で重要である。予測立体構造ではこうしたペプチドはループ型のコンフォメーションをとっており、タンパク質の中で最も可動性の高い部分となっている。また、いくつかの生物種のHsp90βの配列解析からは、EP6ペプチドはよく保存されたモチーフの一部であることが明らかにされている。 EP6に対して作製されたポリクローナル抗体は患者の血清の抗Hsp90自己抗体と類似した生化学的・細胞的な免疫反応性を示すことが確認されている[66]。
進化[編集]
Hsp90の全てのホモログは約40%の配列同一性を有することが配列アラインメントによって示されているように、Hsp90は高度に保存されたタンパク質である。Hsp90には細胞質基質型と小胞体型の少なくとも2つのホモログが存在する。こうした2種類のホモログの存在は、真核生物の進化のごく初期に生じた遺伝子重複によるものである可能性が高く、小胞体や核の進化を伴うイベントであった可能性がある。こうした推論は、真核生物の中で主で最も初期に分岐した種の1つであるランブル鞭毛虫Giardia duodenalis(Giardia lamblia)でも重複が生じていることからも裏付けられる。また、菌類や脊椎動物にみられるさまざまな形態のHsp90は、他に少なくとも2度の遺伝子重複が生じたことで説明される。1つ目の分岐は出芽酵母Saccharomyces cerevisiaeに存在する恒常発現型と熱誘導型のHsp90を生み出したものであり、2つ目の遺伝子重複は全ての脊椎動物に存在するα、βサブファミリーを生み出したものである。Hsp90の配列に基づいた系統樹では、植物と動物は菌類よりも互いに近縁な関係にある[67]。Hsp90と同様に、Hsp70をコードする遺伝子も真核細胞形成の極めて初期段階に生じた遺伝子重複によって、細胞質型と小胞体型のホモログが生じている[68]。こうした遺伝子重複は、真核細胞や小胞体の起源において重要なイベントであったと考えられている[69][70]。
出典[編集]
- ^ a b PDB: 2CG9; “Crystal structure of an Hsp90-nucleotide-p23/Sba1 closed chaperone complex”. Nature 440 (7087): 1013–7. (April 2006). doi:10.1038/nature04716. PMC 5703407. PMID 16625188.
- ^ “A molecular clamp in the crystal structure of the N-terminal domain of the yeast Hsp90 chaperone”. Nat. Struct. Biol. 4 (6): 477–82. (June 1997). doi:10.1038/nsb0697-477. PMID 9187656.
- ^ “The 90-kDa molecular chaperone family: structure, function, and clinical applications. A comprehensive review”. Pharmacol. Ther. 79 (2): 129–68. (August 1998). doi:10.1016/S0163-7258(98)00013-8. PMID 9749880.
- ^ “The Drosophila Dpit47 protein is a nuclear Hsp90 co-chaperone that interacts with DNA polymerase alpha”. J. Cell Sci. 114 (Pt 11): 2015–25. (1 June 2001). doi:10.1242/jcs.114.11.2015. PMID 11493638.
- ^ a b c “Comparative genomics and evolution of the HSP90 family of genes across all kingdoms of organisms”. BMC Genomics 7: 156. (2006). doi:10.1186/1471-2164-7-156. PMC 1525184. PMID 16780600.
- ^ “Roles of the Escherichia coli Small Heat Shock Proteins IbpA and IbpB in Thermal Stress Management: Comparison with ClpA, ClpB, and HtpG In Vivo”. J. Bacteriol. 180 (19): 5165–72. (October 1998). doi:10.1128/JB.180.19.5165-5172.1998. PMC 107554. PMID 9748451.
- ^ “The ATPase cycle of Hsp90 drives a molecular 'clamp' via transient dimerization of the N-terminal domains”. EMBO J. 19 (16): 4383–92. (August 2000). doi:10.1093/emboj/19.16.4383. PMC 302038. PMID 10944121.
- ^ a b c “The HSP90 family of genes in the human genome: insights into their divergence and evolution”. Genomics 86 (6): 627–37. (December 2005). doi:10.1016/j.ygeno.2005.08.012. PMID 16269234.
- ^ “The role of Hsp90N, a new member of the Hsp90 family, in signal transduction and neoplastic transformation”. J. Biol. Chem. 277 (10): 8312–20. (March 2002). doi:10.1074/jbc.M109200200. PMID 11751906.
- ^ “Hsp90n - An accidental product of a fortuitous chromosomal translocation rather than a regular Hsp90 family member of human proteome”. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics 1784 (11): 1844–6. (November 2008). doi:10.1016/j.bbapap.2008.06.013. PMID 18638579.
- ^ “Structure and in vivo function of Hsp90”. Curr. Opin. Struct. Biol. 10 (1): 46–51. (February 2000). doi:10.1016/S0959-440X(99)00047-0. PMID 10679459.
- ^ a b c “Structure and functional relationships of Hsp90”. Curr Cancer Drug Targets 3 (5): 301–23. (October 2003). doi:10.2174/1568009033481877. PMID 14529383.
- ^ “Structure, function, and mechanism of the Hsp90 molecular chaperone”. Protein Folding in the Cell. Advances in Protein Chemistry. 59. (2001). 157–86. doi:10.1016/S0065-3233(01)59005-1. ISBN 978-0-12-034259-4. PMID 11868271
- ^ a b “Crystal structure of an Hsp90-geldanamycin complex: targeting of a protein chaperone by an antitumor agent”. Cell 89 (2): 239–50. (April 1997). doi:10.1016/S0092-8674(00)80203-2. PMID 9108479.
- ^ “Identification and structural characterization of the ATP/ADP-binding site in the Hsp90 molecular chaperone”. Cell 90 (1): 65–75. (July 1997). doi:10.1016/S0092-8674(00)80314-1. PMID 9230303.
- ^ “A molecular clamp in the crystal structure of the N-terminal domain of the yeast Hsp90 chaperone”. Nat. Struct. Biol. 4 (6): 477–82. (June 1997). doi:10.1038/nsb0697-477. PMID 9187656.
- ^ a b c “Structural and functional analysis of the middle segment of hsp90: implications for ATP hydrolysis and client protein and cochaperone interactions”. Mol. Cell 11 (3): 647–58. (March 2003). doi:10.1016/S1097-2765(03)00065-0. PMID 12667448.
- ^ “Structural Analysis of E. coli hsp90 reveals dramatic nucleotide-dependent conformational rearrangements”. Cell 127 (2): 329–40. (October 2006). doi:10.1016/j.cell.2006.09.027. PMID 17055434.
- ^ “Crystal structure of an Hsp90-nucleotide-p23/Sba1 closed chaperone complex”. Nature 440 (7087): 1013–7. (April 2006). doi:10.1038/nature04716. PMC 5703407. PMID 16625188.
- ^ “Structures of GRP94-nucleotide complexes reveal mechanistic differences between the hsp90 chaperones”. Mol. Cell 28 (1): 41–56. (October 2007). doi:10.1016/j.molcel.2007.08.024. PMC 2094010. PMID 17936703.
- ^ “The hsp90 chaperone machinery”. J. Biol. Chem. 283 (27): 18473–7. (July 2008). doi:10.1074/jbc.R800007200. PMID 18442971.
- ^ a b “Domain mapping studies reveal that the M domain of hsp90 serves as a molecular scaffold to regulate Akt-dependent phosphorylation of endothelial nitric oxide synthase and NO release”. Circ. Res. 90 (8): 866–73. (May 2002). doi:10.1161/01.RES.0000016837.26733.BE. PMID 11988487.
- ^ a b “Modulation of Akt kinase activity by binding to Hsp90”. Proc. Natl. Acad. Sci. U.S.A. 97 (20): 10832–7. (September 2000). doi:10.1073/pnas.170276797. PMC 27109. PMID 10995457.
- ^ “Activation of the ATPase activity of hsp90 by the stress-regulated cochaperone aha1”. Mol. Cell 10 (6): 1307–18. (December 2002). doi:10.1016/S1097-2765(02)00785-2. PMID 12504007.
- ^ “The heat shock protein 90 antagonist novobiocin interacts with a previously unrecognized ATP-binding domain in the carboxyl terminus of the chaperone”. J. Biol. Chem. 275 (47): 37181–6. (November 2000). doi:10.1074/jbc.M003701200. PMID 10945979.
- ^ “A Nucleotide-dependent molecular switch controls ATP binding at the C-terminal domain of Hsp90. N-terminal nucleotide binding unmasks a C-terminal binding pocket”. J. Biol. Chem. 277 (9): 7066–75. (March 2002). doi:10.1074/jbc.M105568200. PMID 11751878.
- ^ “Specific binding of tetratricopeptide repeat proteins to the C-terminal 12-kDa domain of hsp90”. J. Biol. Chem. 273 (29): 18007–10. (July 1998). doi:10.1074/jbc.273.29.18007. PMID 9660753.
- ^ a b c d “The Hsp90 chaperone complex as a novel target for cancer therapy”. Ann. Oncol. 14 (8): 1169–76. (August 2003). doi:10.1093/annonc/mdg316. PMID 12881371.
- ^ “Hsp90 structure and function studied by NMR spectroscopy”. Biochim. Biophys. Acta 1823 (3): 636–47. (March 2012). doi:10.1016/j.bbamcr.2011.11.009. PMID 22155720.
- ^ a b c Hsp70 and Hsp90--a relay team for protein folding. Reviews of Physiology, Biochemistry and Pharmacology. 151. (2004). 1–44. doi:10.1007/s10254-003-0021-1. ISBN 978-3-540-22096-1. PMID 14740253
- ^ “Heat shock protein-90 inhibitors: a chronicle from geldanamycin to today's agents”. Curr Opin Investig Drugs 7 (6): 534–41. (June 2006). PMID 16784024.
- ^ “Regulation of signaling protein function and trafficking by the hsp90/hsp70-based chaperone machinery”. Exp. Biol. Med. (Maywood) 228 (2): 111–33. (1 February 2003). doi:10.1177/153537020322800201. PMID 12563018.
- ^ “Structural basis for depletion of heat shock protein 90 client proteins by deguelin”. J. Natl. Cancer Inst. 99 (12): 949–61. (June 2007). doi:10.1093/jnci/djm007. PMID 17565155.
- ^ “Derrubone, an inhibitor of the Hsp90 protein folding machinery”. J. Nat. Prod. 70 (12): 2014–8. (December 2007). doi:10.1021/np070190s. PMID 18020309.
- ^ “Molecular characterization of macbecin as an Hsp90 inhibitor”. J. Med. Chem. 51 (9): 2853–7. (May 2008). doi:10.1021/jm701558c. PMID 18357975.
- ^ “Lead identification of β-lactam and related imine inhibitors of the molecular chaperone heat shock protein 90”. Bioorg. Med. Chem. 19 (20): 6055–68. (October 2011). doi:10.1016/j.bmc.2011.08.048. PMID 21920765.
- ^ “The amino-terminal domain of heat shock protein 90 (hsp90) that binds geldanamycin is an ATP/ADP switch domain that regulates hsp90 conformation”. J. Biol. Chem. 272 (38): 23843–50. (September 1997). doi:10.1074/jbc.272.38.23843. PMID 9295332.
- ^ “The crystal structure of the asymmetric GroEL-GroES-(ADP)7 chaperonin complex”. Nature 388 (6644): 741–50. (August 1997). doi:10.1038/41944. PMID 9285585.
- ^ “A model for the mechanism of strand passage by DNA gyrase”. Proc. Natl. Acad. Sci. U.S.A. 96 (15): 8414–9. (July 1999). doi:10.1073/pnas.96.15.8414. PMC 17530. PMID 10411889.
- ^ Buchner J (April 1999). “Hsp90 & Co. - a holding for folding”. Trends Biochem. Sci. 24 (4): 136–41. doi:10.1016/S0968-0004(99)01373-0. PMID 10322418.
- ^ “The 90-kDa heat shock protein, Hsp90, binds and protects casein kinase II from self-aggregation and enhances its kinase activity”. J. Biol. Chem. 267 (10): 7042–7. (April 1992). doi:10.1016/S0021-9258(19)50533-6. PMID 1551911.
- ^ “Hsp90 chaperones protein folding in vitro”. Nature 358 (6382): 169–70. (July 1992). doi:10.1038/358169a0. PMID 1614549.
- ^ “Transient interaction of Hsp90 with early unfolding intermediates of citrate synthase. Implications for heat shock in vivo”. J. Biol. Chem. 270 (13): 7288–94. (March 1995). doi:10.1074/jbc.270.13.7288. PMID 7706269.
- ^ Picard D (October 2002). “Heat-shock protein 90, a chaperone for folding and regulation”. Cell. Mol. Life Sci. 59 (10): 1640–8. doi:10.1007/PL00012491. PMID 12475174.
- ^ a b “The molecular chaperone Hsp90 plays a role in the assembly and maintenance of the 26S proteasome”. EMBO J. 22 (14): 3557–67. (July 2003). doi:10.1093/emboj/cdg349. PMC 165619. PMID 12853471.
- ^ “Cytochrome P450 ubiquitination: branding for the proteolytic slaughter?”. Annu. Rev. Pharmacol. Toxicol. 45: 439–64. (2005). doi:10.1146/annurev.pharmtox.45.120403.100127. PMID 15822184.
- ^ “Temperature-sensitive mutants of hsp82 of the budding yeast Saccharomyces cerevisiae”. Mol. Gen. Genet. 242 (5): 517–27. (March 1994). doi:10.1007/BF00285275. PMID 8121410.
- ^ “Structure and function of molecular chaperone HSP90”. Sowriemiennyj Naucznyj Wiestnik Ser. Biologija Chimija 15 (23): 46–65. (2007).
- ^ “Chaperoning of glucocorticoid receptors”. Molecular Chaperones in Health and Disease. Handbook of Experimental Pharmacology. 172. (2006). pp. 111–38. doi:10.1007/3-540-29717-0_5. ISBN 978-3-540-25875-9. PMID 16610357
- ^ “The glucocorticoid responses are shaped by molecular chaperones”. Mol. Cell. Endocrinol. 275 (1–2): 2–12. (September 2007). doi:10.1016/j.mce.2007.05.018. PMID 17628337.
- ^ “Role of molecular chaperones in steroid receptor action”. Essays Biochem. 40: 41–58. (2004). doi:10.1042/bse0400041. PMID 15242338.
- ^ “Mineralocorticosteroid receptor of the chick intestine. Oligomeric structure and transformation”. J. Biol. Chem. 264 (16): 9304–9. (June 1989). doi:10.1016/S0021-9258(18)60531-9. PMID 2542305.
- ^ “Common non-hormone binding component in non-transformed chick oviduct receptors of four steroid hormones”. Nature 308 (5962): 850–3. (1984). doi:10.1038/308850a0. PMID 6201744.
- ^ “Subunit composition of the molybdate-stabilized "8-9 S" nontransformed estradiol receptor purified from calf uterus”. J. Biol. Chem. 262 (15): 6969–75. (May 1987). doi:10.1016/S0021-9258(18)48188-4. PMID 3584104.
- ^ “The common 90-kd protein component of non-transformed '8S' steroid receptors is a heat-shock protein”. EMBO J. 4 (12): 3131–5. (December 1985). doi:10.1002/j.1460-2075.1985.tb04055.x. PMC 554632. PMID 2419124.
- ^ “EGFR Signaling and Drug Discovery”. Oncology 77 (6): 400–410. (2009). doi:10.1159/000279388. PMID 20130423.
- ^ “Inhibition of Hsp90 down-regulates mutant epidermal growth factor receptor (EGFR) expression and sensitizes EGFR mutant tumors to paclitaxel”. Cancer Res. 68 (2): 589–96. (January 2008). doi:10.1158/0008-5472.CAN-07-1570. PMC 4011195. PMID 18199556.
- ^ “Targeting senescent cells in translational medicine”. EMBO Molecular Medicine 11 (12): e10234. (2019). doi:10.15252/emmm.201810234. PMC 6895604. PMID 31746100.
- ^ “Neoadjuvant trastuzumab induces apoptosis in primary breast cancers”. J. Clin. Oncol. 23 (11): 2460–8. (April 2005). doi:10.1200/JCO.2005.00.661. PMID 15710948.
- ^ “Nanoengineered Disruption of Heat Shock Protein 90 Targets Drug-Induced Resistance and Relieves Natural Killer Cell Suppression in Breast Cancer”. Cancer Research 80 (23): 5355–5366. (December 2020). doi:10.1158/0008-5472.CAN-19-4036. PMC 7718318. PMID 33077554.
- ^ a b “Heat shock proteins in cancer: chaperones of tumorigenesis”. Trends in Biochemical Sciences 31 (3): 164–72. (March 2006). doi:10.1016/j.tibs.2006.01.006. PMID 16483782.
- ^ “Functional proteomic screens reveal an essential extracellular role for hsp90 alpha in cancer cell invasiveness”. Nat. Cell Biol. 6 (6): 507–14. (June 2004). doi:10.1038/ncb1131. PMID 15146192.
- ^ “Hsp90 and the chaperoning of cancer”. Nat. Rev. Cancer 5 (10): 761–72. (October 2005). doi:10.1038/nrc1716. PMID 16175177.
- ^ “Update on Hsp90 inhibitors in clinical trial”. Curr Top Med Chem 9 (15): 1479–92. (2009). doi:10.2174/156802609789895728. PMC 7241864. PMID 19860730.
- ^ “A block in the road to fertility: autoantibodies to heat-shock protein 90-beta in human ovarian autoimmunity”. Fertil Steril 92 (4): 1395–1409. (2009). doi:10.1016/j.fertnstert.2008.08.068. PMID 19022436.
- ^ “Anti-HSP90 autoantibodies in sera of infertile women identify a dominant, conserved epitope EP6 (380-389) of HSP90 beta protein”. Reprod Biol Endocrinol 9 (16): 13. (2011). doi:10.1186/1477-7827-9-16. PMC 3039567. PMID 21272367.
- ^ Gupta RS (November 1995). “Phylogenetic analysis of the 90 kD heat shock family of protein sequences and an examination of the relationship among animals, plants, and fungi species”. Mol. Biol. Evol. 12 (6): 1063–73. doi:10.1093/oxfordjournals.molbev.a040281. PMID 8524040.
- ^ “Cloning of Giardia lamblia heat shock protein HSP70 homologs: implications regarding origin of eukaryotic cells and of endoplasmic reticulum”. Proc. Natl. Acad. Sci. U.S.A. 91 (8): 2895–9. (April 1994). doi:10.1073/pnas.91.8.2895. PMC 43480. PMID 8159675.
- ^ “The origin of the eukaryotic cell”. Trends Biochem. Sci. 21 (5): 166–71. (May 1996). doi:10.1016/S0968-0004(96)20013-1. PMID 8871398.
- ^ Gupta RS (December 1998). “Protein phylogenies and signature sequences: A reappraisal of evolutionary relationships among archaebacteria, eubacteria, and eukaryotes”. Microbiol. Mol. Biol. Rev. 62 (4): 1435–91. doi:10.1128/MMBR.62.4.1435-1491.1998. PMC 98952. PMID 9841678.
関連項目[編集]
外部リンク[編集]
- Hsp90 Heat-Shock Proteins - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス(英語)
- Didier Picard. “Hsp90 web site”. VisibilityWeb. 2004年11月23日時点のオリジナルよりアーカイブ。2008年7月7日閲覧。 “A web site for the community of scientists interested in the Hsp90 molecular chaperone machine.”
