Damメチラーゼ
| Site-specific DNA-methyltransferase (adenine-specific) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 識別子 | |||||||||
| EC番号 | 2.1.1.72 | ||||||||
| CAS登録番号 | 69553-52-2 | ||||||||
| データベース | |||||||||
| IntEnz | IntEnz view | ||||||||
| BRENDA | BRENDA entry | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | KEGG entry | ||||||||
| MetaCyc | metabolic pathway | ||||||||
| PRIAM | profile | ||||||||
| PDB構造 | RCSB PDB PDBj PDBe PDBsum | ||||||||
| |||||||||
Damメチラーゼ(英: Dam methylase)またはDamメチル化酵素は、5'-GATC-3'のDNA配列中のアデニン塩基にメチル基を付加する、DNAメチル化酵素の一種である(EC 2.1.1.72)[1][2]。
大腸菌K-12株から見いだされ、元々はDNAアデニンメチル化(DNA adenine methylation; Dam)を担う遺伝子だということからDam遺伝子と名付けられた[3][4]。ただしその後、DNAアデニントランスフェラーゼ(英: DNA adenine methyltransferase)の頭文字としてDamの名称が利用される論文も出版され[5]、現在ではこちらの説明が論文等でよく記述されている[6]。
アデニンをメチル化する数多くのメチル化酵素としては、Dam以外にも様々な原核生物やバクテリオファージから発見されている[7]。
Damメチラーゼは制限修飾系の一部を構成しないオーファンメチルトランスフェラーゼ(orphan methyltransferase)であり、遺伝子発現を調節する[8][8][9][10][11]。この酵素は次の化学反応を触媒する。
- S-アデノシル-L-メチオニン + DNA アデニン S-アデノシル-L-ホモシステイン + DNA 6-メチルアミノプリン
大腸菌由来のDamメチラーゼは、クロマチンプロファイリング技術であるDamIDで広く利用されている。Damを目的のDNA結合タンパク質と融合させてモデル生物でトランスジーンとして発現させることで、目的タンパク質の結合部位を同定することができる[12]。
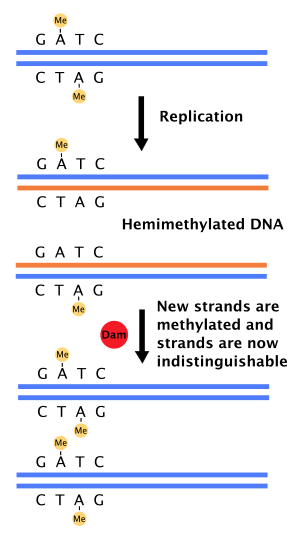
DNAミスマッチ修復における役割[編集]
DNA合成時のDNAポリメラーゼのエラーによってミスマッチ塩基対や小さな挿入・欠失が生じた際には、細胞はミスマッチ修復と呼ばれる経路でDNA修復を行う。この際に、細胞は鋳型鎖と新生鎖を見分けなければならない。一部の細菌ではDNAはDamメチラーゼによってメチル化されているため、複製直後はDNAはヘミメチル化(半メチル化)状態となっている[13]。修復酵素MutSはDNAのミスマッチ部位に結合してMutLをリクルートし、その後MutLはエンドヌクレアーゼMutHを活性化する。MutHはヘミメチル化GATC部位に結合してメチル化されていない娘鎖を選択的に切断し、ヘリカーゼとエキソヌクレアーゼによるミスマッチ周辺領域の除去を可能にする[13][14]。除去部はDNAポリメラーゼIIIによって再合成される。
複製の調節における役割[編集]
細菌細胞での複製起点(oriC)の発火は、DNA複製が各細胞分裂の間に1回だけ行われるよう保証する機構によって高度に制御されている。その機構の一部は、oriCの反復配列に結合して複製を開始するタンパク質であるDnaAのゆっくりとしたATPの加水分解によって説明される。大腸菌のoriCには5'-GATC-3'配列が含まれるため、Damメチラーゼもこの過程に関与している。DNA複製の直後、oriCはヘミメチル化状態となり、しばらくの間隔離される。再びDNA複製が起こるためには、oriCはこの過程を経た後にDamメチラーゼによって完全にメチル化されなければならない[15]。
遺伝子発現の調節における役割[編集]
DamはRNAの転写の促進と抑制にも関与している。大腸菌では、下流のGATC配列がメチル化されている場合、転写は促進される。例えば、尿路病原性大腸菌のP線毛(pyelonephritis-associated pili、PAP線毛、腎盂腎炎関連線毛)の相変異は、PAPプロモーターの近位と遠位の2か所のGATC部位のDamによるメチル化によって制御されている[16]。
大腸菌ではDamメチラーゼはタンパク質の調節の役割を果たしていており、Damメチラーゼの遺伝子は非必須遺伝子で、遺伝子をノックアウトしても細菌は生存する[17]。dam遺伝子をノックアウトしても生存が維持されることは、サルモネラやAggregatibacter actinomycetemcomitansでも観察されている[18][19]。一方でコレラ菌Vibrio choleraeや仮性結核菌Yersinia pseudotuberculosisなどの生物では、dam遺伝子は生存に必須である[20]。Aggregatibacter actinomycetemcomitansでのdam遺伝子のノックアウトはタンパク質ロイコトキシン(leukotoxin)レベルの調節異常と、口腔上皮細胞への侵入能力の低下をもたらす[19]。さらに、歯周病原因菌であるStreptococcus mutansでのDamメチラーゼ欠損の研究からは、齲蝕原性を有するものを含む、103の遺伝子に調節異常がみられることが明らかにされた[20]。
構造的特徴[編集]
メチルトランスフェラーゼまたはメチラーゼは、9つのモチーフと標的認識ドメイン(TRD)の並び順に基づいて3つのグループ(α、β、γ)へと分類される[21]。モチーフIはGly-X-Glyトリペプチドを含むためGループとも呼ばれ、補因子S-アデノシルメチオニン(SAM)の結合に関与することが示唆されている[22]。モチーフIIはN4、N6-アデニンメチルトランスフェラーゼで高度に保存されており、β2ストランドの最後の残基は負に帯電したアミノ酸で疎水的側鎖がそれに続き、SAMに結合する[21]。モチーフIIIもSAMの結合への関与が示唆されている。モチーフIVは特に重要であり、メチラーゼの特徴付けに関して良く知られている。2つ並んだプロリンを含み、N6-アデニンメチルトランスフェラーゼではDPPYモチーフとして高度に保存されているが、N4-アデニン、C5-シトシンメチルトランスフェラーゼでは変化がある。DPPYモチーフはSAMの結合に必要不可欠であることが知られている[23]。モチーフIVからVIIIは触媒活性に関与するが、モチーフIからIII、Xは補因子の結合に関与する。N6-アデニンメチルトランスフェラーゼの場合、これらのモチーフの並び順はN末端 - X - I - II - III - TRD - IV - V - VI - VII - VIII - C末端であり、大腸菌のDamメチラーゼもこの順序に従う[21]。2015年の結晶学的実験からは大腸菌のDamメチラーゼが非GATC配列にも結合することが示され、メチル化非依存的に転写リプレッサーとして機能している可能性が示唆されている[24]。
オーファンメチルトランスフェラーゼ[編集]
Damメチラーゼはオーファンメチルトランスフェラーゼであり制限修飾系の一部を構成しないが、独立して遺伝子発現、ミスマッチ修復、細菌の複製など多くの機能を調節する。これはオーファンメチルトランスフェラーゼの唯一の例ではなく、Caulobacter crescentusや他の関連生物種において5'-GANTC-3'ヘミメチル化DNAをメチル化することで生活環を制御するCcrMなどが存在する[25]。
細菌に存在するものとは別に、ファージにもオーファンメチルトランスフェラーゼは存在し、例えば大腸菌に感染するT2やT4、他のT偶数型バクテリオファージのものが挙げられる[8]。大腸菌とT4のDamメチラーゼには塩基配列の相同性は全くみられないにもかかわらず、アミノ酸配列では11-33残基長の4つの領域で最大64%の同一性がみられ、細菌とファージのメチラーゼ遺伝子は共通の進化的祖先を持つことが示唆されている[26]。T2とT4のメチラーゼは5-ヒドロキシメチルシトシンをメチル化するだけでなく、非典型的DNA部位のメチル化も行う点で、Damメチラーゼとは異なる特徴を持つ。このようなファージが持つオーファンメチルトランスフェラーゼは、in vitroによる特性解析が広く行われているが、生物学的意義については未だ明らかになっていない[8]。
出典[編集]
- ^ “Isolation of deoxyribonucleic acid methylase mutants of Escherichia coli K-12”. Journal of Bacteriology 114 (3): 1143-1150. (June 1973). doi:10.1128/JB.114.3.1143-1150.1973. PMC 285375. PMID 4576399.
- ^ “Recognition sequence of the dam methylase of Escherichia coli K12 and mode of cleavage of Dpn I endonuclease”. The Journal of Biological Chemistry 254 (4): 1408-1413. (February 1979). PMID 368070.
- ^ Marinus, M. G.; Morris, N. Ronald (1973-06). “Isolation of Deoxyribonucleic Acid Methylase Mutants of Escherichia coli K-12” (英語). Journal of Bacteriology 114 (3): 1143-1150. doi:10.1128/jb.114.3.1143-1150.1973. ISSN 0021-9193.
- ^ Marinus, M. G. (1973-03). “Location of DNA methylation genes on the Escherichia coli K-12 genetic map”. Molecular and General Genetics MGG 127 (1): 47-55. doi:10.1007/bf00267782. ISSN 0026-8925.
- ^ Capaldo, Florence N.; Barbour, Stephen D. (1975-01). “DNA content, synthesis and integrity in dividing and non-dividing cells of rec− strains of Escherichia coli K12” (英語). Journal of Molecular Biology 91 (1): 53-66. doi:10.1016/0022-2836(75)90371-X.
- ^ Marinus, M. G.; Løbner-Olesen, A. (2009-02-13). Lovett, Susan T.. ed. “DNA Methylation” (英語). EcoSal Plus 3 (2): ecosalplus.4.4.5. doi:10.1128/ecosalplus.4.4.5. ISSN 2324-6200. PMC 4231299. PMID 26442938.
- ^ “The Restriction Enzyme Database”. REBASE. 2020年2月22日閲覧。
- ^ a b c d “Bacteriophage orphan DNA methyltransferases: insights from their bacterial origin, function, and occurrence”. Applied and Environmental Microbiology 79 (24): 7547-7555. (December 2013). doi:10.1128/aem.02229-13. PMC 3837797. PMID 24123737.
- ^ “Specificity of restriction endonucleases and DNA modification methyltransferases a review (Edition 3)”. Gene 92 (1-2): 1-240. (August 1990). doi:10.1016/0378-1119(90)90486-B. PMID 2172084.
- ^ “Restriction enzymes and their isoschizomers”. Nucleic Acids Research 18 Suppl: 2331-2365. (April 1990). doi:10.1093/nar/18.suppl.2331. PMC 331877. PMID 2159140.
- ^ “Structure and mechanism of multifunctional restriction endonucleases”. Annual Review of Biochemistry 50: 285-319. (1981). doi:10.1146/annurev.bi.50.070181.001441. PMID 6267988.
- ^ “Dam it's good! DamID profiling of protein-DNA interactions”. Wiley Interdisciplinary Reviews. Developmental Biology 5 (1): 25-37. (January 2016). doi:10.1002/wdev.205. PMC 4737221. PMID 26383089.
- ^ a b “The great GATC: DNA methylation in E. coli”. Trends in Genetics 5 (5): 139-143. (1989). doi:10.1016/0168-9525(89)90054-1. PMID 2667217.
- ^ “Dam methylation: coordinating cellular processes”. Current Opinion in Microbiology 8 (2): 154-160. (April 2005). doi:10.1016/j.mib.2005.02.009. PMID 15802246.
- ^ Marinus, Martin G.; Casadesus, Josep (2009-05). “Roles of DNA adenine methylation in host-pathogen interactions: mismatch repair, transcriptional regulation, and more”. FEMS microbiology reviews 33 (3): 488-503. doi:10.1111/j.1574-6976.2008.00159.x. ISSN 1574-6976. PMC 2941194. PMID 19175412.
- ^ “Epigenetic gene regulation in the bacterial world”. Microbiology and Molecular Biology Reviews 70 (3): 830-856. (September 2006). doi:10.1128/MMBR.00016-06. PMC 1594586. PMID 16959970.
- ^ “Characterization of DNA adenine methylation mutants of Escherichia coli K12”. Mutation Research 59 (2): 157-165. (February 1979). doi:10.1016/0027-5107(79)90153-2. PMID 375073.
- ^ “DNA methylation-dependent regulation of pef expression in Salmonella typhimurium”. Molecular Microbiology 35 (4): 728-742. (February 2000). doi:10.1046/j.1365-2958.2000.01743.x. PMID 10692151.
- ^ a b “Inactivation of DNA adenine methyltransferase alters virulence factors in Actinobacillus actinomycetemcomitans”. Oral Microbiology and Immunology 21 (4): 238-244. (August 2006). doi:10.1111/j.1399-302x.2006.00284.x. PMID 16842508.
- ^ a b “DNA adenine methylase is essential for viability and plays a role in the pathogenesis of Yersinia pseudotuberculosis and Vibrio cholerae”. Infection and Immunity 69 (12): 7610-7615. (December 2001). doi:10.1128/iai.69.12.7610-7615.2001. PMC 98854. PMID 11705940.
- ^ a b c “Structure-guided analysis reveals nine sequence motifs conserved among DNA amino-methyltransferases, and suggests a catalytic mechanism for these enzymes”. Journal of Molecular Biology 253 (4): 618-632. (November 1995). doi:10.1006/jmbi.1995.0577. PMID 7473738.
- ^ “Universal catalytic domain structure of AdoMet-dependent methyltransferases”. Journal of Molecular Biology 247 (1): 16-20. (March 1995). doi:10.1006/jmbi.1994.0117. PMID 7897657.
- ^ “Conserved sequence motif DPPY in region IV of the phage T4 Dam DNA-[N-adenine-methyltransferase is important for S-adenosyl-L-methionine binding”]. Nucleic Acids Research 21 (15): 3563-3566. (July 1993). doi:10.1093/nar/21.15.3563. PMC 331459. PMID 16617501.
- ^ “Structures of Escherichia coli DNA adenine methyltransferase (Dam) in complex with a non-GATC sequence: potential implications for methylation-independent transcriptional repression”. Nucleic Acids Research 43 (8): 4296-4308. (April 2015). doi:10.1093/nar/gkv251. PMC 4417163. PMID 25845600.
- ^ “A Caulobacter DNA methyltransferase that functions only in the predivisional cell”. Journal of Molecular Biology 235 (2): 472-485. (January 1994). doi:10.1006/jmbi.1994.1007. PMID 8289276.
- ^ “Common evolutionary origin of the phage T4 dam and host Escherichia coli dam DNA-adenine methyltransferase genes”. Journal of Bacteriology 164 (2): 932-937. (November 1985). doi:10.1128/JB.164.2.932-937.1985. PMC 214344. PMID 3902803.

