「RNA干渉」の版間の差分
-改名提案、ソートキー・冒頭表記修正 |
4th protocol (会話 | 投稿記録) タグ: 2017年版ソースエディター |
||
| (他の1人の利用者による、間の3版が非表示) | |||
| 1行目: | 1行目: | ||
[[File:ShRNA Lentivirus.svg|thumb|upright=1.5|right|{{仮リンク|レンチウイルス属|en|Lentivirus|label=レンチウイルス}}による哺乳類細胞へのshRNAのデリバリーとRNAi機構]] |
|||
[[ファイル:RNAi.jpg|thumb|right|350px|RNAi]] |
|||
'''RNA干渉'''(RNAかんしょう、{{Lang-en-short|RNA interference}}、'''RNAi''')は、二本鎖[[リボ核酸|RNA]](dsRNA)が[[翻訳 (生物学)|翻訳]]抑制または[[転写 (生物学)|転写]]抑制によって[[遺伝子発現|遺伝子の発現]]を配列特異的に抑制する生物学的過程である。RNAiは歴史的には、"co-suppression"、"post-transcriptional gene silencing"(PTGS)、"quelling"といった名称で知られていた。これらの過程は見かけ上異なるものの、それぞれに対して詳細な研究が行われ、これらの実体はすべてRNAiであることが明らかにされた。[[アンドリュー・ファイアー]]と[[クレイグ・メロー]]は、1998年に発表された線虫''[[Caenorhabditis elegans]]''におけるRNAiに関する業績によって、2006年に[[ノーベル生理学・医学賞]]を受賞した。RNAiとその調節の可能性が発見されて以降、RNAiが目的の遺伝子を抑制する多大な可能性を有していることが明らかとなった。現在ではRNAiは、遺伝子抑制を目的とした[[アンチセンスRNA|アンチセンス]]治療よりも正確かつ効率的で安定なより良い治療法であることが知られている<ref name="Saurabh Satyajit, Vidyarthi AS, Prasad D 2014 543–564">{{cite journal|date=March 2014|title=RNA interference: concept to reality in crop improvement|journal=Planta|volume=239|issue=3|pages=543–64|doi=10.1007/s00425-013-2019-5|pmid=24402564|vauthors=Saurabh S, Vidyarthi AS, Prasad D|doi-access=free}}</ref>。 |
|||
'''RNA干渉'''('''RNAかんしょう'''、'''RNA interference'''、'''RNAi''')は、二本鎖[[リボ核酸|RNA]]と相補的な[[塩基配列]]を持つ[[mRNA]]が分解される現象。[[RNAi法]]は、この現象を利用して人工的に二本鎖RNAを導入することにより、任意の[[遺伝子]]の発現を抑制する手法<ref name = "RNAi2008"> |
|||
{{cite journal |
|||
| author = Mahmood-ur-Rahman, Ali I, Husnain T, Riazuddin S. |
|||
| title = RNA interference: the story of gene silencing in plants and humans |
|||
| journal = Biotechnol Adv. |
|||
| year = 2008 |
|||
| volume = 26 |
|||
| issue = 3 |
|||
| pages = 202-209 |
|||
| doi = 10.1016/j.biotechadv.2007.12.002 |
|||
}}</ref>。[[アンチセンスRNA法]]や[[コサプレッション]]もRNAiの一形態と考えられる。 |
|||
RNAiでは、[[miRNA]]と[[siRNA]]という2つのタイプの低分子RNAが中心的役割を果たす。RNAは遺伝子の直接産物であり、こうした低分子RNAは酵素複合体を指揮し、[[伝令RNA|mRNA]]の分解や翻訳の阻害といった、転写後段階での遺伝子サイレンシングを介して標的遺伝子の活性を低下させる。さらに、siRNAやmiRNAに相補的なゲノム領域でDNAのメチル化を触媒する酵素複合体による、転写前段階でのサイレンシング機構も存在し、それによって転写が阻害されることもある。RNAiは、[[ウイルス]]や[[トランスポゾン]]といった寄生性のヌクレオチド配列に対する細胞の防御に重要な役割を果たす。また、発生にも影響を与える。 |
|||
通常、遺伝子の機能阻害は[[染色体]]上の遺伝子を破壊することで行われてきた。しかし、RNAi法はこのような煩雑な操作は必要なく、塩基配列さえ知ることができれば合成したRNAを導入するなどの簡便な手法で遺伝子の機能を調べることができる。[[ゲノムプロジェクト]]によって全塩基配列を知ることのできる[[生物]]種では、[[逆遺伝学]]的解析の速度を上げる大きな要因の一つともなった。一方、完全な機能喪失とはならないこと、非特異的な影響を考慮する必要があることなどの問題もある。 |
|||
RNAi経路は[[動物]]を含む多くの[[真核生物]]でみられ、[[Dicer]]によって開始される。Dicerは長いdsRNA分子を約21[[ヌクレオチド]]からなる短いsiRNA二本鎖断片へと切断する酵素である。その後、siRNAは2つの一本鎖RNA(ssRNA)、すなわちパッセンジャー鎖とガイド鎖へと巻き戻される。パッセンジャー鎖は分解され、ガイド鎖は[[RNA誘導サイレンシング複合体]](RISC)へと取り込まれる。RNAi経路の中で最もよく研究されているのは転写後段階での遺伝子サイレンシングであり、この過程ではガイド鎖がmRNA分子中の相補的な配列と対合し、RISCの触媒要素である[[アルゴノート (タンパク質)|Argonaute]]2(Ago2)による切断が誘導される。一部の生物種では、当初のsiRNA濃度が限定的なものであっても、この過程が増幅し、全身に拡散する。 |
|||
[[1998年]]に[[アンドリュー・ファイアー]]等は[[センチュウ]]の一種である[[モデル生物]]の''Caenorhabditis elegans'' (''[[C. elegans]]'')を用いて、センス鎖とアンチセンス鎖の混合RNAが、それぞれの単独RNAより大きな阻害効果があることを示した<ref>{{cite journal|author=Fire A, Xu S, Montgomery M, Kostas S, Driver S, Mello C|year=1998|title=Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans|journal=Nature|volume=391|issue=6669|pages=806–811|doi=10.1038/35888}}</ref>。この効果は、標的mRNAとの[[モル]]比などから単純にアンチセンス鎖がmRNAに1:1で張り付いて阻害するのではなく、何らかの増幅過程を含むか、[[酵素]]的活性をもつことが予想された。その後、RNase IIIの一種である[[Dicer]]によって、長い二本鎖RNAが、[[siRNA]](small interfering RNA)と呼ばれる21-23 ntの短い3'突出型二本鎖RNAに切断されること、siRNAといくつかの蛋白質から成るRNA蛋白質複合体であるRISC複合体が再利用されながら相補的な配列を持つmRNAを分解することがわかってきた。 |
|||
細胞に導入された合成dsRNAは目的の遺伝子の選択的かつ強固な抑制を誘導することとができるため、RNAiは培養細胞においても個体レベルにおいても有益な研究ツールである。RNAiは細胞内の各遺伝子を体系的にオフにする大規模スクリーニングに利用することができ、[[細胞分裂]]などのイベントや特定の細胞過程に必要な要素の同定のために活用することができる。また、この経路は[[バイオテクノロジー]]や[[医療]]、そして[[殺虫剤]]など実用的なツールとしても利用されている<ref name="s1308">{{cite journal|date=August 2013|title=A lethal dose of RNA|journal=Science|volume=341|issue=6147|pages=732–3|bibcode=2013Sci...341..732K|doi=10.1126/science.341.6147.732|pmid=23950525|vauthors=Kupferschmidt K}}</ref>。 |
|||
[[2001年]]には[[哺乳類]]の[[細胞]]でsiRNAを導入することで、それまで問題となってきた二本鎖RNA依存性[[プロテインキナーゼ]]の反応を回避することができた<ref name = "RNAi2001"> |
|||
{{cite journal |
|||
| author = Elbashir SM, Harborth J, Lendeckel W, Yalcin A, Weber K, Tuschl T. |
|||
| title = Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells. |
|||
| journal = Nature |
|||
| year = 2001 |
|||
| volume = 411 |
|||
| issue = 6836 |
|||
| pages = 494-498 |
|||
| doi = 10.1038/35078107 |
|||
}}</ref>。これにより、遺伝子治療応用への期待が高まっている。RNAi機構は[[酵母]]から[[ヒト]]に至るまで多くの生物種で保存されている。その生物学的な意義としては[[ウイルス]]などに対する防御機構として[[進化]]してきたという仮説が提唱されている。さらに、染色体再構成などにも関わる可能性が示され、またstRNAなど作用機構の一部を共有する[[miRNA]]が[[発生 (生物学)|発生]]過程の遺伝子発現制御を行っていることなどが明らかとなり、小分子RNAが果たす機能に注目が集まるきっかけの一つとなった。また、酵母を用いた研究では、[[染色体]]の[[セントロメア]]や[[テロメア]]の[[ヘテロクロマチン]]形成にRNAiの機構が関与していることが報告されている。 |
|||
== 細胞機構 == |
|||
[[2006年]]、[[アンドリュー・ファイアー]]と[[クレイグ・メロー]]はRNAi発見の功績より[[ノーベル生理学・医学賞]]を受賞した。 |
|||
[[File:2ffl-by-domain.png|thumb|right|[[ジアルジア]]''Giardia intestinalis''由来Dicerタンパク質。dsRNAからsiRNAへの切断を触媒する。RNaseドメインは緑色、PAZドメインは黄色、プラットホームドメインは赤色、コネクターヘリックスは青色で示されている<ref name="Macrae">{{cite journal|date=January 2006|title=Structural basis for double-stranded RNA processing by Dicer|url=https://semanticscholar.org/paper/2f3cda257860e2a220f5ba223ab51618605f22e2|journal=Science|volume=311|issue=5758|pages=195–8|bibcode=2006Sci...311..195M|doi=10.1126/science.1121638|pmid=16410517|vauthors=Macrae IJ, Zhou K, Li F, Repic A, Brooks AN, Cande WZ, Adams PD, Doudna JA|s2cid=23785494}}</ref>。]] |
|||
[[2019年]]、[[アンドリュー・ファイアー]]と[[クレイグ・メロー]]を共同設立者として2002年に設立された[[Alnylam|アルナイラム]]社が開発した遺伝性難病[[トランスサイレチン型家族性アミロイドポリニューロパチー]]の治療薬として[[Patisiran sodium|オンパットロ]]が発売された。<ref> {{Cite press release |
|||
| title = 核酸医薬のアルナイラム「オンパットロ」で日本市場に参入―2品目目 来年にも申請、事業加速へ |
|||
| publisher = AnswersNews |
|||
| date = 2019/09/19 |
|||
| url = https://answers.ten-navi.com/pharmanews/16924/ |
|||
| accessdate = }} </ref> |
|||
RNAiは、RNA誘導サイレンシング複合体(RISC)によって制御されるRNA依存的遺伝子サイレンシング過程であり、[[細胞質]]に存在する短い二本鎖RNA(dsRNA)分子によって開始される。dsRNAが外因性のもの(RNAゲノムを持つ[[ウイルス]]の感染や実験室的操作に由来するもの)である場合には、RNAは直接細胞質に取り込まれ、Dicerによって短い断片へと切断される。ゲノム中のRNAコーディング遺伝子から発現したpre-miRNAなどのように、RNAi経路を開始するdsRNAは内因性のもの(細胞に由来するもの)である場合もある。こうした遺伝子に由来する[[一次転写産物]]は、まず[[細胞核|核]]内でpre-miRNAに特徴的な[[ステムループ]]構造を形成するようプロセシングされる。その後、外因性と内因性の2つのdsRNA経路はRISCへと集約される<ref name="pmid15614608">{{cite journal|date=August 2004|title=RNA interference: the molecular immune system|journal=Journal of Molecular Histology|volume=35|issue=6|pages=545–53|doi=10.1007/s10735-004-2192-8|pmid=15614608|vauthors=Bagasra O, Prilliman KR|s2cid=2966105|citeseerx=10.1.1.456.1701}}</ref>。 |
|||
トランスサイレチン型家族性アミロイドポリニューロパチーの根治的治療法は[[肝移植]]しか無かったがオンパットロ及びトランスサイレチン4量体安定化薬の[[Tafamidis meglumine|ビンダケル]]の発売により治療選択肢が拡大した。 |
|||
外因性のdsRNAは[[リボヌクレアーゼ]]タンパク質であるDicerによってRNAiを開始する<ref name="Bernstein">{{cite journal|date=January 2001|title=Role for a bidentate ribonuclease in the initiation step of RNA interference|journal=Nature|volume=409|issue=6818|pages=363–6|bibcode=2001Natur.409..363B|doi=10.1038/35053110|pmid=11201747|vauthors=Bernstein E, Caudy AA, Hammond SM, Hannon GJ|s2cid=4371481}} {{closed access}}</ref>。Dicerは植物ではdsRNA、ヒトでは[[shRNA]]と結合して切断を行い、3'末端に2ヌクレオチドの突出部を持つ20–25塩基対の二本鎖断片を形成する<ref name=":2">{{cite journal|date=January 2009|title=On the road to reading the RNA-interference code|journal=Nature|volume=457|issue=7228|pages=396–404|bibcode=2009Natur.457..396S|doi=10.1038/nature07754|pmid=19158785|vauthors=Siomi H, Siomi MC|s2cid=205215974}}<br />{{cite journal|date=March 2000|title=RNAi: double-stranded RNA directs the ATP-dependent cleavage of mRNA at 21 to 23 nucleotide intervals|journal=Cell|volume=101|issue=1|pages=25–33|doi=10.1016/S0092-8674(00)80620-0|pmid=10778853|vauthors=Zamore PD, Tuschl T, Sharp PA, Bartel DP|doi-access=free}}<br />{{cite journal|date=May 2005|title=The contributions of dsRNA structure to Dicer specificity and efficiency|journal=RNA|volume=11|issue=5|pages=674–82|doi=10.1261/rna.7272305|pmid=15811921|pmc=1370754|vauthors=Vermeulen A, Behlen L, Reynolds A, Wolfson A, Marshall WS, Karpilow J, Khvorova A}}<br />{{cite journal|date=January 2009|title=The promises and pitfalls of RNA-interference-based therapeutics|journal=Nature|volume=457|issue=7228|pages=426–33|bibcode=2009Natur.457..426C|doi=10.1038/nature07758|pmid=19158789|pmc=2702667|vauthors=Castanotto D, Rossi JJ}}</ref>。この長さは、標的遺伝子に対する特異性を最大化し、かつ非特異的な効果を最小化することが複数の生物種のゲノムに対する[[バイオインフォマティクス]]研究から示唆されている<ref name="Qiu">{{cite journal|year=2005|title=A computational study of off-target effects of RNA interference|journal=Nucleic Acids Research|volume=33|issue=6|pages=1834–47|doi=10.1093/nar/gki324|pmid=15800213|pmc=1072799|vauthors=Qiu S, Adema CM, Lane T}}</ref>。こうした短い二本鎖断片はsiRNAと呼ばれる。その後、RISCローディング複合体(RLC)によって、siRNAは一本鎖へと分離されて活性型のRISCへ取り込まれる。[[ショウジョウバエ]]のRLCはDcr-2(Dicer2)とR2D2を含み、Ago2とRISCの一体化のために重要である<ref name="Nakanishi">{{cite journal|date=September 2016|title=Anatomy of RISC: how do small RNAs and chaperones activate Argonaute proteins?|journal=Wiley Interdisciplinary Reviews: RNA|volume=7|issue=5|pages=637–60|doi=10.1002/wrna.1356|pmid=27184117|pmc=5084781|vauthors=Nakanishi K}}</ref>。{{仮リンク|TAF11|en|TAF11}}はDcr-2とR2D2の四量体化を促進してRLCを組み立て、siRNAに対する結合親和性を10倍増加させる。TAF11との結合によって、R2D2/Dcr2-initiator(RDI)複合体はRLCへと変換される<ref name=":3">{{cite journal|date=September 2015|title=TAF11 Assembles the RISC Loading Complex to Enhance RNAi Efficiency|journal=Molecular Cell|volume=59|issue=5|pages=807–18|doi=10.1016/j.molcel.2015.07.006|pmid=26257286|pmc=4560963|vauthors=Liang C, Wang Y, Murota Y, Liu X, Smith D, Siomi MC, Liu Q}}</ref>。R2D2にはタンデムに並んだdsRNA結合ドメインが存在し、siRNA二本鎖の熱力学的に安定な末端を認識する。一方、Dcr-2は熱力学的安定性の低い末端を認識する。RISCへのRNAのローディングは非対称的であり、Ago2のMIDドメインはsiRNAの熱力学的に不安定な末端を認識する。そのため、5'末端がMIDドメインに認識されなかったパッセンジャー鎖は放出され、もう一方のガイド鎖はAgoと協調的にRISCを形成する<ref name="Nakanishi" />。 |
|||
リポナノ粒子に内包されたRNAが肝細胞に取り込まれるとRNAi作用により[[トランスサイレチン]]の合成を抑制し、異常トランスサイレチンの[[フォールディング|ミスフォールディング]]によるアミロイド形成を抑制する。 |
|||
RISCへと取り込まれた後、siRNAは標的mRNAと塩基対を形成して切断を行い、そのmRNAが翻訳の鋳型として利用されることを防ぐ<ref name=":4">{{cite journal|date=May 2002|title=RNA-dependent RNA polymerases, viruses, and RNA silencing|journal=Science|volume=296|issue=5571|pages=1270–3|bibcode=2002Sci...296.1270A|doi=10.1126/science.1069132|pmid=12016304|vauthors=Ahlquist P|s2cid=42526536}}</ref>。siRNAとは異なり、miRNAがロードされたRISCはmRNA上の相補性領域を探してスキャンする。 miRNAは通常不完全な相補性でmRNAの[[3' 非翻訳領域|3' UTR]]領域に結合し、[[リボソーム]]が翻訳のためにアクセスすることを防ぐ役割を果たす<ref name=":5">{{cite journal|year=2015|title=The microRNA Machinery|journal=Advances in Experimental Medicine and Biology|volume=887|pages=15–30|doi=10.1007/978-3-319-22380-3_2|isbn=978-3-319-22379-7|pmid=26662984|vauthors=Roberts TC}}</ref>。 |
|||
== RNAiの効果が確認されている生物 == |
|||
[[センチュウ]], |
|||
[[コクヌストモドキ]], |
|||
[[ナミテントウ]], |
|||
[[ワモンゴキブリ]], |
|||
[[コオロギ]], |
|||
[[シロアリ]], |
|||
[[酵母]], |
|||
[[イネ]], |
|||
[[シロイヌナズナ]], |
|||
[[タバコ]],ショウジョウバエ |
|||
== |
=== miRNA === |
||
{{Reflist}} |
|||
[[File:Microrna secondary structure.png|thumb|right|''[[ヤセイカンラン|Brassica oleracea]]''由来pri-miRNAのステムループ型[[核酸の二次構造|二次構造]]]] |
|||
{{main|miRNA}} |
|||
miRNAは[[ゲノム]]にコードされた[[ノンコーディングRNA]]であり、特に発生過程において遺伝子発現の調節を補助する<ref name="Zhao Y 2007">{{cite journal|date=May 2007|title=The functions of microRNAs in plants|journal=Frontiers in Bioscience|volume=12|pages=3975–82|doi=10.2741/2364|pmid=17485351|pmc=2851543|vauthors=Wang QL, Li ZH|s2cid=23014413}}<br />{{cite journal|date=April 2007|title=A developmental view of microRNA function|journal=Trends in Biochemical Sciences|volume=32|issue=4|pages=189–97|doi=10.1016/j.tibs.2007.02.006|pmid=17350266|vauthors=Zhao Y, Srivastava D}}</ref>。広義のRNAiには、外来dsRNAから産生されるsiRNAによるものに加え、miRNAによる内因性の遺伝子サイレンシング効果も含まれる。成熟したmiRNAは外因性dsRNAから産生されたsiRNAと構造的に類似しているが、成熟するまでに広範囲の[[転写後修飾]]を必要とする。miRNAは最終産物よりもずっと長いRNAコーディング遺伝子からpri-miRNA(primary miRNA)と呼ばれる一次転写産物として発現し、核内で{{仮リンク|マイクロプロセッサー複合体|en|Microprocessor complex}}によってステムループ構造を持つ約70ヌクレオチドの長さのpre-miRNA(precursor miRNA)へとプロセシングされる。マイクロプロセッサー複合体には、{{仮リンク|Drosha|en|Drosha}}と呼ばれる{{仮リンク|リボヌクレアーゼIII|en|Ribonuclease III|label=RNase III}}酵素とdsRNA結合タンパク質{{仮リンク|DGCR8|en|Microprocessor complex subunit DGCR8}}が含まれる。Dicerはこのpre-miRNAのdsRNA部分に結合して切断を行い、RISCに取り込まれる成熟型miRNA分子が産生される。これより下流では、miRNAとsiRNAは同様の装置を利用する<ref name="Denli">{{cite book|vauthors=Gregory RI, Chendrimada TP, Shiekhattar R|title=MicroRNA Protocols|chapter=MicroRNA biogenesis: isolation and characterization of the microprocessor complex|series=Methods in Molecular Biology|volume=342|pages=33–47|year=2006|pmid=16957365|doi=10.1385/1-59745-123-1:33|isbn=978-1-59745-123-9}}</ref>。ウイルス由来のmiRNAとしては、[[エプスタイン・バール・ウイルス]](EBV)にコードされたものが最初に記載された<ref name="Pfeffer 734–736">{{cite journal|date=April 2004|title=Identification of virus-encoded microRNAs|url=https://semanticscholar.org/paper/bfcee1dfc85e9b172ff5bda21c9b77e38057c24d|journal=Science|volume=304|issue=5671|pages=734–6|bibcode=2004Sci...304..734P|doi=10.1126/science.1096781|pmid=15118162|vauthors=Pfeffer S, Zavolan M, Grässer FA, Chien M, Russo JJ, Ju J, John B, Enright AJ, Marks D, Sander C, Tuschl T|s2cid=25287167}}</ref>。それ以降、ウイルスでは多くのmiRNAの記載がなされている。VIRmiRNAは、ウイルス性のmiRNAとその標的、そして抗ウイルス性miRNAに関する総合的なカタログである<ref name="Qureshi bau103">{{cite journal|date=1 January 2014|title=VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets|journal=Database|volume=2014|pages=bau103|doi=10.1093/database/bau103|pmid=25380780|pmc=4224276|vauthors=Qureshi A, Thakur N, Monga I, Thakur A, Kumar M}}</ref>。 |
|||
miRNAは長いdsRNA前駆体に由来するsiRNAとはいくつかの点で異なる。特に動物では、miRNAと標的mRNAとの塩基対形成は不完全であることが一般的であり、また類似した配列を持つ多くの異なるmRNAの翻訳を阻害する。対照的に、siRNAは通常は完全な塩基対形成を行い、唯一の特異的な標的に対してのみmRNAの切断を誘導する<ref name="Pillai_2006">{{cite journal|year=2007|title=Repression of protein synthesis by miRNAs: how many mechanisms?|journal=Trends Cell Biol|volume=17|issue=3|pages=118–26|doi=10.1016/j.tcb.2006.12.007|pmid=17197185|vauthors=Pillai RS, Bhattacharyya SN, Filipowicz W}}</ref>。ショウジョウバエや''C. elegans''では、miRNAとsiRNAはそれぞれ異なるArgonauteタンパク質とDicer酵素によってプロセシングされる<ref name="Okamura">{{cite journal|year=2004|title=Distinct roles for Argonaute proteins in small RNA-directed RNA cleavage pathways|journal=Genes Dev|volume=18|issue=14|pages=1655–66|doi=10.1101/gad.1210204|pmid=15231716|pmc=478188|vauthors=Okamura K, Ishizuka A, Siomi H, Siomi M}}</ref><ref name="Lee">{{cite journal|year=2004|title=Distinct roles for Drosophila Dicer-1 and Dicer-2 in the siRNA/miRNA silencing pathways|journal=Cell|volume=117|issue=1|pages=69–81|doi=10.1016/S0092-8674(04)00261-2|pmid=15066283|vauthors=Lee Y, Nakahara K, Pham J, Kim K, He Z, Sontheimer E, Carthew R|doi-access=free}}</ref>。 |
|||
=== 3' UTRとmiRNA === |
|||
{{main|3' 非翻訳領域}} |
|||
mRNAの3' UTRには、転写後にRNAiを引き起こす調節配列が存在することが多い。こうした3' UTRには、miRNAの結合部位と調節タンパク質の結合部位の双方が存在することが多い。miRNAは3' UTR内の特定の部位に結合することで、翻訳の阻害または転写産物の分解によって遺伝子発現を低下させる。また、3' UTRにはmRNAの発現を阻害する[[リプレッサー]]タンパク質が結合する[[サイレンサー (遺伝学)|サイレンサー]]領域が存在する場合もある。 |
|||
miRNAの配列とアノテーションがアーカイブされているウェブサイト{{仮リンク|miRBase|en|miRBase}}<ref>{{Cite web |title=miRBase |url=https://mirbase.org/ |website=mirbase.org |accessdate=2022-03-06}}</ref>には、2014年時点で233の生物種の28,645種類のエントリが登録されている。miRNAには平均して約400種類の標的mRNAが存在する(数百の遺伝子の発現に影響を与える)ことが予測されている<ref name="Friedman">{{cite journal|year=2009|title=Most mammalian mRNAs are conserved targets of microRNAs|journal=Genome Res.|volume=19|issue=1|pages=92–105|doi=10.1101/gr.082701.108|pmid=18955434|pmc=2612969|vauthors=Friedman RC, Farh KK, Burge CB, Bartel DP}}</ref>。ヒトのmRNAの3' UTRにはバックグラウンドレベルよりも高い水準で保存されている標的部位が45,000か所以上存在し、タンパク質コーディング遺伝子の60%以上に対してmiRNAとの対合を維持するような選択圧がはたらいていると推定されている<ref name="Friedman" />。 |
|||
1種類のmiRNAが数百種類のmRNAの安定性を低下させる場合があることは、直接的な実験により示されている<ref name="pmid15685193">{{cite journal|date=February 2005|title=Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs|journal=Nature|volume=433|issue=7027|pages=769–73|bibcode=2005Natur.433..769L|doi=10.1038/nature03315|pmid=15685193|vauthors=Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM|s2cid=4430576}}</ref>。一方他の実験では、1種類のmiRNAが数百種類のタンパク質の産生を抑制する可能性があるものの、多くの場合こうした抑制は比較的弱いもの(2倍未満)であることが示されている<ref name=":6">{{cite journal|date=September 2008|title=Widespread changes in protein synthesis induced by microRNAs|journal=Nature|volume=455|issue=7209|pages=58–63|bibcode=2008Natur.455...58S|doi=10.1038/nature07228|pmid=18668040|vauthors=Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N|s2cid=4429008}}</ref><ref name=":7">{{cite journal|date=September 2008|title=The impact of microRNAs on protein output|journal=Nature|volume=455|issue=7209|pages=64–71|bibcode=2008Natur.455...64B|doi=10.1038/nature07242|pmid=18668037|pmc=2745094|vauthors=Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP}}</ref>。 |
|||
miRNAによる遺伝子発現の調節の異常は、[[悪性腫瘍|がん]]において重要であるようである<ref name="pmid21931505">{{cite journal|date=July 2011|title=Mechanisms and role of microRNA deregulation in cancer onset and progression|journal=Genetics and Molecular Biology|volume=34|issue=3|pages=363–70|doi=10.1590/S1415-47572011000300001|pmid=21931505|pmc=3168173|vauthors=Palmero EI, de Campos SG, Campos M, de Souza NC, Guerreiro ID, Carvalho AL, Marques MM}}</ref>。一例として、消化器がんでは9種類のmiRNAに[[エピジェネティクス|エピジェネティック]]な変化が生じ、[[DNA修復]]酵素をダウンレギュレーションする作用を示していることが同定されている<ref name="pmid25987950">{{cite journal|date=May 2015|title=Epigenetic reduction of DNA repair in progression to gastrointestinal cancer|journal=World Journal of Gastrointestinal Oncology|volume=7|issue=5|pages=30–46|doi=10.4251/wjgo.v7.i5.30|pmid=25987950|pmc=4434036|vauthors=Bernstein C, Bernstein H}}</ref>。 |
|||
miRNAによる遺伝子発現調節の異常は、[[統合失調症]]、[[双極性障害]]、[[大うつ病]]、[[パーキンソン病]]、[[アルツハイマー病]]、[[自閉症スペクトラム障害]]など精神神経疾患にも重要であるようである<ref name="pmid24653674">{{cite journal|year=2014|title=Micro spies from the brain to the periphery: new clues from studies on microRNAs in neuropsychiatric disorders|journal=Frontiers in Cellular Neuroscience|volume=8|pages=75|doi=10.3389/fncel.2014.00075|pmid=24653674|pmc=3949217|vauthors=Maffioletti E, Tardito D, Gennarelli M, Bocchio-Chiavetto L|doi-access=free}}</ref><ref name="pmid22539927">{{cite journal|year=2012|title=The Emerging Role of microRNAs in Schizophrenia and Autism Spectrum Disorders|journal=Frontiers in Psychiatry|volume=3|pages=39|doi=10.3389/fpsyt.2012.00039|pmid=22539927|pmc=3336189|vauthors=Mellios N, Sur M|doi-access=free}}</ref><ref name="pmid25636176">{{cite journal|date=August 2015|title=MicroRNA and Posttranscriptional Dysregulation in Psychiatry|journal=Biological Psychiatry|volume=78|issue=4|pages=231–9|doi=10.1016/j.biopsych.2014.12.009|pmid=25636176|vauthors=Geaghan M, Cairns MJ|doi-access=free}}</ref>。 |
|||
=== RISCの活性化と触媒 === |
|||
外因性のdsRNAには、''C. elegans''ではRDE-4、ショウジョウバエではR2D2と呼ばれるエフェクタータンパク質が検知して結合し、Dicerの活性を刺激する<ref name="Parker">{{cite journal|year=2006|title=RDE-4 preferentially binds long dsRNA and its dimerization is necessary for cleavage of dsRNA to siRNA|journal=RNA|volume=12|issue=5|pages=807–18|doi=10.1261/rna.2338706|pmid=16603715|pmc=1440910|vauthors=Parker G, Eckert D, Bass B}}</ref>。このタンパク質は長いdsRNAにのみ結合するが、こうした長さに対する特異性を生み出す機構は不明である<ref name="Parker" />。その後、このRNA結合タンパク質は切断されたsiRNAのRISCへの移行を促進する<ref name="Liu2003">{{cite journal|date=September 2003|title=R2D2, a bridge between the initiation and effector steps of the Drosophila RNAi pathway|journal=Science|volume=301|issue=5641|pages=1921–5|bibcode=2003Sci...301.1921L|doi=10.1126/science.1088710|pmid=14512631|vauthors=Liu Q, Rand TA, Kalidas S, Du F, Kim HE, Smith DP, Wang X|s2cid=41436233}}</ref>。 |
|||
''C. elegans''では、Dicerによって産生された「一次性」のsiRNAを鋳型として「二次性」のsiRNAが合成されることで、この開始応答は増幅される<ref name="Baulcombe D 2007 199–200">{{cite journal|date=January 2007|title=Molecular biology. Amplified silencing|journal=Science|volume=315|issue=5809|pages=199–200|doi=10.1126/science.1138030|pmid=17218517|vauthors=Baulcombe DC|s2cid=46285020}}</ref>。こうした「二次性」のsiRNAはDicerによって産生されたsiRNAとは構造的に異なり、[[RNA依存性RNAポリメラーゼ]](RdRP)によって産生されているようである<ref name="Pak">{{cite journal|date=January 2007|title=Distinct populations of primary and secondary effectors during RNAi in C. elegans|url=https://semanticscholar.org/paper/81e2af9f3952ed7b474665559fdadca2c27505c4|journal=Science|volume=315|issue=5809|pages=241–4|bibcode=2007Sci...315..241P|doi=10.1126/science.1132839|pmid=17124291|vauthors=Pak J, Fire A|s2cid=46620298}}</ref><ref name="Sijen">{{cite journal|date=January 2007|title=Secondary siRNAs result from unprimed RNA synthesis and form a distinct class|journal=Science|volume=315|issue=5809|pages=244–7|bibcode=2007Sci...315..244S|doi=10.1126/science.1136699|pmid=17158288|vauthors=Sijen T, Steiner FA, Thijssen KL, Plasterk RH|s2cid=9483460}}</ref>。[[File:RNAi overview.png|thumb|400x400px|低分子RNAの生合成: pri-miRNAは核内で転写された後、ヘアピン型に折り返され、マイクロプロセッサー複合体によってトリミングされて60–70ヌクレオチドのpre-miRNAが形成される。pre-miRNAは[[核膜孔複合体]](NPC)を通って細胞質へ輸送され、そこでDicerによって約20ヌクレオチドのmiRNA二本鎖へとさらにトリミングされる(pre-siRNAもこの段階で経路に加わる)。その後、この二本鎖はAgoへロードされてpre-RISCが形成され、パッセンジャー鎖が放出されて活性型RISCが形成される。]] |
|||
<nowiki/><nowiki/><nowiki/>[[File:Argonaute 1u04 1ytu composite.png|thumb|left|300px|左: [[古細菌]] ''[[Pyrococcus furiosus]]''由来のArgonauteタンパク質全長構造。右: dsRNAと複合体を形成したArgonauteタンパク質の{{仮リンク|Piwi|en|Piwi}}ドメイン。]] |
|||
RISCの活性を担う構成要素はArgonauteタンパク質と呼ばれる{{仮リンク|エンドヌクレアーゼ|en|Endonuclease|redirect=1}}であり、結合したsiRNAに相補的な標的mRNAを切断する<ref name="Daneholt2006">{{cite web |title=Advanced Information: RNA interference |vauthors=Daneholt B |website=The Nobel Prize in Physiology or Medicine 2006 |url=http://nobelprize.org/nobel_prizes/medicine/laureates/2006/adv.html |access-date=25 January 2007 |archive-url=https://web.archive.org/web/20070120113455/http://nobelprize.org/nobel_prizes/medicine/laureates/2006/adv.html <!-- Added by H3llBot --> |archive-date=20 January 2007}}</ref>。Dicerによって形成される断片は二本鎖であるため、理論上は双方の鎖が機能的なsiRNAとなることができる。しかしながら、2本の鎖のうちArgonauteタンパク質に結合し、遺伝子サイレンシングを指揮するのは、ガイド鎖と呼ばれる一方の鎖のみである。もう一方の鎖はパッセンジャー鎖またはアンチガイド鎖と呼ばれ、RISCの活性化の過程で分解される<ref name="Gregory">{{cite journal|date=November 2005|title=Human RISC couples microRNA biogenesis and posttranscriptional gene silencing|journal=Cell|volume=123|issue=4|pages=631–40|doi=10.1016/j.cell.2005.10.022|pmid=16271387|vauthors=Gregory RI, Chendrimada TP, Cooch N, Shiekhattar R|doi-access=free}}</ref>。当初は[[アデノシン三リン酸|ATP]]依存的な[[ヘリカーゼ]]によって2つの鎖が分離されると考えられていたが<ref name="Lodish">{{cite book|title=Molecular Cell Biology|edition=5th|publisher=WH Freeman: New York, NY|year=2004|url=https://archive.org/details/molecularcellbio00harv|isbn=978-0-7167-4366-8|vauthors=Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipurksy SL, Darnell J|url-access=registration}}</ref>、この過程は実際にはATP非依存的であり、RISCの構成要素によって直接行われることが示されている<ref name="Matranga">{{cite journal|date=November 2005|title=Passenger-strand cleavage facilitates assembly of siRNA into Ago2-containing RNAi enzyme complexes|journal=Cell|volume=123|issue=4|pages=607–20|doi=10.1016/j.cell.2005.08.044|pmid=16271386|vauthors=Matranga C, Tomari Y, Shin C, Bartel DP, Zamore PD|doi-access=free}}</ref><ref name="Leuschner">{{cite journal|date=March 2006|title=Cleavage of the siRNA passenger strand during RISC assembly in human cells|journal=EMBO Reports|volume=7|issue=3|pages=314–20|doi=10.1038/sj.embor.7400637|pmid=16439995|pmc=1456892|vauthors=Leuschner PJ, Ameres SL, Kueng S, Martinez J}}</ref>。しかしながら、''in vitro''におけるATP存在下と非存在下でのRNAiの速度論的解析からは、触媒後の複合体から切断されたmRNAを巻き戻して除去するためにATPが必要である可能性が示されている<ref name="Haley 2004 599–606">{{cite journal|date=July 2004|title=Kinetic analysis of the RNAi enzyme complex|journal=Nature Structural & Molecular Biology|volume=11|issue=7|pages=599–606|doi=10.1038/nsmb780|pmid=15170178|vauthors=Haley B, Zamore PD|s2cid=12400060}}</ref>。ガイド鎖は5'末端の対合の安定性が低い傾向があるが<ref name=":8">{{cite journal|date=October 2003|title=Asymmetry in the assembly of the RNAi enzyme complex|journal=Cell|volume=115|issue=2|pages=199–208|doi=10.1016/S0092-8674(03)00759-1|pmid=14567917|vauthors=Schwarz DS, Hutvágner G, Du T, Xu Z, Aronin N, Zamore PD|doi-access=free}}</ref>、RISCへの取り込みの前のDicerによるdsRNAの切断の方向には影響を与えない<ref name="Preall">{{cite journal|date=March 2006|title=Short interfering RNA strand selection is independent of dsRNA processing polarity during RNAi in Drosophila|journal=Current Biology|volume=16|issue=5|pages=530–5|doi=10.1016/j.cub.2006.01.061|pmid=16527750|vauthors=Preall JB, He Z, Gorra JM, Sontheimer EJ|doi-access=free}}</ref>。Dicerではなく、R2D2タンパク質がより安定性の高いパッセンジャー鎖の5'末端に結合することが鎖の識別因子として機能している可能性がある<ref name="Tomari">{{cite journal|date=November 2004|title=A protein sensor for siRNA asymmetry|journal=Science|volume=306|issue=5700|pages=1377–80|bibcode=2004Sci...306.1377T|doi=10.1126/science.1102755|pmid=15550672|vauthors=Tomari Y, Matranga C, Haley B, Martinez N, Zamore PD|s2cid=31558409}}</ref>。 |
|||
Argonauteタンパク質のRNA結合の構造的基盤は、RNAが結合したArgonauteタンパク質のドメインの[[X線結晶構造解析]]によって研究されている。RNA鎖のリン酸化5'末端はArgonauteタンパク質の保存された塩基性ポケットに入り、[[マグネシウム]]などの二価[[カチオン]]を介して、また5'末端のヌクレオチドと保存された[[チロシン]]残基との[[Π-π相互作用|スタッキング]]によって接触を行っている。この部位はsiRNAが標的mRNAへ結合する際の核形成部位となると考えられている<ref name="Ma">{{cite journal|date=March 2005|title=Structural basis for 5'-end-specific recognition of guide RNA by the A. fulgidus Piwi protein|journal=Nature|volume=434|issue=7033|pages=666–70|bibcode=2005Natur.434..666M|doi=10.1038/nature03514|pmid=15800629|pmc=4694588|vauthors=Ma JB, Yuan YR, Meister G, Pei Y, Tuschl T, Patel DJ}}</ref>。ガイド鎖の5'末端または3'末端のミスマッチによる阻害効果の解析からは、ガイド鎖の5'末端は標的mRNAとのマッチングと結合を担う一方で、3'末端は標的mRNAをRISCが切断を行いやすい領域へ物理的に配置をする役割を担っている可能性が高いことが示されている<ref name="Haley 2004 599–606" />。 |
|||
活性化されたRISC複合体が細胞内のmRNA標的をどのように見つけているのかに関しては未解明である。切断過程と翻訳との関連性が提唱されているが、標的mRNAの翻訳はRNAiを介した分解に必要不可欠なものではない<ref name=":9">{{cite journal|year=2005|title=mRNA translation is not a prerequisite for small interfering RNA-mediated mRNA cleavage|journal=Differentiation|volume=73|issue=6|pages=287–93|doi=10.1111/j.1432-0436.2005.00029.x|pmid=16138829|vauthors=Sen G, Wehrman T, Blau H|s2cid=41117614}}</ref>。事実、RNAiは翻訳されていない標的mRNAに対してより効率的に働いている可能性がある<ref name="Gu">{{cite journal|year=2005|title=Uncoupling of RNAi from active translation in mammalian cells|journal=RNA|volume=11|issue=1|pages=38–44|doi=10.1261/rna.7158605|pmid=15574516|pmc=1370689|vauthors=Gu S, Rossi J}}</ref>。Argonauteタンパク質は{{仮リンク|P-body|en|P-bodies}}(cytoplasmic body、GW bodyとも)と呼ばれる細胞質の特定の領域に局在している。この領域ではmRNAの分解が高率で行われており<ref name="SenBlau">{{cite journal|year=2005|title=Argonaute 2/RISC resides in sites of mammalian mRNA decay known as cytoplasmic bodies|journal=Nat Cell Biol|volume=7|issue=6|pages=633–6|doi=10.1038/ncb1265|pmid=15908945|vauthors=Sen G, Blau H|s2cid=6085169}}</ref>、miRNAの活性もP-bodyに集中している<ref name="Lian">{{cite journal|year=2006|title=GW bodies, microRNAs and the cell cycle|journal=Cell Cycle|volume=5|issue=3|pages=242–5|doi=10.4161/cc.5.3.2410|pmid=16418578|vauthors=Lian S, Jakymiw A, Eystathioy T, Hamel J, Fritzler M, Chan E|doi-access=free}}</ref>。P-bodyの破壊によってRNAiの効率が低下することから、この部位がRNAi過程に重要であることが示唆される<ref name="Jakymiw">{{cite journal|year=2005|title=Disruption of P bodies impairs mammalian RNA interference|journal=Nat Cell Biol|volume=7|issue=12|pages=1267–74|doi=10.1038/ncb1334|pmid=16284622|vauthors=Jakymiw A, Lian S, Eystathioy T, Li S, Satoh M, Hamel J, Fritzler M, Chan E|s2cid=36630239}}</ref>。 |
|||
=== 転写サイレンシング === |
|||
[[File:RNAi-simplified.png|thumb|350px|right|DicerはdsRNAをトリミングし、siRNAまたはmiRNAを形成する。こうしたプロセシングを受けたRNAはRISCに取り込まれ、DNAや新生RNA鎖を標的として転写をサイレンシングしたり、mRNAを標的として翻訳を阻害したりする<ref name="Hammond2000">{{cite journal|year=2000|title=An RNA-directed nuclease mediates post-transcriptional gene silencing in Drosophila cells|journal=[[Nature (journal)|Nature]]|volume=404|issue=6775|pages=293–6|bibcode=2000Natur.404..293H|doi=10.1038/35005107|pmid=10749213|vauthors=Hammond S, Bernstein E, Beach D, [[Gregory Hannon|Hannon G]]|s2cid=9091863}} {{closed access}}</ref>。]] |
|||
RNAi経路の構成要素は、多くの真核生物においてゲノムの組織化と構造の維持に利用されている。[[ヒストン]]の修飾とそれに関連した[[ヘテロクロマチン]]形成の誘導は、転写前段階で遺伝子をダウンレギュレーションする役割を果たす<ref name="Holmquist_2006">{{cite journal|year=2006|title=Chromosome organization and chromatin modification: influence on genome function and evolution|journal=Cytogenetic and Genome Research|volume=114|issue=2|pages=96–125|doi=10.1159/000093326|pmid=16825762|vauthors=Holmquist GP, Ashley T|s2cid=29910065}}</ref>。この過程は{{仮リンク|RNA誘導転写サイレンシング|en|RNA-induced transcriptional silencing}}(RITS)と呼ばれ、RITS複合体と呼ばれるタンパク質複合体によって行われる。[[分裂酵母]]''Schizosaccharomyces pombe''では、この複合体にはArgonaute、{{仮リンク|クロモドメイン|en|Chromodomain}}タンパク質Chp1、そしてTas3と呼ばれる機能未知タンパク質が含まれる<ref name="Verdel">{{cite journal|date=January 2004|title=RNAi-mediated targeting of heterochromatin by the RITS complex|journal=Science|volume=303|issue=5658|pages=672–6|bibcode=2004Sci...303..672V|doi=10.1126/science.1093686|pmid=14704433|pmc=3244756|vauthors=Verdel A, Jia S, Gerber S, Sugiyama T, Gygi S, Grewal SI, Moazed D}}</ref>。ヘテロクロマチン領域の誘導と拡大にはArgonauteとRdRPタンパク質が必要である<ref name="Irvine">{{cite journal|date=August 2006|title=Argonaute slicing is required for heterochromatic silencing and spreading|url=https://semanticscholar.org/paper/4e7cc7c6cf13fe3874f83d4ede6ad61db2c3dcdf|journal=Science|volume=313|issue=5790|pages=1134–7|bibcode=2006Sci...313.1134I|doi=10.1126/science.1128813|pmid=16931764|vauthors=Irvine DV, Zaratiegui M, Tolia NH, Goto DB, Chitwood DH, Vaughn MW, Joshua-Tor L, Martienssen RA|s2cid=42997104}}</ref>。これらの遺伝子を欠失させた分裂酵母は、{{仮リンク|ヒストンメチル化|en|Histone methylation}}と[[セントロメア]]形成が破壊され<ref name="Volpe_2002">{{cite journal|date=September 2002|title=Regulation of heterochromatic silencing and histone H3 lysine-9 methylation by RNAi|url=https://semanticscholar.org/paper/b334b093326b71ba98bb761d5b99fc6e9e32bf3e|journal=Science|volume=297|issue=5588|pages=1833–7|bibcode=2002Sci...297.1833V|doi=10.1126/science.1074973|pmid=12193640|vauthors=Volpe TA, Kidner C, Hall IM, Teng G, Grewal SI, Martienssen RA|s2cid=2613813}}</ref>、[[細胞分裂]]の進行は遅くなるか、もしくは[[後期 (細胞分裂)|後期]]の段階で停止する<ref name="Volpe_2003">{{cite journal|year=2003|title=RNA interference is required for normal centromere function in fission yeast|journal=Chromosome Research|volume=11|issue=2|pages=137–46|doi=10.1023/A:1022815931524|pmid=12733640|vauthors=Volpe T, Schramke V, Hamilton GL, White SA, Teng G, Martienssen RA, Allshire RC|s2cid=23813417}}</ref>。またある場合には、ヒストン修飾と関係した同様の過程によって遺伝子の転写がアップレギュレーションされることも観察されている<ref name="Li">{{cite journal|date=November 2006|title=Small dsRNAs induce transcriptional activation in human cells|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=103|issue=46|pages=17337–42|bibcode=2006PNAS..10317337L|doi=10.1073/pnas.0607015103|pmid=17085592|pmc=1859931|vauthors=Li LC, Okino ST, Zhao H, Pookot D, Place RF, Urakami S, Enokida H, Dahiya R|doi-access=free}}</ref>。 |
|||
RITS複合体がヘテロクロマチンの形成や組織化を誘導する機構の詳細は未解明である。多くの研究は分裂酵母の接合型遺伝子座に焦点を当てているが、この遺伝子座における活性は他の生物やゲノム領域における活性を代表するものではない可能性もある。既存のヘテロクロマチン領域の維持に際しては、RITSはその領域の遺伝子に相補的なsiRNAと複合体を形成してその領域のメチル化ヒストンと安定に結合し、RNAポリメラーゼによって転写が開始されたpre-mRNA新生鎖を、転写と共役した形で分解している。ヘテロクロマチン領域の維持ではなく形成過程はDicer依存的であるが、それはおそらく転写産物を標的とするsiRNAが最初に形成される際にはDicerが必要であるためである<ref name="Noma">{{cite journal|date=November 2004|title=RITS acts in cis to promote RNA interference-mediated transcriptional and post-transcriptional silencing|journal=Nature Genetics|volume=36|issue=11|pages=1174–80|doi=10.1038/ng1452|pmid=15475954|vauthors=Noma K, Sugiyama T, Cam H, Verdel A, Zofall M, Jia S, Moazed D, Grewal SI|doi-access=free}}</ref>。新たなsiRNAは偶発的な転写による新生鎖からRdRPによって形成され、その領域に位置するRITS複合体へ取り込まれるため、ヘテロクロマチンの維持は自己強化型フィードバックループとして機能することが示唆されている<ref name="Sugiyama">{{cite journal|date=January 2005|title=RNA-dependent RNA polymerase is an essential component of a self-enforcing loop coupling heterochromatin assembly to siRNA production|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=102|issue=1|pages=152–7|bibcode=2005PNAS..102..152S|doi=10.1073/pnas.0407641102|pmid=15615848|pmc=544066|vauthors=Sugiyama T, Cam H, Verdel A, Moazed D, Grewal SI|doi-access=free}}</ref>。分裂酵母の接合型遺伝子座やセントロメアにおける観察と哺乳類での現象との対応は明らかではなく、哺乳類細胞におけるヘテロクロマチンの維持はRNAi経路の構成要素とは無関係である可能性もある<ref name="Wang_2006">{{cite journal|date=June 2006|title=The assembly and maintenance of heterochromatin initiated by transgene repeats are independent of the RNA interference pathway in mammalian cells|journal=Molecular and Cellular Biology|volume=26|issue=11|pages=4028–40|doi=10.1128/MCB.02189-05|pmid=16705157|pmc=1489094|vauthors=Wang F, Koyama N, Nishida H, Haraguchi T, Reith W, Tsukamoto T}}</ref>。 |
|||
=== RNA編集とのクロストーク === |
|||
高等真核生物で最も広くみられる[[RNA編集]]は、[[ADAR]]によるdsRNA中の[[アデノシン]]ヌクレオチド(A)の[[イノシン]](I)への変換である<ref name="Bass_2002">{{cite journal|year=2002|title=RNA editing by adenosine deaminases that act on RNA|journal=Annual Review of Biochemistry|volume=71|pages=817–46|doi=10.1146/annurev.biochem.71.110601.135501|pmid=12045112|pmc=1823043|vauthors=Bass BL}}</ref>。RNAiとA→IのRNA編集経路が共通したdsRNA基質をめぐって競合する可能性は2000年に提唱された<ref name="Bass_2000">{{cite journal|date=April 2000|title=Double-stranded RNA as a template for gene silencing|journal=Cell|volume=101|issue=3|pages=235–8|doi=10.1016/S0092-8674(02)71133-1|pmid=10847677|vauthors=Bass BL|doi-access=free}}</ref>。一部のpre-miRNAはA→I RNA編集を受け<ref name="Luciano_2004">{{cite journal|date=August 2004|title=RNA editing of a miRNA precursor|journal=RNA|volume=10|issue=8|pages=1174–7|doi=10.1261/rna.7350304|pmid=15272117|pmc=1370607|vauthors=Luciano DJ, Mirsky H, Vendetti NJ, Maas S}}</ref><ref name="yang2006">{{cite journal|date=January 2006|title=Modulation of microRNA processing and expression through RNA editing by ADAR deaminases|journal=Nature Structural & Molecular Biology|volume=13|issue=1|pages=13–21|doi=10.1038/nsmb1041|pmid=16369484|pmc=2950615|vauthors=Yang W, Chendrimada TP, Wang Q, Higuchi M, Seeburg PH, Shiekhattar R, Nishikura K}}</ref>、この機構は成熟型miRNAへのプロセシングと発現を調節している可能性がある<ref name="yang2006" />。さらに、哺乳類のADARの少なくとも1種類に関してはsiRNAをRNAi経路の構成要素から隔離することが示されている<ref name="Yang_2005">{{cite journal|date=February 2005|title=ADAR1 RNA deaminase limits short interfering RNA efficacy in mammalian cells|journal=The Journal of Biological Chemistry|volume=280|issue=5|pages=3946–53|doi=10.1074/jbc.M407876200|pmid=15556947|pmc=2947832|vauthors=Yang W, Wang Q, Howell KL, Lee JT, Cho DS, Murray JM, Nishikura K|doi-access=free}}</ref>。ADARを持たない''C. elegans''系統を用いた研究からは、内在性遺伝子や導入遺伝子のRNAiによるサイレンシングにA→I RNA編集が対抗していることが示されており、このこともこのモデルを支持している<ref name="Nishikura_2006">{{cite journal|date=December 2006|title=Editor meets silencer: crosstalk between RNA editing and RNA interference|journal=Nature Reviews Molecular Cell Biology|volume=7|issue=12|pages=919–31|doi=10.1038/nrm2061|pmid=17139332|pmc=2953463|vauthors=Nishikura K}}</ref>。[[File:Rnai diagram retrovirology.png|thumb|300px|right|植物と動物における遺伝子サイレンシグの大きな差異。内因性に発現したmiRNAまたは外因性のsiRNAはDicerによってプロセシングされてRISCに取り込まれ、遺伝子サイレンシングを媒介する<ref name="Saumet">{{cite journal|year=2006|title=Anti-viral RNA silencing: do we look like plants ?|journal=Retrovirology|volume=3|issue=1|page=3|doi=10.1186/1742-4690-3-3|pmid=16409629|pmc=1363733|vauthors=Saumet A, Lecellier CH}}</ref>。]] |
|||
=== 生物種間の多様性 === |
|||
外来dsRNAを取り込み、そしてそれらをRNAi経路で利用する能力は、生物によって差がある。RNAiの効果は、植物や''C. elegan''では全身的かつ遺伝性のものであるが、ショウジョウバエや哺乳類ではそうではない。植物では、RNAiは[[原形質連絡]](細胞間のコミュニケーションや輸送を可能にする、[[細胞壁]]に存在するチャネル)を介した細胞間でのsiRNAの輸送によって全身へ伝播していくと考えられている<ref name="Lodish" />。またその遺伝性はRNAiの標的となったプロモーターがメチル化されることによるものであり、メチル化パターンは新たな世代の細胞が生じるたびにコピーされる<ref name=":10">{{cite journal|date=May 2001|title=RNA-directed transcriptional gene silencing in plants can be inherited independently of the RNA trigger and requires Met1 for maintenance|journal=Current Biology|volume=11|issue=10|pages=747–57|doi=10.1016/S0960-9822(01)00226-3|pmid=11378384|vauthors=Jones L, Ratcliff F, Baulcombe DC|s2cid=16789197|doi-access=free}}</ref>。植物と動物のおおまかな違いは、内因的に産生されるmiRNAの標的性にある。植物では、通常miRNAはその標的遺伝子に対して(ほぼ)完全に相補的であり、RISCによる直接的なmRNAの切断が誘導されるのに対し、動物のmiRNAは標的となる配列がより多様である傾向があり、翻訳抑制が誘導される<ref name="Saumet" />。この翻訳抑制効果は、翻訳開始因子とmRNAの[[ポリアデニル化]]テールとの相互作用の阻害によって行われている可能性がある<ref name="Humphreys">{{cite journal|date=November 2005|title=MicroRNAs control translation initiation by inhibiting eukaryotic initiation factor 4E/cap and poly(A) tail function|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=102|issue=47|pages=16961–6|bibcode=2005PNAS..10216961H|doi=10.1073/pnas.0506482102|pmid=16287976|pmc=1287990|vauthors=Humphreys DT, Westman BJ, Martin DI, Preiss T|doi-access=free}}</ref>。 |
|||
[[リーシュマニア]]''Leishmania major''や[[トリパノソーマ]]''[[Trypanosoma cruzi]]''など、一部の原生動物にはRNAi経路が全く存在しない<ref name="DaRocha_2004">{{cite journal|date=February 2004|title=Tests of cytoplasmic RNA interference (RNAi) and construction of a tetracycline-inducible T7 promoter system in Trypanosoma cruzi|journal=Molecular and Biochemical Parasitology|volume=133|issue=2|pages=175–86|doi=10.1016/j.molbiopara.2003.10.005|pmid=14698430|vauthors=DaRocha WD, Otsu K, Teixeira SM, Donelson JE}}</ref><ref name="Robinson_2003">{{cite journal|date=May 2003|title=Improvements in transfection efficiency and tests of RNA interference (RNAi) approaches in the protozoan parasite Leishmania|journal=Molecular and Biochemical Parasitology|volume=128|issue=2|pages=217–28|doi=10.1016/S0166-6851(03)00079-3|pmid=12742588|vauthors=Robinson KA, Beverley SM}}</ref>。一部の[[菌類]]でも大部分またはすべての構成要素が存在せず、そうした生物として最も有名なのは[[モデル生物]]でもある[[出芽酵母]]''Saccharomyces cerevisiae''である<ref name="Aravind">{{cite journal|date=October 2000|title=Lineage-specific loss and divergence of functionally linked genes in eukaryotes|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=97|issue=21|pages=11319–24|bibcode=2000PNAS...9711319A|doi=10.1073/pnas.200346997|pmid=11016957|pmc=17198|vauthors=Aravind L, Watanabe H, Lipman DJ, Koonin EV|doi-access=free}}</ref>。''Saccharomyces castellii''や[[カンジダ]]''Candida albicans''などの他の出芽酵母にはRNAiが存在し、''S. castellii''由来の2つのRNAi関連タンパク質を誘導することで、''S. cerevisiae''でもRNAiが促進されることが示されている<ref name=":11">{{cite journal|date=October 2009|title=RNAi in budding yeast|journal=Science|volume=326|issue=5952|pages=544–550|bibcode=2009Sci...326..544D|doi=10.1126/science.1176945|pmid=19745116|pmc=3786161|vauthors=Drinnenberg IA, Weinberg DE, Xie KT, Mower JP, Wolfe KH, Fink GR, Bartel DP}}</ref>。[[子嚢菌門]]や[[担子菌門]]の特定の種でRNAi経路が存在しないことは、RNAサイレンシングに必要なタンパク質が多くの菌類系統で独立に失われたことを示しており、おそらく類似した機能を持つ新たな経路の進化か、特定の[[ニッチ]]において選択上の有利さが存在しなかったためであると考えられる<ref name="Nakayashiki">{{cite journal|date=July 2006|title=Evolution and diversification of RNA silencing proteins in fungi|url=http://www.lib.kobe-u.ac.jp/repository/90000024.pdf|journal=Journal of Molecular Evolution|volume=63|issue=1|pages=127–35|bibcode=2006JMolE..63..127N|doi=10.1007/s00239-005-0257-2|pmid=16786437|vauthors=Nakayashiki H, Kadotani N, Mayama S|s2cid=22639035}}</ref>。 |
|||
=== 関連する原核生物のシステム === |
|||
[[原核生物]]における遺伝子発現は、いくつかの面でRNAiと類似したRNAベースの系の影響を受ける。RNAをコードする遺伝子は、mRNAとアニーリングする相補的なRNAを産生することで、mRNAの存在量や翻訳を制御する。しかし、こうした調節性RNAにはDicerは関与せず、一般的にはmiRNAに類似したものとはみなされていない<ref name="Morita_2006">{{cite journal|date=March 2006|title=Translational repression is sufficient for gene silencing by bacterial small noncoding RNAs in the absence of mRNA destruction|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=103|issue=13|pages=4858–63|bibcode=2006PNAS..103.4858M|doi=10.1073/pnas.0509638103|pmid=16549791|pmc=1458760|vauthors=Morita T, Mochizuki Y, Aiba H|doi-access=free}}</ref>。原核生物における[[CRISPR]]系が真核生物におけるRNAi系と類似したものであることも示唆されているが、どのタンパク質構成要素も[[オーソロガス]]ではない<ref name="makarova">{{cite journal|date=March 2006|title=A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action|journal=Biology Direct|volume=1|pages=7|doi=10.1186/1745-6150-1-7|pmid=16545108|pmc=1462988|vauthors=Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV}}</ref>。 |
|||
== 生物学的機能 == |
|||
=== 免疫 === |
|||
RNAiはウイルスやその他の外来性遺伝物質に対する[[免疫応答]]の重要な部分をなしており、特に植物では[[トランスポゾン]]の自己増殖も防いでいる<ref name="Stram_2006">{{cite journal|date=June 2006|title=Inhibition of viruses by RNA interference|journal=Virus Genes|volume=32|issue=3|pages=299–306|doi=10.1007/s11262-005-6914-0|pmid=16732482|pmc=7088519|vauthors=Stram Y, Kuzntzova L}}</ref>。[[シロイヌナズナ]]''Arabidopsis thaliana''などの植物は複数のDicerホモログを発現しており、これらは植物が異なるウイルスに曝露した際には異なる反応を示すよう専門化されている<ref name="Blevins">{{cite journal|year=2006|title=Four plant Dicers mediate viral small RNA biogenesis and DNA virus induced silencing|journal=Nucleic Acids Research|volume=34|issue=21|pages=6233–46|doi=10.1093/nar/gkl886|pmid=17090584|pmc=1669714|vauthors=Blevins T, Rajeswaran R, Shivaprasad PV, Beknazariants D, Si-Ammour A, Park HS, Vazquez F, Robertson D, Meins F, Hohn T, Pooggin MM}}</ref>。RNAi経路が十分に理解される前から、植物における遺伝子サイレンシングの誘導がその植物全体に伝播したり、また[[接ぎ木]]によって台木から接ぎ穂へ移行したりすることがあることが知られていた<ref name="Palauqui">{{cite journal|date=August 1997|title=Systemic acquired silencing: transgene-specific post-transcriptional silencing is transmitted by grafting from silenced stocks to non-silenced scions|journal=The EMBO Journal|volume=16|issue=15|pages=4738–45|doi=10.1093/emboj/16.15.4738|pmid=9303318|pmc=1170100|vauthors=Palauqui JC, Elmayan T, Pollien JM, Vaucheret H}}</ref>。この現象は、ウイルスに最初に遭遇した後、植物全体がウイルスへ応答することを可能にする、植物の免疫系の特徴として認識されていた<ref name="Voinnet">{{cite journal|date=August 2001|title=RNA silencing as a plant immune system against viruses|journal=Trends in Genetics|volume=17|issue=8|pages=449–59|doi=10.1016/S0168-9525(01)02367-8|pmid=11485817|vauthors=Voinnet O}}</ref>。一方、多くの植物ウイルスもRNAi経路を抑制する精巧な機構を進化させてきた<ref name="Lucy">{{cite journal|date=April 2000|title=Suppression of post-transcriptional gene silencing by a plant viral protein localized in the nucleus|journal=The EMBO Journal|volume=19|issue=7|pages=1672–80|doi=10.1093/emboj/19.7.1672|pmid=10747034|pmc=310235|vauthors=Lucy AP, Guo HS, Li WX, Ding SW}}</ref>。こうしたものの中には、Dicerによって産生される一本鎖オーバーハング末端を持つ短い二本鎖RNA断片に対して結合するウイルスタンパク質などが含まれる<ref name="Merai">{{cite journal|date=June 2006|title=Double-stranded RNA binding may be a general plant RNA viral strategy to suppress RNA silencing|journal=Journal of Virology|volume=80|issue=12|pages=5747–56|doi=10.1128/JVI.01963-05|pmid=16731914|pmc=1472586|vauthors=Mérai Z, Kerényi Z, Kertész S, Magna M, Lakatos L, Silhavy D}}</ref>。一部の植物のゲノムは、特定種の[[細菌]]の感染に対する応答として、内因性のsiRNAを発現する<ref name="Katiyar-Agarwal">{{cite journal|date=November 2006|title=A pathogen-inducible endogenous siRNA in plant immunity|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=103|issue=47|pages=18002–7|bibcode=2006PNAS..10318002K|doi=10.1073/pnas.0608258103|pmid=17071740|pmc=1693862|vauthors=Katiyar-Agarwal S, Morgan R, Dahlbeck D, Borsani O, Villegas A, Zhu JK, Staskawicz BJ, Jin H|doi-access=free}}</ref>。これらは、感染過程の助けとなりうる宿主のあらゆる代謝過程をダウンレギュレーションする、病原体に対する全般的応答の一部である可能性がある<ref name="Fritz">{{cite journal|date=June 2006|title=Innate immune defense through RNA interference|journal=Science's STKE|volume=2006|issue=339|pages=pe27|doi=10.1126/stke.3392006pe27|pmid=16772641|vauthors=Fritz JH, Girardin SE, Philpott DJ|s2cid=33972766}}</ref>。 |
|||
一般的に動物で発現しているDicerの種類は植物よりも少ないが、一部の動物ではRNAiが抗ウイルス応答を行っている。ショウジョウバエでは幼体と成体の双方において、RNAiは抗ウイルス免疫応答に重要であり、[[ショウジョウバエXウイルス]]などの病原体に対して活性を示す<ref name="Zambon">{{cite journal|date=May 2006|title=RNAi is an antiviral immune response against a dsRNA virus in Drosophila melanogaster|journal=Cellular Microbiology|volume=8|issue=5|pages=880–9|doi=10.1111/j.1462-5822.2006.00688.x|pmid=16611236|vauthors=Zambon RA, Vakharia VN, Wu LP|s2cid=32439482|doi-access=free}}</ref><ref name="Wang">{{cite journal|date=April 2006|title=RNA interference directs innate immunity against viruses in adult Drosophila|journal=Science|volume=312|issue=5772|pages=452–4|bibcode=2006Sci...312..452W|doi=10.1126/science.1125694|pmid=16556799|pmc=1509097|vauthors=Wang XH, Aliyari R, Li WX, Li HW, Kim K, Carthew R, Atkinson P, Ding SW}}</ref>。免疫における同様の役割は''C. elegans''でも作用している可能性があり、ウイルスに応答してArgonauteタンパク質はアップレギュレーションされ、またRNAi経路のの構成要素を過剰発現した線虫はウイルス感染に対して耐性を示す<ref name="Lu">{{cite journal|date=August 2005|title=Animal virus replication and RNAi-mediated antiviral silencing in Caenorhabditis elegans|journal=Nature|volume=436|issue=7053|pages=1040–1043|bibcode=2005Natur.436.1040L|doi=10.1038/nature03870|pmid=16107851|pmc=1388260|vauthors=Lu R, Maduro M, Li F, Li HW, Broitman-Maduro G, Li WX, Ding SW}}</ref><ref name="Wilkins">{{cite journal|date=August 2005|title=RNA interference is an antiviral defence mechanism in Caenorhabditis elegans|journal=Nature|volume=436|issue=7053|pages=1044–7|bibcode=2005Natur.436.1044W|doi=10.1038/nature03957|pmid=16107852|vauthors=Wilkins C, Dishongh R, Moore SC, Whitt MA, Chow M, Machaca K|s2cid=4431035}}</ref>。 |
|||
哺乳類の[[自然免疫系|自然免疫]]におけるRNAiの役割の理解は進んでおらず、比較的わずかなデータしか存在しない。哺乳類細胞における機能的な抗ウイルスRNAi経路の存在を示す証拠は提示されており<ref name=":12">{{cite journal|date=October 2013|title=Antiviral RNA interference in mammalian cells|journal=Science|volume=342|issue=6155|pages=235–8|bibcode=2013Sci...342..235M|doi=10.1126/science.1241930|pmid=24115438|pmc=3853215|vauthors=Maillard PV, Ciaudo C, Marchais A, Li Y, Jay F, Ding SW, Voinnet O}}</ref><ref name=":13">{{cite journal|date=October 2013|title=RNA interference functions as an antiviral immunity mechanism in mammals|journal=Science|volume=342|issue=6155|pages=231–4|bibcode=2013Sci...342..231L|doi=10.1126/science.1241911|pmid=24115437|pmc=3875315|vauthors=Li Y, Lu J, Han Y, Fan X, Ding SW}}</ref>、また哺乳類細胞のRNAi応答を抑制する遺伝子をコードするウイルスの存在は哺乳類におけるRNAi依存的な免疫応答を支持する証拠となる可能性がある<ref name="Berkhout">{{cite journal|date=May 2006|title=The interplay between virus infection and the cellular RNA interference machinery|journal=FEBS Letters|volume=580|issue=12|pages=2896–902|doi=10.1016/j.febslet.2006.02.070|pmid=16563388|pmc=7094296|vauthors=Berkhout B, Haasnoot J}}</ref><ref name="Schutz">{{cite journal|date=January 2006|title=Interaction of viruses with the mammalian RNA interference pathway|journal=Virology|volume=344|issue=1|pages=151–7|doi=10.1016/j.virol.2005.09.034|pmid=16364746|vauthors=Schütz S, Sarnow P|doi-access=free}}</ref>。しかしながら、この仮説には十分な証拠がないとして異議も唱えられている<ref name="Cullen">{{cite journal|date=June 2006|title=Is RNA interference involved in intrinsic antiviral immunity in mammals?|journal=Nature Immunology|volume=7|issue=6|pages=563–7|doi=10.1038/ni1352|pmid=16715068|vauthors=Cullen BR|s2cid=23467688}}</ref>。 |
|||
哺乳類のウイルスにおけるRNAiには他の機能もあり、[[ヘルペスウイルス]]が発現するmiRNAはウイルス潜伏を媒介するヘテロクロマチン組織化の引き金として作用している可能性がある<ref name="Li3">{{cite journal|date=October 2005|title=Antiviral silencing in animals|journal=FEBS Letters|volume=579|issue=26|pages=5965–73|doi=10.1016/j.febslet.2005.08.034|pmid=16154568|pmc=1350842|vauthors=Li HW, Ding SW}}</ref>。 |
|||
=== 遺伝子のダウンレギュレーション === |
|||
内因的に発現しているmiRNAは、[[イントロン]]内に位置するのものも遺伝子間領域に位置するものも、翻訳抑制<ref name="Saumet" />そして発生の調節に最も重要であり、[[形態形成]]の時期の決定や、[[幹細胞]]などの未分化状態や不完全分化状態の細胞種の維持に特に重要である<ref name="Carrington">{{cite journal|date=July 2003|title=Role of microRNAs in plant and animal development|url=https://semanticscholar.org/paper/d4f2825f15677fec2a138ac3fc0cbd96e33aa58c|journal=Science|volume=301|issue=5631|pages=336–8|bibcode=2003Sci...301..336C|doi=10.1126/science.1085242|pmid=12869753|vauthors=Carrington JC, Ambros V|s2cid=43395657}}</ref>。遺伝子発現のダウンレギュレーションにおける内因性miRNAの役割は、1993年に''C. elegans''で初めて記載された<ref name="Lee_1993">{{cite journal|date=December 1993|title=The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14|journal=Cell|volume=75|issue=5|pages=843–54|doi=10.1016/0092-8674(93)90529-Y|pmid=8252621|vauthors=Lee RC, Feinbaum RL, Ambros V|doi-access=free}}</ref>。植物では、この機能はシロイヌナズナのmiR-JAWが植物の形状を制御するいくつかの遺伝子の調節に関与していることが示された際に発見された<ref name="Palatnik_2003">{{cite journal|date=September 2003|title=Control of leaf morphogenesis by microRNAs|url=http://edoc.mpg.de/47023|journal=Nature|volume=425|issue=6955|pages=257–63|bibcode=2003Natur.425..257P|doi=10.1038/nature01958|pmid=12931144|vauthors=Palatnik JF, Allen E, Wu X, Schommer C, Schwab R, Carrington JC, Weigel D|s2cid=992057}}</ref>。植物では、miRNAによって調節される遺伝子の大部分は[[転写因子]]である<ref name="Zhang_plant">{{cite journal|date=January 2006|title=Plant microRNA: a small regulatory molecule with big impact|journal=Developmental Biology|volume=289|issue=1|pages=3–16|doi=10.1016/j.ydbio.2005.10.036|pmid=16325172|vauthors=Zhang B, Pan X, Cobb GP, Anderson TA|doi-access=free}}</ref>。そのためmiRNAの活性は特に広範囲にわたり、転写因子や[[Fボックスタンパク質]]など重要な調節遺伝子を調節することで発生時に遺伝子ネットワーク全体を調節する<ref name="Jones-Rhoades">{{cite journal|year=2006|title=MicroRNAS and their regulatory roles in plants|url=https://semanticscholar.org/paper/f636ce0db7e83e6526b27c9101f2038d2e045b5e|journal=Annual Review of Plant Biology|volume=57|pages=19–53|doi=10.1146/annurev.arplant.57.032905.105218|pmid=16669754|vauthors=Jones-Rhoades MW, Bartel DP, Bartel B|s2cid=13010154}}</ref>。ヒトを含む多くの生物では、miRNAは[[腫瘍]]形成や[[細胞周期]]の調節異常と関連づけられている。miRNAは[[がん遺伝子]]としても[[がん抑制遺伝子]]としても機能する<ref name="Zhang_cancer">{{cite journal|date=February 2007|title=microRNAs as oncogenes and tumor suppressors|journal=Developmental Biology|volume=302|issue=1|pages=1–12|doi=10.1016/j.ydbio.2006.08.028|pmid=16989803|vauthors=Zhang B, Pan X, Cobb GP, Anderson TA|doi-access=free}}</ref>。 |
|||
== 進化 == |
|||
[[最大節約法]]による[[系統学]]的解析に基づくと、全真核生物の[[最も近い共通祖先|最近共通祖先]]はすでに原始的なRNAi経路を持っていた可能性が極めて高く、特定の真核生物にRNAi経路が存在しないことは派生形質であると考えられている<ref name="Cerutti">{{cite journal|date=August 2006|title=On the origin and functions of RNA-mediated silencing: from protists to man|journal=Current Genetics|volume=50|issue=2|pages=81–99|doi=10.1007/s00294-006-0078-x|pmid=16691418|pmc=2583075|vauthors=Cerutti H, Casas-Mollano JA}}</ref>。この祖先型のRNAi系は、少なくとも1つのDicer様タンパク質、1つのArgonauteタンパク質、1つの{{仮リンク|Piwi|en|Piwi}}タンパク質、そしてRNA依存性RNAポリメラーゼを含んでいたと考えられ、これらは細胞中で他の役割を果たしていた可能性もある。大規模な[[比較ゲノミクス]]研究でも同様に、真核生物の[[クラウングループ]]はすでにこれらの構成要素を持っており、[[エキソソーム複合体|エキソソーム]]などの一般的なRNA分解系とより密接な機能的関係を持っていた可能性があることが示唆されている<ref name="Anantharaman">{{cite journal|date=April 2002|title=Comparative genomics and evolution of proteins involved in RNA metabolism|journal=Nucleic Acids Research|volume=30|issue=7|pages=1427–64|doi=10.1093/nar/30.7.1427|pmid=11917006|pmc=101826|vauthors=Anantharaman V, Koonin EV, Aravind L}}</ref>。またこの研究は、真核生物、大部分の古細菌、そして少なくとも一部の細菌(''{{仮リンク|Aquifex aeolicus|en|Aquifex aeolicus}}''など)に共通して存在するRNA結合性のArgonauteタンパク質ファミリーは、翻訳開始系の構成要素と相同であり、そしてそこから進化したものであることも示唆している<ref name="Anantharaman" />。 |
|||
== 応用 == |
|||
=== 遺伝子ノックダウンのためのRNAi経路 === |
|||
RNAi経路は実験生物学において、培養細胞やモデル生物における''in vivo''での遺伝子機能の研究のためにしばしば利用される<ref name="Daneholt2006" />。目的の遺伝子に対して相補的な配列を持つ二本鎖RNAが合成され、細胞または個体に導入される。そこで二本鎖RNAは外因性の遺伝子物質と認識され、RNAi経路が活性化される。この機構を用いて標的遺伝子の発現の劇的な低下を引き起こすことが可能であり、そしてこの低下の影響を研究することで遺伝子産物の生理学的役割を示すことができる。RNAiでは遺伝子の発現が完全には抑制されない場合があるため、遺伝子の発現が完全に除去される「[[遺伝子ノックアウト|ノックアウト]]」と区別して、「[[遺伝子ノックダウン|ノックダウン]]」と呼ばれることがある<ref name="pmid12480342">{{cite journal|date=January 2003|title=Knockdown stands up|journal=Trends in Biotechnology|volume=21|issue=1|pages=2–4|doi=10.1016/S0167-7799(02)00002-1|pmid=12480342|vauthors=Voorhoeve PM, Agami R}}</ref>。遺伝子アレイデータを用いたRNAiのサイレンシング効率の検証では、429の独立した実験において失敗率は18.5%であることが示されている<ref name=":14">{{cite journal|date=September 2016|title=Validation of RNAi Silencing Efficiency Using Gene Array Data shows 18.5% Failure Rate across 429 Independent Experiments|journal=Molecular Therapy: Nucleic Acids|volume=5|issue=9|pages=e366|doi=10.1038/mtna.2016.66|pmid=27673562|pmc=5056990|vauthors=Munkácsy G, Sztupinszki Z, Herman P, Bán B, Pénzváltó Z, Szarvas N, Győrffy B}}</ref>。 |
|||
遺伝子ノックダウン効果を最大化しオフターゲット効果を最小化するdsRNAの設計法に関して、[[計算生物学]]では多くの研究がなされている。オフターゲット効果は、導入されたRNAの配列が複数の遺伝子と対合して発現を低下させるために生じる。ヒト、''C. elegans''、''S. pombe''のゲノム研究からは、可能なsiRNA配列のうち約10%で重大なオフターゲット効果が生じる可能性があると推定されている<ref name="Qiu" />。一般的<ref name="Naito_ds">{{cite journal|date=July 2005|title=dsCheck: highly sensitive off-target search software for double-stranded RNA-mediated RNA interference|journal=Nucleic Acids Research|volume=33|issue=Web Server issue|pages=W589–91|doi=10.1093/nar/gki419|pmid=15980542|pmc=1160180|vauthors=Naito Y, Yamada T, Matsumiya T, Ui-Tei K, Saigo K, Morishita S}}</ref><ref name="Henschel">{{cite journal|date=July 2004|title=DEQOR: a web-based tool for the design and quality control of siRNAs|journal=Nucleic Acids Research|volume=32|issue=Web Server issue|pages=W113–20|doi=10.1093/nar/gkh408|pmid=15215362|pmc=441546|vauthors=Henschel A, Buchholz F, Habermann B}}</ref>、哺乳類特異的<ref name="Naito_si">{{cite journal|date=July 2004|title=siDirect: highly effective, target-specific siRNA design software for mammalian RNA interference|journal=Nucleic Acids Research|volume=32|issue=Web Server issue|pages=W124–9|doi=10.1093/nar/gkh442|pmid=15215364|pmc=441580|vauthors=Naito Y, Yamada T, Ui-Tei K, Morishita S, Saigo K}}</ref>、そしてウイルス特異的<ref name="Naito_virus">{{cite journal|date=July 2006|title=siVirus: web-based antiviral siRNA design software for highly divergent viral sequences|journal=Nucleic Acids Research|volume=34|issue=Web Server issue|pages=W448–50|doi=10.1093/nar/gkl214|pmid=16845046|pmc=1538817|vauthors=Naito Y, Ui-Tei K, Nishikawa T, Takebe Y, Saigo K}}</ref>なsiRNAを設計し、自動的に交差反応性のチェックを行うアルゴリズムを備えたソフトウェアツールが多く開発されている。 |
|||
生物種や実験系に応じて、外因性RNAはDicerによって切断されるように設計された長鎖RNAである場合や、siRNA基質として作用するよう設計された短鎖RNAである場合がある。ほとんどの哺乳類細胞では、長い二本鎖RNA分子に対しては外来遺伝物質に非特異的に作用する[[自然免疫系|自然免疫]]の一種である[[インターフェロン]]応答が誘導されるため、短いRNAが利用される<ref name="Reynolds">{{cite journal|date=June 2006|title=Induction of the interferon response by siRNA is cell type- and duplex length-dependent|journal=RNA|volume=12|issue=6|pages=988–93|doi=10.1261/rna.2340906|pmid=16611941|pmc=1464853|vauthors=Reynolds A, Anderson EM, Vermeulen A, Fedorov Y, Robinson K, Leake D, Karpilow J, Marshall WS, Khvorova A}}</ref>。マウスの[[卵母細胞]]や初期[[胚]]は外因性dsRNAに対するこの応答を欠くため、哺乳類の遺伝子ノックダウン効果を研究するための一般的なモデル系となっている<ref name="Stein_oocyte">{{cite journal|date=October 2005|title=Absence of non-specific effects of RNA interference triggered by long double-stranded RNA in mouse oocytes|journal=Developmental Biology|volume=286|issue=2|pages=464–71|doi=10.1016/j.ydbio.2005.08.015|pmid=16154556|vauthors=Stein P, Zeng F, Pan H, Schultz RM|doi-access=free}}</ref>。siRNAが転写される適切な配列をコードしたプラスミドの安定トランスフェクションや<ref name="Brummelkamp">{{cite journal|date=April 2002|title=A system for stable expression of short interfering RNAs in mammalian cells|journal=Science|volume=296|issue=5567|pages=550–3|bibcode=2002Sci...296..550B|doi=10.1126/science.1068999|pmid=11910072|vauthors=Brummelkamp TR, Bernards R, Agami R|s2cid=18460980|author-link2=René Bernards|hdl=1874/15573|hdl-access=free}}</ref>、より精巧な{{仮リンク|レンチウイルス属|en|Lentivirus|label=レンチウイルス}}ベクターシステムによって転写の活性化や不活性化の誘導を可能にしたコンディショナルRNAi(conditional RNAi)と呼ばれる技術など<ref name="Tiscornia">{{cite journal|date=May 2004|title=CRE recombinase-inducible RNA interference mediated by lentiviral vectors|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=101|issue=19|pages=7347–51|bibcode=2004PNAS..101.7347T|doi=10.1073/pnas.0402107101|pmid=15123829|pmc=409921|vauthors=Tiscornia G, Tergaonkar V, Galimi F, Verma IM|doi-access=free}}</ref><ref name="Ventura">{{cite journal|date=July 2004|title=Cre-lox-regulated conditional RNA interference from transgenes|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=101|issue=28|pages=10380–5|bibcode=2004PNAS..10110380V|doi=10.1073/pnas.0403954101|pmid=15240889|pmc=478580|vauthors=Ventura A, Meissner A, Dillon CP, McManus M, Sharp PA, Van Parijs L, Jaenisch R, Jacks T|doi-access=free}}</ref>、siRNAの直接導入を回避することで哺乳類系におけるRNAiの有用性を改善した特殊な実験技術も開発されている。 |
|||
=== 機能ゲノミクス === |
|||
[[File:Drosophila melanogaster - side (aka).jpg|thumb|left|250px|RNAi実験で一般的に利用されるモデル生物であるショウジョウバエの正常な成体]] |
|||
ゲノムワイドRNAiライブラリの設計には、一定の実験条件のセットに対して単一のsiRNAを設計するよりも高度な技術が必要となる場合がある。siRNAライブラリの設計<ref name="Huesken">{{cite journal|date=August 2005|title=Design of a genome-wide siRNA library using an artificial neural network|journal=Nature Biotechnology|volume=23|issue=8|pages=995–1001|doi=10.1038/nbt1118|pmid=16025102|vauthors=Huesken D, Lange J, Mickanin C, Weiler J, Asselbergs F, Warner J, Meloon B, Engel S, Rosenberg A, Cohen D, Labow M, Reinhardt M, Natt F, Hall J|s2cid=11030533}}</ref>や遺伝子ノックダウン時の効率の予測<ref name="Ge">{{cite journal|date=October 2005|title=Prediction of siRNA knockdown efficiency using artificial neural network models|journal=Biochemical and Biophysical Research Communications|volume=336|issue=2|pages=723–8|doi=10.1016/j.bbrc.2005.08.147|pmid=16153609|vauthors=Ge G, Wong GW, Luo B}}</ref>には、[[ニューラルネットワーク]]がよく利用される。マスゲノムスクリーニングは{{仮リンク|DNAアノテーション|en|DNA annotation|label=ゲノムアノテーション}}のための有望な方法として広く知られており、[[マイクロアレイ]]ベースの[[ハイスループットスクリーニング]]の開発の引き金となった<ref name="Janitz">{{cite book|vauthors=Janitz M, Vanhecke D, Lehrach H|volume=173|issue=173|pages=97–104|year=2006|pmid=16594612|doi=10.1007/3-540-27262-3_5|isbn=978-3-540-27261-8|series=Handbook of Experimental Pharmacology|title=RNA Towards Medicine|chapter=High-Throughput RNA Interference in Functional Genomics}}</ref><ref name="Vanhecke">{{cite journal|date=February 2005|title=Functional genomics using high-throughput RNA interference|url=http://edoc.mpg.de/276165|journal=Drug Discovery Today|volume=10|issue=3|pages=205–12|doi=10.1016/S1359-6446(04)03352-5|pmid=15708535|vauthors=Vanhecke D, Janitz M|hdl=11858/00-001M-0000-0010-86E7-8|hdl-access=free}}</ref>。しかし、こうしたスクリーニングの有用性や、モデル生物で開発された技術が近縁種にも一般化できるのか、例えば''C. elegans''の技術が関連する寄生性線虫に応用可能であるのかに関しては疑問視されている<ref name="Geldhof">{{cite journal|date=June 2006|title=Testing the efficacy of RNA interference in Haemonchus contortus|journal=International Journal for Parasitology|volume=36|issue=7|pages=801–10|doi=10.1016/j.ijpara.2005.12.004|pmid=16469321|vauthors=Geldhof P, Murray L, Couthier A, Gilleard JS, McLauchlan G, Knox DP, Britton C}}</ref><ref name="Geldhof2">{{cite journal|date=May 2007|title=RNA interference in parasitic helminths: current situation, potential pitfalls and future prospects|journal=Parasitology|volume=134|issue=Pt 5|pages=609–19|doi=10.1017/S0031182006002071|pmid=17201997|vauthors=Geldhof P, Visser A, Clark D, Saunders G, Britton C, Gilleard J, Berriman M, Knox D|s2cid=27296716}}</ref>。 |
|||
=== 医療 === |
|||
'''医療におけるRNAiの利用の歴史''' |
|||
[[File:Vertical Timeline of RNAi's Use in Medicine.pdf|thumb|upright=1.5|医療におけるRNAiの利用の時系列]] |
|||
動物におけるRNAサイレンシングの最初の例は1996年に記載された。線虫''C. elegans''において、par-1 mRNAのセンス鎖とアンチセンス鎖のRNAを導入することで、par-1 mRNAの分解が引き起こされることが観察された<ref name="Sen">{{cite journal|date=July 2006|title=A brief history of RNAi: the silence of the genes|journal=FASEB Journal|volume=20|issue=9|pages=1293–9|doi=10.1096/fj.06-6014rev|pmid=16816104|vauthors=Sen GL, Blau HM|s2cid=12917676}}</ref>。この分解は一本鎖RNAによって開始されると考えられていたが、その2年後の1998年、ファイアーとメローによってこの''par-1''遺伝子発現のサイレンシング能力は実際には二本鎖RNAによって開始されていることが発見された<ref name="Sen" />。この発見によって、彼らはノーベル生理学・医学賞を受賞した<ref name=":15">{{Cite web |url=https://www.nobelprize.org/nobel_prizes/medicine/laureates/2006/advanced.html |title=The Nobel Prize in Physiology or Medicine 2006 |vauthors=Daneholt B |date=2 October 2006 |website=nobelprize.org |access-date=30 October 2017}}</ref>。この画期的な発見の直後、合成siRNAを用いることで、サイレンシングの標的は遺伝子全体ではなく遺伝子中の特定の配列とすることが可能であることが発見された<ref name=":16">{{cite journal|year=2001|title=Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells|journal=Nature|volume=411|issue=6836|pages=494–498|bibcode=2001Natur.411..494E|doi=10.1038/35078107|pmid=11373684|vauthors=Elbashir S, Harborth J, Lendeckel W et al.|s2cid=710341}}</ref>。そのわずか1年後、トランスジェニックマウスにおいて[[C型肝炎ウイルス]]配列を標的として、この配列特異的なサイレンシングの治療応用が実証された<ref name=":17">{{cite journal|date=July 2002|title=RNA interference in adult mice|journal=Nature|volume=418|issue=6893|pages=38–9|bibcode=2002Natur.418...38M|doi=10.1038/418038a|pmid=12097900|vauthors=McCaffrey AP, Meuse L, Pham TT, Conklin DS, Hannon GJ, Kay MA|s2cid=4361399|author-link5=Gregory Hannon}} {{closed access}}</ref>。それ以降、RNAiの治療応用を広げる試みが多くの研究者によってなされ、具体的にはさまざまなタイプのがんを引き起こす遺伝子を標的とすることに関心が寄せられた<ref name=":18">{{cite journal|date=September 2006|title=siRNA-based approaches in cancer therapy|journal=Cancer Gene Therapy|volume=13|issue=9|pages=819–29|doi=10.1038/sj.cgt.7700931|pmid=16424918|vauthors=Devi GR|doi-access=free}}</ref><ref name=":19">{{cite journal|date=October 2003|title=Small RNA: can RNA interference be exploited for therapy?|journal=Lancet|volume=362|issue=9393|pages=1401–3|doi=10.1016/s0140-6736(03)14637-5|pmid=14585643|vauthors=Wall NR, Shi Y|s2cid=25034627}}</ref>。2006年までに臨床試験に到達した最初の応用は、[[加齢黄斑変性|黄斑変性]]と[[RSウイルス]]の治療であった<ref name=":20">{{cite journal|author=Sah D|year=2006|title=Therapeutic potential of RNA interference for neurological disorders|journal=Life Sci|volume=79|issue=19|pages=1773–80|doi=10.1016/j.lfs.2006.06.011|pmid=16815477}}</ref>。その4年後、ナノ粒子デリバリーシステムを用いて、固形腫瘍を標的としたヒトでの初めての第I相臨床試験が開始された<ref name=":21">{{cite journal|date=April 2010|title=Evidence of RNAi in humans from systemically administered siRNA via targeted nanoparticles|journal=Nature|volume=464|issue=7291|pages=1067–70|bibcode=2010Natur.464.1067D|doi=10.1038/nature08956|pmid=20305636|pmc=2855406|vauthors=Davis ME, Zuckerman JE, Choi CH, Seligson D, Tolcher A, Alabi CA, Yen Y, Heidel JD, Ribas A}}</ref>。現在のところ大部分の研究はがん治療へのRNAiの応用を試みるものであるが、可能な応用は広範囲にわたる。RNAiは、[[ウイルス]]<ref name=":22">{{cite journal|date=November 2001|title=RNAi-mediated oncogene silencing confers resistance to crown gall tumorigenesis|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=98|issue=23|pages=13437–42|bibcode=2001PNAS...9813437E|doi=10.1073/pnas.241276898|pmid=11687652|pmc=60889|vauthors=Escobar MA, Civerolo EL, Summerfelt KR, Dandekar AM|doi-access=free}}</ref>、[[細菌]]<ref name=":23">{{cite journal|date=April 2008|title=Schistosoma mansoni: evaluation of an RNAi-based treatment targeting HGPRTase gene|journal=Experimental Parasitology|volume=118|issue=4|pages=619–23|doi=10.1016/j.exppara.2007.11.017|pmid=18237732|vauthors=Pereira TC, Pascoal VD, Marchesini RB, Maia IG, Magalhães LA, Zanotti-Magalhães EM, Lopes-Cendes I}}</ref>、[[寄生虫]]による感染症<ref name=":24">{{cite journal|date=April 2005|title=Lentiviral-mediated silencing of SOD1 through RNA interference retards disease onset and progression in a mouse model of ALS|url=http://www.hal.inserm.fr/inserm-00484530|journal=Nature Medicine|volume=11|issue=4|pages=423–8|doi=10.1038/nm1207|pmid=15768028|vauthors=Raoul C, Abbas-Terki T, Bensadoun JC, Guillot S, Haase G, Szulc J, Henderson CE, Aebischer P|s2cid=25445264}}</ref>、不適応な遺伝子変異の治療<ref name=":25">{{cite journal|date=November 2004|title=Short interfering RNA (siRNA) targeting the Lyn kinase induces apoptosis in primary, and drug-resistant, BCR-ABL1(+) leukemia cells|journal=Nature Medicine|volume=10|issue=11|pages=1187–9|doi=10.1038/nm1127|pmid=15502840|vauthors=Ptasznik A, Nakata Y, Kalota A, Emerson SG, Gewirtz AM|s2cid=21770360}}</ref>、[[薬物乱用|薬物使用]]の制御<ref name=":26">{{cite journal|date=November 2009|title=Effective relief of neuropathic pain by adeno-associated virus-mediated expression of a small hairpin RNA against GTP cyclohydrolase 1|journal=Molecular Pain|volume=5|pages=1744–8069–5–67|doi=10.1186/1744-8069-5-67|pmid=19922668|pmc=2785765|vauthors=Kim SJ, Lee WI, Lee YS, Kim DH, Chang JW, Kim SW, Lee H}}</ref>、[[疼痛]]管理<ref name=":27">{{cite journal|date=November 2010|title=Knockdown of orexin type 1 receptor in rat locus coeruleus increases REM sleep during the dark period|journal=The European Journal of Neuroscience|volume=32|issue=9|pages=1528–36|doi=10.1111/j.1460-9568.2010.07401.x|pmid=21089218|pmc=3058252|vauthors=Chen L, McKenna JT, Bolortuya Y, Winston S, Thakkar MM, Basheer R, Brown RE, McCarley RW}}</ref>、さらには睡眠の調節<ref name=":28">{{cite journal|date=December 2003|title=Size selective recognition of siRNA by an RNA silencing suppressor|journal=Cell|volume=115|issue=7|pages=799–811|doi=10.1016/s0092-8674(03)00984-x|pmid=14697199|vauthors=Vargason JM, Szittya G, Burgyán J, Hall TM|doi-access=free}}</ref>にも利用できる可能性がある。 |
|||
==== 治療応用 ==== |
|||
===== ウイルス感染 ===== |
|||
抗ウイルス治療はRNAiベースの医療応用として最初期に提唱されたものであり、2つの異なる種類のものが開発されている。1つはウイルスのRNAを標的とするものである。ウイルスのRNAを標的化することで、[[ヒト免疫不全ウイルス|HIV]]<ref name=":29">{{cite journal|date=April 2004|title=RNA interference as an antiviral approach: targeting HIV-1|journal=Current Opinion in Molecular Therapeutics|volume=6|issue=2|pages=141–5|pmid=15195925|vauthors=Berkhout B}}</ref>、[[ヒトパピローマウイルス|HPV]]<ref name=":30">{{cite journal|date=September 2002|title=Selective silencing of viral gene expression in HPV-positive human cervical carcinoma cells treated with siRNA, a primer of RNA interference|journal=Oncogene|volume=21|issue=39|pages=6041–8|doi=10.1038/sj.onc.1205878|pmid=12203116|vauthors=Jiang M, Milner J|doi-access=free}}</ref>、[[A型肝炎ウイルス]]<ref name=":31">{{cite journal|date=June 2006|title=Silencing of hepatitis A virus infection by small interfering RNAs|journal=Journal of Virology|volume=80|issue=11|pages=5599–610|doi=10.1128/jvi.01773-05|pmid=16699041|pmc=1472172|vauthors=Kusov Y, Kanda T, Palmenberg A, Sgro JY, Gauss-Müller V}}</ref>、[[B型肝炎ウイルス]]<ref name=":32">{{cite journal|date=October 2006|title=A retrovirus-based system to stably silence hepatitis B virus genes by RNA interference|journal=Biotechnology Letters|volume=28|issue=20|pages=1679–85|doi=10.1007/s10529-006-9138-z|pmid=16900331|vauthors=Jia F, Zhang YZ, Liu CM|s2cid=34511611}}</ref>、[[インフルエンザウイルス]]<ref name=":33">{{cite journal|date=December 2005|title=Construction of influenza virus siRNA expression vectors and their inhibitory effects on multiplication of influenza virus|journal=Avian Diseases|volume=49|issue=4|pages=562–73|doi=10.1637/7365-041205R2.1|pmid=16405000|vauthors=Li YC, Kong LH, Cheng BZ, Li KS|s2cid=86214047}}</ref><ref name=":34">{{cite journal|date=2015|title=Gene silencing: a therapeutic approach to combat influenza virus infections|journal=Future Microbiology|volume=10|issue=1|pages=131–40|doi=10.2217/fmb.14.94|pmid=25598342|vauthors=Khanna M, Saxena L, Rajput R, Kumar B, Prasad R}}</ref><ref name=":35">{{cite journal|date=December 2012|title=Small interfering RNA targeting the nonstructural gene 1 transcript inhibits influenza A virus replication in experimental mice|journal=Nucleic Acid Therapeutics|volume=22|issue=6|pages=414–22|doi=10.1089/nat.2012.0359|pmid=23062009|vauthors=Rajput R, Khanna M, Kumar P, Kumar B, Sharma S, Gupta N, Saxena L}}</ref>、[[RSウイルス]]<ref name=":0">{{cite journal|date=December 2018|title=Advancements in Nucleic Acid Based Therapeutics against Respiratory Viral Infections|journal=Journal of Clinical Medicine|volume=8|issue=1|pages=6|doi=10.3390/jcm8010006|pmid=30577479|pmc=6351902|vauthors=Asha K, Kumar P, Sanicas M, Meseko CA, Khanna M, Kumar B|doi-access=free}}</ref>、[[SARSコロナウイルス]]<ref name=":0" />、[[アデノウイルス]]<ref name=":0" />、[[麻疹ウイルス]]<ref name=":36">{{cite journal|year=2005|title=Inhibition of Measles virus multiplication in cell culture by RNA interference|journal=Acta Virologica|volume=49|issue=4|pages=227–34|pmid=16402679|vauthors=Hu L, Wang Z, Hu C, Liu X, Yao L, Li W, Qi Y}}</ref>など、多数のウイルスの複製を抑制できることが多くの研究により示されている。もう1つの戦略は、宿主細胞の遺伝子を標的とすることでウイルスの侵入を防ぐものである<ref name=":37">{{cite journal|year=2014|title=VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets|journal=Database|volume=2014|doi=10.1093/database/bau103|pmid=25380780|pmc=4224276|vauthors=Qureshi A, Thakur N, Monga I, Thakur A, Kumar M}}</ref>。たとえば、宿主細胞の{{仮リンク|ケモカイン受容体|en|Chemokine receptor}}({{仮リンク|CXCR4|en|CXCR4}}と[[CCR5]])を抑制することでHIVの侵入を防ぐことができる<ref name=":38">{{cite journal|year=2003|title=Suppression of chemokine receptor expression by RNA interference allows for inhibition of HIV-1 replication, by Martínez et al|journal=AIDS|volume=17 Suppl 4|pages=S103–5|pmid=15080188|vauthors=Crowe S}}</ref>。 |
|||
===== がん ===== |
|||
伝統的な[[化学療法 (悪性腫瘍)|化学療法]]はがん細胞を効果的に殺すことができるが、正常細胞とがん細胞を区別する特異性を欠くために通常は重大な副作用を伴う。RNAiはがんと関連した遺伝子([[がん遺伝子]]など)を標的とすることで腫瘍の成長を阻害する、より特異的なアプローチとなることが多くの研究で示されている<ref name=":39">{{cite journal|date=August 2004|title=Silencing of disease-related genes by small interfering RNAs|journal=Current Molecular Medicine|volume=4|issue=5|pages=507–17|doi=10.2174/1566524043360492|pmid=15267222|vauthors=Fuchs U, Damm-Welk C, Borkhardt A}}</ref>。RNAiはがん細胞の化学療法薬に対する感受性を高めることも可能であり、化学療法との併用療法も提唱されている<ref name=":40">{{cite journal|date=February 2003|title=RNA interference is a functional pathway with therapeutic potential in human myeloid leukemia cell lines|journal=Cancer Gene Therapy|volume=10|issue=2|pages=125–33|doi=10.1038/sj.cgt.7700544|pmid=12536201|vauthors=Cioca DP, Aoki Y, Kiyosawa K|doi-access=free}}</ref>。細胞の浸潤や[[遊走]]の阻害も、RNAiベースの他の治療法となる可能性がある<ref name=":41">{{cite journal|date=January 2005|title=CXCR4 knockdown by small interfering RNA abrogates breast tumor growth in vivo|journal=Cancer Gene Therapy|volume=12|issue=1|pages=84–9|doi=10.1038/sj.cgt.7700770|pmid=15472715|vauthors=Lapteva N, Yang AG, Sanders DE, Strube RW, Chen SY|doi-access=free}}</ref>。 |
|||
===== 神経疾患 ===== |
|||
RNAiは[[神経変性疾患]]の治療法となる可能性が示されている。細胞やマウスでの研究では、[[アミロイドβ]](Aβ)を産生する遺伝子([[Β-セクレターゼ1|''BACE1'']]や[[アミロイド前駆体タンパク質|''APP'']]など)をRNAiで特異的に標的化することで、[[アルツハイマー病]]と関係するAβペプチドの量を大きく低下させることができることが示されている<ref name=":42">{{cite journal|date=October 2005|title=Targeting BACE1 with siRNAs ameliorates Alzheimer disease neuropathology in a transgenic model|journal=Nature Neuroscience|volume=8|issue=10|pages=1343–9|doi=10.1038/nn1531|pmid=16136043|vauthors=Singer O, Marr RA, Rockenstein E, Crews L, Coufal NG, Gage FH, Verma IM, Masliah E|s2cid=6978101}}</ref><ref name=":43">{{cite journal|date=September 2009|title=Allele-specific RNAi mitigates phenotypic progression in a transgenic model of Alzheimer's disease|journal=Molecular Therapy|volume=17|issue=9|pages=1563–73|doi=10.1038/mt.2009.123|pmid=19532137|pmc=2835271|vauthors=Rodríguez-Lebrón E, Gouvion CM, Moore SA, Davidson BL, Paulson HL}}</ref><ref name=":44">{{cite journal|date=October 2010|title=Silencing of CDK5 reduces neurofibrillary tangles in transgenic alzheimer's mice|journal=The Journal of Neuroscience|volume=30|issue=42|pages=13966–76|doi=10.1523/jneurosci.3637-10.2010|pmid=20962218|pmc=3003593|vauthors=Piedrahita D, Hernández I, López-Tobón A, Fedorov D, Obara B, Manjunath BS, Boudreau RL, Davidson B, Laferla F, Gallego-Gómez JC, Kosik KS, Cardona-Gómez GP}}</ref>。さらに、こうしたサイレンシングベースのアプローチは、[[パーキンソン病]]や{{仮リンク|トリプレットリピート病|en|Trinucleotide repeat disorder|label=ポリグルタミン病}}の治療においても有望な結果をもたらしている<ref name=":45">{{cite journal|date=March 2006|title=Viral-based modelling and correction of neurodegenerative diseases by RNA interference|journal=Gene Therapy|volume=13|issue=6|pages=487–95|doi=10.1038/sj.gt.3302690|pmid=16319945|vauthors=Raoul C, Barker SD, Aebischer P|doi-access=free}}</ref><ref name=":46">{{cite journal|date=April 2005|title=RNA interference improves motor and neuropathological abnormalities in a Huntington's disease mouse model|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=102|issue=16|pages=5820–5|bibcode=2005PNAS..102.5820H|doi=10.1073/pnas.0501507102|pmid=15811941|pmc=556303|vauthors=Harper SQ, Staber PD, He X, Eliason SL, Martins IH, Mao Q, Yang L, Kotin RM, Paulson HL, Davidson BL|doi-access=free}}</ref><ref name=":47">{{cite journal|date=April 2011|title=RNAi medicine for the brain: progresses and challenges|journal=Human Molecular Genetics|volume=20|issue=R1|pages=R21–7|doi=10.1093/hmg/ddr137|pmid=21459775|pmc=3095054|vauthors=Boudreau RL, Rodríguez-Lebrón E, Davidson BL}}</ref>。 |
|||
==== 治療応用における困難 ==== |
|||
RNAiの臨床的可能性の実現のためには、siRNAが効率的に標的組織の細胞へ輸送される必要がある。しかしながら、臨床利用までに克服すべきさまざまな障壁が存在する。例えば、「裸」のsiRNAはその治療効力を低下させるいくつかの障害の影響を受けやすい<ref name="Kanasty nmat3765">{{cite journal|date=November 2013|title=Delivery materials for siRNA therapeutics|journal=Nature Materials|volume=12|issue=11|pages=967–77|bibcode=2013NatMa..12..967K|doi=10.1038/nmat3765|pmid=24150415|vauthors=Kanasty R, Dorkin JR, Vegas A, Anderson D}}</ref>。いったんsiRNAが血流に移行すると、裸のRNAは血清中の[[ヌクレアーゼ]]によって分解されたり、自然免疫系を刺激したりする<ref name="Kanasty nmat3765" />。また、そのサイズと高い[[アニオン]]性のため、未修飾のsiRNA分子が[[細胞膜]]を通って細胞内へ移行するのは容易ではない。そのため、人工的なsiRNAやナノ粒子に封入したsiRNAを利用する必要がある。しかしながら、細胞膜を越えたsiRNAの輸送にはさらに固有の問題が存在する。siRNAが細胞膜を越えて輸送された場合、その量が最適化されていなければ意図しない毒性が生じたり、オフターゲット効果(部分的な配列相補性を持つ遺伝子に対する意図しないダウンレギュレーションなど)が生じたりする可能性がある<ref name="ReferenceA">{{cite journal|date=September 2015|title=Knocking down disease: a progress report on siRNA therapeutics|journal=Nature Reviews Genetics|volume=16|issue=9|pages=543–52|doi=10.1038/nrg3978|pmid=26281785|pmc=4756474|vauthors=Wittrup A, Lieberman J}}</ref>。細胞に移行した後も、その効果は細胞分裂ごとに希釈されるため繰り返し投与が必要である。また、dsRNAを運搬するベクターの一部には調節作用がある場合があるため、非特異的な副作用も考慮し、制御される必要がある<ref name=":48">{{cite journal|date=May 2019|title=RNA interference may result in unexpected phenotypes in Caenorhabditis elegans|journal=Nucleic Acids Research|volume=47|issue=8|pages=3957–3969|doi=10.1093/nar/gkz154|pmid=30838421|pmc=6486631|vauthors=De-Souza EA, Camara H, Salgueiro WG, Moro RP, Knittel TL, Tonon G, Pinto S, Pinca AP, Antebi A, Pasquinelli AE, Massirer KB, Mori MA|display-authors=6}}</ref>。 |
|||
==== 免疫応答の刺激 ==== |
|||
ヒトの免疫系は、[[自然免疫系]]と[[獲得免疫系]]の2種類に分類される<ref name="Whitehead 2011 77–96">{{cite journal|date=2011|title=Silencing or stimulation? siRNA delivery and the immune system|url=https://semanticscholar.org/paper/be05a74656a866c8992589e85c1dba391e8373df|journal=Annual Review of Chemical and Biomolecular Engineering|volume=2|pages=77–96|doi=10.1146/annurev-chembioeng-061010-114133|pmid=22432611|vauthors=Whitehead KA, Dahlman JE, Langer RS, Anderson DG|s2cid=28803811}}</ref>。自然免疫系は感染に対する第一の防御機構であり、病原体に対して一般的応答を行う<ref name="Whitehead 2011 77–96" />。一方、獲得免疫系は自然免疫系よりも後で進化した系であり、病原体の分子の特定の部分に反応するよう訓練された、高度に専門化された[[B細胞]]と[[T細胞]]によって構成される<ref name="Whitehead 2011 77–96" />。 |
|||
siRNAは自然免疫系によって制御されおり、自然免疫系による応答はさらに急性炎症応答と抗ウイルス応答に分類される<ref name="Whitehead 2011 77–96" />。炎症応答では、低分子シグナル伝達分子や[[インターロイキン-1|IL-1]]、[[インターロイキン-6|IL-6]]、[[インターロイキン-12|IL-12]]、[[TNF-α]]などの[[サイトカイン]]が誘導される。こうした炎症性サイトカインは[[食作用]]を刺激し、侵入した病原体を破壊する<ref name="Whitehead 2011 77–96" />。抗ウイルス応答では、IFN-αやIFN-βなどのタイプI[[インターフェロン]]の放出や抗ウイルス遺伝子のアップレギュレーションが誘導される<ref name="Whitehead 2011 77–96" />。どちらの応答も、[[パターン認識受容体]](PRR)の刺激を介して引き起こされる。複数のPRRによってRNA構造のさまざまな側面が認識されるため、免疫刺激を避けることは困難なものとなっている<ref name="Whitehead 2011 77–96" />。 |
|||
==== 治療技術としての展望 ==== |
|||
2015年から2017年にかけて行われたsiRNA治療の第I・II相試験では、肝臓での強力かつ持続的な遺伝子ノックダウン効果と臨床効果を示す一部の徴候がみられ、許容できない毒性はみられなかった<ref name="ReferenceA" />。[[トランスサイレチン]]の変異によって引き起こされる家族性神経変性・心筋症の治療へ向けた2つの第III相試験が進行中である<ref name="ReferenceA" />。多くの研究で''in vivo''デリバリーシステムの有望性は示されており、またそれらの多様な特性は無数の応用を可能にしている。最も有望なものはナノ粒子デリバリーシステムであるが、製品の安定した品質を確保するためには厳密に制御された混合過程が必要となることなど、製造過程のスケールアップにはさらなる課題が残されている<ref name="Kanasty nmat3765" />。 |
|||
=== バイオテクノロジー === |
|||
RNAiは[[バイオテクノロジー]]分野で応用されており、他の分野でも商業化が近い。[[ニコチン]]を含まないタバコ、[[カフェイン]]を含まないコーヒー、栄養素を強化した野菜、低[[アレルゲン]]の作物など、RNAiを利用した新たな作物が発されている。遺伝子改変されたリンゴ{{仮リンク|Arctic Apples|en|Arctic Apples}}は2015年にFDAの承認を受けた<ref name=":49">{{cite news|title=FDA concludes Arctic Apples and Innate Potatoes are safe for consumption|url=https://www.fda.gov/newsevents/newsroom/pressannouncements/ucm439121.htm|access-date=29 September 2017}}</ref>。このリンゴはPPO({{仮リンク|ポリフェノールオキシダーゼ|en|Polyphenol oxidase}})遺伝子をRNAiによって抑制することで、果実を切った後の褐変が起こらないようになっている。PPOがサイレンシングされたリンゴは、[[クロロゲン酸]]を標準的な[[キノン]]産物へと変換することができないため、変色が起こらない<ref name="Saurabh Satyajit, Vidyarthi AS, Prasad D 2014 543–564" />。 |
|||
[[作物学]]におけるRNAiの応用には、ストレス耐性の付与や栄養素の強化などの改善などいくつかの可能性がある。RNAiはC3植物の生産性の向上のための光呼吸の阻害のほか、早期の開花、成熟や老化の遅れ、休眠の解除、ストレスに強い植物、自家不和合性の克服などの誘導に有用である可能性がある<ref name="Saurabh Satyajit, Vidyarthi AS, Prasad D 2014 543–564" />。 |
|||
==== 食品 ==== |
|||
RNAiは食料生産における将来的有望性が示されているが、まだ若い技術であるためその利点と欠点に対する理解に欠けるところがある。そのため、よりよく理解し誤解を取り除く必要がある<ref name="Herrero-et-al-2020"> |
|||
{{cite journal|last1=Herrero|first1=Mario|last3=Thornton|first3=Philip K.|last5=Mason-D’Croz|first5=Daniel|last7=Palmer|first7=Jeda|last8=Benton|first8=Tim G.|year=2020|title=Innovation can accelerate the transition towards a sustainable food system|url=http://eprints.whiterose.ac.uk/163942/1/Herrero_Perspective_Accepted.pdf|journal=Nature Food|volume=1|issue=5|pages=266–272|publisher=Nature Portfolio|doi=10.1038/s43016-020-0074-1|issn=2662-1355|author26=([[ORCID]] [http://orcid.org/0000-0002-1112-1954 0000-0002-1112-1954])|author9=([[ORCID]] [http://orcid.org/0000-0002-7448-1973 0000-0002-7448-1973])|last10=Bodirsky|first10=Benjamin L.|author11=([[ORCID]] [http://orcid.org/0000-0002-8242-6712 0000-0002-8242-6712])|last12=Bogard|first12=Jessica R.|last16=Lee|author13=([[ORCID]] [http://orcid.org/0000-0001-5503-5284 0000-0001-5503-5284])|last14=Hall|first14=Andrew|author15=([[ORCID]] [http://orcid.org/0000-0002-8580-6569 0000-0002-8580-6569])|first21=Graham D.|first16=Bernice|last17=Nyborg|last27=Clark|first17=Karine|last19=Pradhan|first19=Prajal|author20=([[ORCID]] [http://orcid.org/0000-0003-0491-5489 0000-0003-0491-5489])|last21=Bonnett|author44=([[ORCID]] [http://orcid.org/0000-0003-2690-0763 0000-0003-2690-0763])|last22=Bryan|first22=Brett A.|author23=([[ORCID]] [http://orcid.org/0000-0003-4834-5641 0000-0003-4834-5641])|first27=Michael|last39=Havlik|first36=James S.|first35=Cecile M.|last29=Cook|first29=Mathew T.|last30=de Boer|first30=Imke J. M.|last31=Downs|first31=Chris|last32=Dizyee|first32=Kanar|last33=Folberth|first33=Christian|author34=([[ORCID]] [http://orcid.org/0000-0002-6738-5238 0000-0002-6738-5238])|last35=Godde|author28=([[ORCID]] [http://orcid.org/0000-0001-7161-7751 0000-0001-7161-7751])|first43=Ana Maria|last36=Gerber|author37=([[ORCID]] [http://orcid.org/0000-0002-6890-0481 0000-0002-6890-0481])|last38=Grundy|first38=Michael|first24=Bruce M.|first39=Petr|last40=Jarvis|author4=([[ORCID]] [http://orcid.org/0000-0002-1854-0182 0000-0002-1854-0182])|last41=King|first41=Richard|author42=([[ORCID]] [http://orcid.org/0000-0001-6404-8052 0000-0001-6404-8052])|last43=Loboguerrero|last24=Campbell|author65=([[ORCID]] [http://orcid.org/0000-0002-6118-745X 0000-0002-6118-745X])|last25=Christensen|author53=([[ORCID]] [http://orcid.org/0000-0003-1351-138X 0000-0003-1351-138X])|last47=McIntyre|first47=C. Lynne|last48=Naylor|first48=Rosamond|last49=Navarro|first49=Javier|last50=Obersteiner|first60=Pete|author51=([[ORCID]] [http://orcid.org/0000-0001-6981-2769 0000-0001-6981-2769])|last52=Parodi|first52=Alejandro|first50=Michael|author63=([[ORCID]] [http://orcid.org/0000-0003-3016-2679 0000-0003-3016-2679])|first54=Mark B.|first59=Michael J.|last59=Robertson|last54=Peoples|first58=Johan|last58=Rockström|author6=([[ORCID]] [http://orcid.org/0000-0003-0673-2301 0000-0003-0673-2301])|last57=Popp|author56=([[ORCID]] [http://orcid.org/0000-0002-1820-9983 0000-0002-1820-9983])|first55=Ilje|last55=Pikaar|first57=Alexander|last60=Smith|author61=([[ORCID]] [http://orcid.org/0000-0002-3784-1124 0000-0002-3784-1124])|first25=Svend|first68=Mark|author18=([[ORCID]] [http://orcid.org/0000-0002-0359-548X 0000-0002-0359-548X])|author-link8=Tim G. Benton|last45=Lopes|author46=([[ORCID]] [http://orcid.org/0000-0003-0671-9940 0000-0003-0671-9940])|first62=Elke|first45=Mauricio A.|last64=Swain|first64=Steve M.|last66=Valin|first66=Hugo|author67=([[ORCID]] [http://orcid.org/0000-0002-0618-773X 0000-0002-0618-773X])|last68=van Wijk|last69=van Zanten|author70=([[ORCID]] [http://orcid.org/0000-0002-5262-5518 0000-0002-5262-5518])|last62=Stehfest|first69=Hannah H. E.|last71=Vermeulen|first71=Sonja|last72=Vervoort|first72=Joost|last73=West|first73=Paul C.|author74=([[ORCID]] [http://orcid.org/0000-0001-9024-1657 0000-0001-9024-1657])|author2=([[ORCID]] [http://orcid.org/0000-0002-7741-5090 0000-0002-7741-5090])|author-link1=Mario Herrero|s2cid=216337248|first40=Andrew}} |
|||
</ref>。RNAiはすでに、天然毒素の産生が少ない遺伝子組み換え植物に利用されている。こうした技術は、植物においてRNAiの表現型が安定かつ遺伝性のものであることを利用している。[[ワタ属|ワタ]]の種子はタンパク質に富むが、有毒[[テルペノイド]]である[[ゴシポール]]を含むためヒトの食物としては適さない。ゴシポール自体は害虫による損傷から植物を守るために重要であるため、植物の他の部分に影響を与えることなく種子でのみゴシポールの産生に重要な酵素{{仮リンク|δ-カジネンシンターゼ|en|Delta-cadinene synthase}}を減少させるためにRNAiが利用されている<ref name="Sunilkumar">{{cite journal|date=November 2006|title=Engineering cottonseed for use in human nutrition by tissue-specific reduction of toxic gossypol|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=103|issue=48|pages=18054–9|doi=10.1073/pnas.0605389103|pmid=17110445|pmc=1838705|vauthors=Sunilkumar G, Campbell LM, Puckhaber L, Stipanovic RD, Rathore KS|doi-access=free}}</ref>。同様に、[[キャッサバ]]で[[シアン化物]]の原料となる[[リナマリン]]を減少させる試みも行われている<ref name="Siritunga">{{cite journal|date=July 2003|title=Generation of cyanogen-free transgenic cassava|journal=Planta|volume=217|issue=3|pages=367–73|doi=10.1007/s00425-003-1005-8|pmid=14520563|vauthors=Siritunga D, Sayre RT|s2cid=13561249}}</ref>。 |
|||
トマト類ではアレルゲンの減少<ref name="Le_tomato">{{cite journal|date=March 2006|title=Design of tomato fruits with reduced allergenicity by dsRNAi-mediated inhibition of ns-LTP (Lyc e 3) expression|journal=Plant Biotechnology Journal|volume=4|issue=2|pages=231–42|doi=10.1111/j.1467-7652.2005.00175.x|pmid=17177799|vauthors=Le LQ, Lorenz Y, Scheurer S, Fötisch K, Enrique E, Bartra J, Biemelt S, Vieths S, Sonnewald U}}</ref>や[[抗酸化物質]]の強化<ref name="Niggeweg">{{cite journal|date=June 2004|title=Engineering plants with increased levels of the antioxidant chlorogenic acid|journal=Nature Biotechnology|volume=22|issue=6|pages=746–54|doi=10.1038/nbt966|pmid=15107863|vauthors=Niggeweg R, Michael AJ, Martin C|s2cid=21588259}}</ref>に成功している。[[Flavr Savr]]トマトや{{仮リンク|パパイヤリングスポットウイルス|en|Papaya ringspot virus}}耐性[[パパイア|パパイヤ]]の2品種など、これまで商業化されている品種はもともと[[アンチセンスRNA|アンチセンス]]技術を用いて開発されたものであるが、実際にはRNAi経路が利用されている可能性が高い<ref name="Sanders">{{cite journal|date=March 2005|title=Tomato transgene structure and silencing|journal=Nature Biotechnology|volume=23|issue=3|pages=287–9|doi=10.1038/nbt0305-287b|pmid=15765076|vauthors=Sanders RA, Hiatt W|s2cid=21191589}}</ref><ref name="Chiang">{{cite journal|date=November 2001|title=Comparative reactions of recombinant papaya ringspot viruses with chimeric coat protein (CP) genes and wild-type viruses on CP-transgenic papaya|journal=The Journal of General Virology|volume=82|issue=Pt 11|pages=2827–36|doi=10.1099/0022-1317-82-11-2827|pmid=11602796|vauthors=Chiang CH, Wang JJ, Jan FJ, Yeh SD, Gonsalves D|s2cid=25659570|doi-access=free}}</ref>。{{仮リンク|アスペルギルス・フラバス|en|Aspergillus flavus}}''Aspergillus flavus''の{{仮リンク|α-アミラーゼ|en|Alpha-amylase|redirect=1}}を標的としたRNAiによるサイレンシングは[[トウモロコシ]]内でのこの菌類の増殖を低下せ、穀物を危険な[[アフラトキシン]]による汚染から防ぐために利用されている<ref name=":50">{{cite journal|date=June 2018|title=RNA interference-based silencing of the alpha-amylase (amy1) gene in Aspergillus flavus decreases fungal growth and aflatoxin production in maize kernels|journal=Planta|volume=247|issue=6|pages=1465–1473|doi=10.1007/s00425-018-2875-0|pmid=29541880|vauthors=Gilbert MK, Majumdar R, Rajasekaran K, Chen ZY, Wei Q, Sickler CM, Lebar MD, Cary JW, Frame BR, Wang K|s2cid=3918937}}</ref>。[[タマネギ]]での催涙因子合成酵素(lachrymatory factor synthase)のサイレンシングは切っても[[涙の出ないタマネギ]]の生産に、[[セイヨウアブラナ|アブラナ]]での''BP1''遺伝子のサイレンシングは[[光合成]]の改善に利用されている<ref name=":51">{{cite journal|date=March 2013|title=RNA interference: a promising technique for the improvement of traditional crops|journal=International Journal of Food Sciences and Nutrition|volume=64|issue=2|pages=248–59|doi=10.3109/09637486.2012.713918|pmid=22861122|vauthors=Katoch R, Thakur N|s2cid=45212581}}</ref>。[[コムギ]]では、[[アミロース]]含量の増大を目的として''SBEIIa''、''SBEIIb''遺伝子が標的となっているほか<ref name=":52">{{cite journal|date=March 2013|title=Advances in RNA interference technology and its impact on nutritional improvement, disease and insect control in plants|journal=Applied Biochemistry and Biotechnology|volume=169|issue=5|pages=1579–605|doi=10.1007/s12010-012-0046-5|pmid=23322250|vauthors=Katoch R, Thakur N|s2cid=23733295}}</ref>、六倍体品種の[[機能ゲノミクス]]研究にRNAiが、そして''Lr21''遺伝子によってもたらされる{{仮リンク|コムギ赤さび病|en|Wheat leaf rust}}耐性機構の研究のためにvirus-induced gene silencing(VIGS、RNAiの一種)が利用されている<ref name="Downie-et-al-2021">{{cite journal|last1=Downie|first1=Rowena C.|last2=Lin|first2=Min|last3=Corsi|first3=Beatrice|last4=Ficke|first4=Andrea|last5=Lillemo|first5=Morten|last6=Oliver|first6=Richard P.|last7=Phan|first7=Huyen T. T.|last8=Tan|first8=Kar-Chun|last9=Cockram|first9=James|date=2021-07-27|title=Septoria Nodorum Blotch of Wheat: Disease Management and Resistance Breeding in the Face of Shifting Disease Dynamics and a Changing Environment|journal=Phytopathology|volume=111|issue=6|pages=PHYTO–07–20–028|publisher=American Phytopathological Society|doi=10.1094/phyto-07-20-0280-rvw|issn=0031-949X|pmid=33245254|hdl=20.500.11937/83208|s2cid=227181536|hdl-access=free}}</ref>。 |
|||
==== その他の作物 ==== |
|||
[[タバコ]]では[[発癌性|発がん性]]を有する可能性が高い物質の前駆体を減少させる取り組みが行われている<ref name="Gavilano">{{cite journal|date=November 2006|title=Genetic engineering of Nicotiana tabacum for reduced nornicotine content|journal=Journal of Agricultural and Food Chemistry|volume=54|issue=24|pages=9071–8|doi=10.1021/jf0610458|pmid=17117792|vauthors=Gavilano LB, Coleman NP, Burnley LE, Bowman ML, Kalengamaliro NE, Hayes A, Bush L, Siminszky B}}</ref>。また実験室レベルでは、一般的な植物ウイルスに対する耐性の付与などの改変が行われている<ref name="Zadeh">{{cite journal|year=2004|title=Transgenic resistance to tobacco ringspot virus|journal=Acta Virologica|volume=48|issue=3|pages=145–52|pmid=15595207|vauthors=Zadeh AH, Foster GD}}</ref>。[[ケシ]]による非[[麻薬]]性[[アルカロイド]]の産生も試みられている<ref name="Allen">{{cite journal|date=December 2004|title=RNAi-mediated replacement of morphine with the nonnarcotic alkaloid reticuline in opium poppy|journal=Nature Biotechnology|volume=22|issue=12|pages=1559–66|doi=10.1038/nbt1033|pmid=15543134|vauthors=Allen RS, Millgate AG, Chitty JA, Thisleton J, Miller JA, Fist AJ, Gerlach WL, Larkin PJ|s2cid=8290821}}</ref>。 |
|||
==== 殺虫剤 ==== |
|||
[[殺虫剤]]としてのRNAiの開発が行われており、遺伝子操作や外部からの投与など複数のアプローチがとられている<ref name="s1308" />。一部の昆虫の中腸の細胞は、environmental RNAiと呼ばれる過程でdsRNA分子を取り込むことがある<ref name=":53">{{cite journal|date=May 2015|title=Environmental RNAi in herbivorous insects|journal=RNA|volume=21|issue=5|pages=840–50|doi=10.1261/rna.048116.114|pmid=25802407|pmc=4408792|vauthors=Ivashuta S, Zhang Y, Wiggins BE, Ramaseshadri P, Segers GC, Johnson S, Meyer SE, Kerstetter RA, McNulty BC, Bolognesi R, Heck GR}}</ref>。一部の昆虫では、その効果は昆虫の体中に広がり、全身に作用する<ref name=":54">{{cite journal|year=2012|title=Dissecting systemic RNA interference in the red flour beetle ''Tribolium castaneum'': parameters affecting the efficiency of RNAi|journal=PLOS ONE|volume=7|issue=10|pages=e47431|bibcode=2012PLoSO...747431M|doi=10.1371/journal.pone.0047431|pmid=23133513|pmc=3484993|vauthors=Miller SC, Miyata K, Brown SJ, Tomoyasu Y|doi-access=free}}</ref>。ヒトがこうした殺虫性のdsRNAを発現する遺伝子組み換え作物を消費することで予想される曝露量の数百万倍の量を曝露した場合でも、動物に悪影響はみられない<ref name=":55">{{cite journal|date=November 2016|title=Corn rootworm-active RNA DvSnf7: Repeat dose oral toxicology assessment in support of human and mammalian safety|journal=Regulatory Toxicology and Pharmacology|volume=81|pages=57–68|doi=10.1016/j.yrtph.2016.07.009|pmid=27436086|vauthors=Petrick JS, Friedrich GE, Carleton SM, Kessenich CR, Silvanovich A, Zhang Y, Koch MS|doi-access=free}}</ref>。 |
|||
RNAiの影響は[[鱗翅目]](チョウやガ)の生物種によってさまざまに異なり、おそらくそれは唾液や消化液のRNA分解能力の違いによるものである。cotton bollworm、{{仮リンク|シロイチモジヨトウ|en|Beet armyworm}}、{{仮リンク|ニカメイガ|en|Chilo suppressalis}}では給餌によるRNAi感受性は示されていない<ref name="s1308" />。 |
|||
RNAiに対する耐性は広域的、すなわちある配列に対する耐性が他のdsRNA配列に対する耐性も付与する可能性が示唆されている。ある{{仮リンク|ウエスタンコーンルートワーム|en|Western corn rootworm}}の実験室集団では、腸からのDvSnf7を標的としたdsRNAの取り込みが起こらないために耐性が生じている<ref name=":56">{{cite journal|date=2018-05-14|title=Development and characterization of the first dsRNA-resistant insect population from western corn rootworm, Diabrotica virgifera virgifera LeConte|journal=PLOS ONE|volume=13|issue=5|pages=e0197059|bibcode=2018PLoSO..1397059K|doi=10.1371/journal.pone.0197059|pmid=29758046|pmc=5951553|vauthors=Khajuria C, Ivashuta S, Wiggins E, Flagel L, Moar W, Pleau M, Miller K, Zhang Y, Ramaseshadri P, Jiang C, Hodge T, Jensen P, Chen M, Gowda A, McNulty B, Vazquez C, Bolognesi R, Haas J, Head G, Clark T|display-authors=6|doi-access=free}}</ref>。DvSnf7に対する他のdsRNA配列を試した際にも有効性は見られず、耐性管理は単純にdsRNAの配列を切り替えるだけでは困難であることが示唆される。[[バチルス・チューリンゲンシス]]''Bacillus thuringiensis''由来のCryタンパク質とRNAiなど複数の戦略を併用することで、耐性の出現は遅らせることができると考えられている<ref name="s1308" /><ref name=":57">{{cite web |url=https://www.theatlantic.com/science/archive/2017/06/monsanto-rna-interference/531288/ |title=The EPA Quietly Approved Monsanto's New Genetic-Engineering Technology: It's the first time RNA interference will be used to kill insect pests. |first1=Sarah |last1=Zhang |work=The Atlantic |date=23 June 2017 |accessdate=2022-04-04}}</ref>。 |
|||
{{仮リンク|ショウジョウバエ属|en|Drosophila|redirect=1}}''Drosophila'' spp.、[[カイコガ]]''Bombyx mori''、[[トノサマバッタ]]属''Locusta'' spp、{{仮リンク|スポドプテラ属|en|Spodoptera}}''Spodoptera'' spp.、[[コクヌストモドキ]]''Tribolium castaneum''、{{仮リンク|トビイロウンカ|en|Brown planthopper}}''Nilaparvata lugens''、{{仮リンク|オオタバコガ|en|Helicoverpa armigera}}''Helicoverpa armigera''、[[セイヨウミツバチ]]''Apis mellifera''は、昆虫の特定の系統内でRNAiがどのように機能するかを知るために広く利用されているモデルである。[[イエバエ]]''Musca domestica''は''Ago2''遺伝子を2つ持ち、[[ツェツェバエ]]''Glossina morsitans''は3つ持つことが知られている<ref name="Mongelli-Saleh-2016">{{cite journal|last1=Mongelli|first1=Vanesa|last2=Saleh|first2=Maria-Carla|date=2016-09-29|title=Bugs Are Not to Be Silenced: Small RNA Pathways and Antiviral Responses in Insects|url=https://hal-pasteur.archives-ouvertes.fr/pasteur-01957180/file/Mongelli%20and%20Saleh.pdf|journal=Annual Review of Virology|volume=3|issue=1|pages=573–589|publisher=Annual Reviews|doi=10.1146/annurev-virology-110615-042447|issn=2327-056X|pmid=27741406|s2cid=38499958}}</ref><ref name="Zhu-Palli-2020">{{cite journal|last1=Zhu|first1=Kun Yan|last2=Palli|first2=Subba Reddy|date=2020-01-07|title=Mechanisms, Applications, and Challenges of Insect RNA Interference|journal=Annual Review of Entomology|volume=65|issue=1|pages=293–311|publisher=Annual Reviews|doi=10.1146/annurev-ento-011019-025224|issn=0066-4170|pmid=31610134|s2cid=204702574}}</ref>。miRNA経路に関しては、{{仮リンク|ロシアコムギアブラムシ|en|Russian wheat aphid}}''Diuraphis noxia''は2つの''Ago1''、''M. domestica''は2つの''Dcr1''、{{仮リンク|エンドウヒゲナガアブラムシ|en|Acyrthosiphon pisum}}''Acyrthosiphon pisum''は''Ago1''、''Loqs''、''Dcr1''を2つずつ持ち、''Pasha''を4つ持つ。{{仮リンク|piRNA|en|Piwi-interacting RNA}}に関しては、''G. morsitans''と''A. pisum''は2つまたは3つの''Ago3''を持つ<ref name="Zhu-Palli-2020" />。こうした研究により、将来的な殺虫剤開発の標的や、作用機序、他の殺虫剤に対する耐性の理由などが明らかとなった<ref name="Zhu-Palli-2020" />。 |
|||
===== トランスジェニック植物 ===== |
|||
トランスジェニック作物はdsRNAを発現するように作製されており、その配列は標的害虫の重要な遺伝子をサイレンシングするよう慎重に選ばれたものである。こうしたdsRNAは、特定の遺伝子配列を発現する昆虫のみに影響を与えるよう設計されている。2009年の実証実験では、RNAが4種のショウジョウバエのうちいずれか1種のみに対して殺虫作用を示し、他の3種には害を及ぼさないことが示された<ref name="s1308" />。 |
|||
2012年、[[シンジェンタ]]はベルギーのRNAi企業Devgenを5億2200万ドルで買収し、[[モンサント (企業)|モンサント]]は{{仮リンク|アルナイラム・ファーマシューティカルズ|en|Alnylam Pharmaceuticals}}から[[知的財産権]]の独占権を2920万ドルで取得した。ペルー・リマの{{仮リンク|国際ポテトセンター|en|International Potato Center}}では、幼虫による[[サツマイモ]]の食害が世界的に問題となっている[[アリモドキゾウムシ]]に対する標的遺伝子の探索が行われている。他にも、アリ、毛虫、pollen beetleなどの遺伝子のサイレンシングが試みられている。モンサントは、アメリカ合衆国だけで毎年10億ドルの被害をもたらしているウエスタンコーンルートワームの''Snf7''遺伝子を標的としたdsRNAを発現する、トランスジェニックトウモロコシの種子を初めて販売することとなる可能性が高い。2012年の論文では、Snf7のサイレンシングは幼虫の成長を止め、数日以内に死滅させることが示されている。2013年に同チームは、このRNAが他の生物種に影響を与えることはほとんどないことを示した<ref name="s1308" />。 |
|||
===== 外部からの投与 ===== |
|||
dsRNAは遺伝子組み換え以外の方法でも供給することができる。1つのアプローチは、[[灌漑]]用水への添加である。RNA分子は植物の[[維管束]]系へ吸収され、その植物を食べる昆虫を殺す。他のアプローチは、従来の農薬のような形でのdsRNAの噴霧である。こうした方法は耐性の出現に対してより早く対応することができるが、dsRNAの低コストでの作製法を必要とし、そうした手法は現在のところ存在しない<ref name="s1308" />。 |
|||
=== ゲノムスケールでのスクリーニング === |
|||
ゲノムスケールでのRNAi研究は、ハイスループットスクリーニング(HTS)技術に依存している。RNAi HTS技術はゲノムワイドな機能喪失スクリーニングを可能にし、特定の表現型と関係する遺伝子の同定に広く利用されている。この技術は、遺伝子発現[[マイクロアレイ]]や[[一塩基多型]]発見プラットフォームといった[[ゲノミクス]]の第一の波に続く、第二の波となる可能性があるとの評価がなされている<ref name="Matson"> |
|||
{{cite book|author=Matson RS|year=2005|title=Applying genomic and proteomic microarray technology in drug discovery|publisher=CRC Press|page=[https://archive.org/details/applyinggenomicp0000mats/page/6 6]|isbn=978-0-8493-1469-8|url-access=registration|url=https://archive.org/details/applyinggenomicp0000mats/page/6}}</ref>。ゲノムスケールでのRNAiスクリーニングの大きな利点の1つは、数千もの遺伝子を同時に調査することができる点である。ゲノムスケールでのRNAiスクリーニングでは1つの実験から大量のデータが生み出されるため、データ生成量の爆発的増加をもたらしている。こうした巨大なデータセットの処理は基本的な課題となっており、適切な統計学やバイオインフォマティクス的手法を必要とする。細胞ベースのRNAiスクリーニングの基本的過程は、RNAiライブラリや頑強で安定した細胞種の選択、RNAi試薬によるトランスフェクション、処理とインキュベーション、シグナル検出、重要な遺伝子または治療標的遺伝子の解析、同定などからなる<ref name="ZhangXHD">{{cite book|author=Zhang XHD|year=2011|title=Optimal High-Throughput Screening: Practical Experimental Design and Data Analysis for Genome-scale RNAi Research|publisher=Cambridge University Press|url=http://www.cambridge.org/9780521734448|isbn=978-0-521-73444-8|pages=ix–xiii}}</ref>。 |
|||
== 歴史 == |
|||
[[File:Rnai phenotype petunia crop.png|thumb|250px|right|RNAiによって色素形成のための遺伝子がサイレンシングされたペチュニアの例。左は野生型、右の2つでは導入遺伝子と内在性遺伝子の双方の発現の抑制を誘導する遺伝子導入の結果、色素を持たない白い領域が生じている<ref name="Matzke">{{cite journal|year=2004|title=Planting the Seeds of a New Paradigm|journal=PLOS Biol|volume=2|issue=5|pages=e133|doi=10.1371/journal.pbio.0020133|pmid=15138502|pmc=406394|vauthors=Matzke MA, Matzke AJ}}</ref>。]] |
|||
RNAi過程は、それがRNAと関連した機構であることが知られる前には、"co-suppression"や"quelling"と呼ばれていた。RNAiの発見に先立って、トランスジェニック植物で発現させたアンチセンスRNAによる転写阻害がまず観察され<ref name="Ecker_1986">{{cite journal|date=August 1986|title=Inhibition of gene expression in plant cells by expression of antisense RNA|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=83|issue=15|pages=5372–6|bibcode=1986PNAS...83.5372E|doi=10.1073/pnas.83.15.5372|pmid=16593734|pmc=386288|vauthors=Ecker JR, Davis RW|doi-access=free}}</ref>、そして1990年代初頭のアメリカ合衆国とオランダの植物学者によって行われた実験による予想外の結果の報告<ref name="Napoli_1990">{{cite journal|date=April 1990|title=Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans|journal=The Plant Cell|volume=2|issue=4|pages=279–289|doi=10.1105/tpc.2.4.279|pmid=12354959|pmc=159885|vauthors=Napoli C, Lemieux C, Jorgensen R}}</ref>によってより直接的にRNAiの発見への道が開かれた。この実験では[[ペチュニア]]の花の色の変化が試みられ、研究者らは正常なピンクまたはスミレ色の花のペチュニアに対し、花の色素形成に重要な酵素である{{仮リンク|カルコンシンターゼ|en|Chalcone synthase}}をコードする遺伝子のさらなるコピーを導入した。コピー数の増加による遺伝子の過剰発現によってより濃い色の花となることが予想されたが、実際には一部の花では紫色の色素は薄くなり、そして斑入りのパターンが形成されることもあった。このことは、カルコンシンターゼの活性は状況依存的に大きく低下するか、または抑制されていることを示していた。後に、一部の形質転換体のゲノム中のさまざまな位置で反対向きのプロモーターに隣接して導入遺伝子が挿入された結果、プロモーターの活性化によってアンチセンス転写産物が発現し、遺伝子がサイレンシングされたという説明がなされた。初期のRNAiの観察の他の例としては[[アカパンカビ]]''Neurospora crassa''の研究のものがあるが<ref name="Romano">{{cite journal|date=November 1992|title=Quelling: transient inactivation of gene expression in Neurospora crassa by transformation with homologous sequences|journal=Molecular Microbiology|volume=6|issue=22|pages=3343–53|doi=10.1111/j.1365-2958.1992.tb02202.x|pmid=1484489|vauthors=Romano N, Macino G|s2cid=31234985}}</ref>、これが関連した現象であるとはすぐには認識されなかった。植物での現象のさらなる研究によって、ダウンレギュレーションはmRNAの分解率の上昇を介した、遺伝子発現の転写後阻害によるものであることが示された<ref name="Van_Blokland_1994">{{cite journal|year=1994|title=Transgene-mediated suppression of chalcone synthase expression in ''Petunia hybrida'' results from an increase in RNA turnover|journal=Plant J|volume=6|issue=6|pages=861–77|doi=10.1046/j.1365-313X.1994.6060861.x|vauthors=Van Blokland R, Van der Geest N, Mol JN, Kooter JM}}</ref>。この現象は"co-suppression of gene expression"と呼ばれたが、その分子機構はいまだ不明のままであった<ref name=":58">{{cite book|title=Antisense nucleic acids and proteins: fundamentals and applications|vauthors=Mol JN, van der Krol AR|year=1991|pages=4, 136|publisher=M. Dekker|isbn=978-0-8247-8516-1}}</ref>。 |
|||
それから間もなく、ウイルス病に対する植物の耐性の改善に取り組んでいた植物ウイルス学者らによって、類似した予想外の現象が観察された。ウイルス特異的タンパク質を発現する植物はウイルス感染に対するトレランス(tolerance)や抵抗性(resistance)の向上がみられることは知られていたが、ウイルスRNA配列の短い非コード領域のみを持つ植物も同様の防御レベルを示すという予想外の結果が得られた。研究者らは導入遺伝子によって産生されるウイルスRNAがウイルスの複製を阻害すると考えた<ref name="Covey_1997">{{cite journal|year=1997|title=Plants combat infection by gene silencing|journal=Nature|volume=385|issue=6619|pages=781–2|bibcode=1997Natur.385..781C|doi=10.1038/385781a0|vauthors=Covey S, Al-Kaff N, Lángara A, Turner D|s2cid=43229760}}</ref>。逆実験として、植物遺伝子の短い配列を導入したウイルスは、感染した植物で標的遺伝子を抑制することが示された<ref name="Kumagai_1995">{{cite journal|date=February 1995|title=Cytoplasmic inhibition of carotenoid biosynthesis with virus-derived RNA|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=92|issue=5|pages=1679–83|bibcode=1995PNAS...92.1679K|doi=10.1073/pnas.92.5.1679|pmid=7878039|pmc=42583|vauthors=Kumagai MH, Donson J, della-Cioppa G, Harvey D, Hanley K, Grill LK|doi-access=free}}</ref>。この現象は"virus-induced gene silencing"(VIGS)と呼ばれ、これらの現象はまとめて"post transcriptional gene silencing"と呼ばれるようになった<ref name="Ratcliff_1997">{{cite journal|date=June 1997|title=A similarity between viral defense and gene silencing in plants|journal=Science|volume=276|issue=5318|pages=1558–60|doi=10.1126/science.276.5318.1558|pmid=18610513|vauthors=Ratcliff F, Harrison BD, Baulcombe DC}}</ref>。 |
|||
こうした植物における初期の観察の後、他の生物種におけるこうした現象の探索が行われた<ref name="Guo">{{cite journal|date=May 1995|title=par-1, a gene required for establishing polarity in C. elegans embryos, encodes a putative Ser/Thr kinase that is asymmetrically distributed|journal=Cell|volume=81|issue=4|pages=611–20|doi=10.1016/0092-8674(95)90082-9|pmid=7758115|vauthors=Guo S, Kemphues KJ|doi-access=free}}</ref><ref name="Pal-Bhadra">{{cite journal|date=August 1997|title=Cosuppression in Drosophila: gene silencing of Alcohol dehydrogenase by white-Adh transgenes is Polycomb dependent|journal=Cell|volume=90|issue=3|pages=479–90|doi=10.1016/S0092-8674(00)80508-5|pmid=9267028|vauthors=Pal-Bhadra M, Bhadra U, Birchler JA|doi-access=free}}</ref>。クレイグ・メローとアンドリュー・ファイアーによる1998年の''Nature''誌の論文では、''C. elegans''に二本鎖RNAを注入した後に強力な遺伝子サイレンシング効果がみられることが報告された<ref name="Fire">{{cite journal|date=February 1998|title=Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans|journal=Nature|volume=391|issue=6669|pages=806–11|bibcode=1998Natur.391..806F|doi=10.1038/35888|pmid=9486653|vauthors=Fire A, Xu S, Montgomery MK, Kostas SA, Driver SE, Mello CC|s2cid=4355692}}</ref>。彼らは筋タンパク質の産生の調節の研究の際に、mRNAやアンチセンスRNAの注入はタンパク質産生に影響を及ぼさないが、二本鎖RNAの注入によって標的遺伝子がサイレンシングされることを発見した。この研究をもとに、RNAiという用語を作った。この発見は、この現象の原因となる因子を初めて同定したこととなる。ファイアーとメローは2006年にノーベル生理学・医学賞を受賞した<ref name="Daneholt2006" />。 |
|||
== 出典 == |
|||
{{reflist|2}} |
|||
== 外部リンク == |
== 外部リンク == |
||
* [http://www.thenakedscientists.com/HTML/articles/article/rna-interference-explained/ Overview of the RNAi process], from ''Cambridge University's The Naked Scientists'' |
|||
*{{脳科学辞典|RNA干渉}} |
|||
* [http://www.nature.com/focus/rnai/animations/animation/animation.htm Animation of the RNAi process], from ''[[Nature (journal)|Nature]]'' |
|||
* [https://www.pbs.org/wgbh/nova/sciencenow/3210/02.html NOVA scienceNOW explains RNAi] – A 15-minute video of the ''Nova'' broadcast that aired on [[Public Broadcasting Service|PBS]], 26 July 2005 |
|||
* [http://www.silencinggenomes.org/ Silencing Genomes] RNA interference (RNAi) experiments and bioinformatics in C. elegans for education. From the Dolan DNA Learning Center of Cold Spring Harbor Laboratory. |
|||
* [https://web.archive.org/web/20061116000124/http://www.natureprotocols.com/2006/07/19/rnai_screens_in_c_elegans_in_a.php RNAi screens in C. elegans in a 96-well liquid format and their application to the systematic identification of genetic interactions (a protocol)] |
|||
* [https://www.nytimes.com/2006/10/03/science/03nobel.html?ex=1180065600&en=e26ec371cb398ea0&ei=5070 2 American ‘Worm People’ Win Nobel for RNA Work], from ''[[The New York Times|NY Times]]'' |
|||
* [http://www.nature.com/mt/webfocus/rnai/index.html#2008 ''Molecular Therapy'' web focus: "The development of RNAi as a therapeutic strategy"], a collection of free articles about RNAi as a therapeutic strategy. |
|||
* [http://www.genomernai.de/GenomeRNAi/ GenomeRNAi]: a database of phenotypes from RNA interference screening experiments in Drosophila melanogaster and Homo sapians |
|||
* [http://dharmacon.horizondiscovery.com/rnai-and-custom-rna-synthesis/ RNAi tools] {{Webarchive|url=https://web.archive.org/web/20180619035904/http://dharmacon.horizondiscovery.com/rnai-and-custom-rna-synthesis/ |date=19 June 2018 }} Pre-designed and custom RNA Interference tools |
|||
* {{Citation|和書|title=RNA干渉|last=塩見|first=美喜子|author-link=塩見美喜子|year=2015|url=https://doi.org/10.14931/bsd.3744|publisher=INCF Japan Node|doi=10.14931/bsd.3744|access-date=2022-04-17}} |
|||
* {{Kotobank}} |
|||
{{DEFAULTSORT:RNAかんしよう}} |
{{DEFAULTSORT:RNAかんしよう}} |
||
2022年4月17日 (日) 00:08時点における版
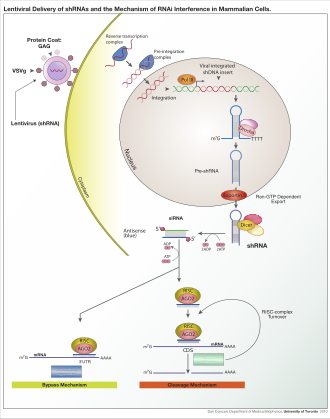
RNA干渉(RNAかんしょう、英: RNA interference、RNAi)は、二本鎖RNA(dsRNA)が翻訳抑制または転写抑制によって遺伝子の発現を配列特異的に抑制する生物学的過程である。RNAiは歴史的には、"co-suppression"、"post-transcriptional gene silencing"(PTGS)、"quelling"といった名称で知られていた。これらの過程は見かけ上異なるものの、それぞれに対して詳細な研究が行われ、これらの実体はすべてRNAiであることが明らかにされた。アンドリュー・ファイアーとクレイグ・メローは、1998年に発表された線虫Caenorhabditis elegansにおけるRNAiに関する業績によって、2006年にノーベル生理学・医学賞を受賞した。RNAiとその調節の可能性が発見されて以降、RNAiが目的の遺伝子を抑制する多大な可能性を有していることが明らかとなった。現在ではRNAiは、遺伝子抑制を目的としたアンチセンス治療よりも正確かつ効率的で安定なより良い治療法であることが知られている[1]。
RNAiでは、miRNAとsiRNAという2つのタイプの低分子RNAが中心的役割を果たす。RNAは遺伝子の直接産物であり、こうした低分子RNAは酵素複合体を指揮し、mRNAの分解や翻訳の阻害といった、転写後段階での遺伝子サイレンシングを介して標的遺伝子の活性を低下させる。さらに、siRNAやmiRNAに相補的なゲノム領域でDNAのメチル化を触媒する酵素複合体による、転写前段階でのサイレンシング機構も存在し、それによって転写が阻害されることもある。RNAiは、ウイルスやトランスポゾンといった寄生性のヌクレオチド配列に対する細胞の防御に重要な役割を果たす。また、発生にも影響を与える。
RNAi経路は動物を含む多くの真核生物でみられ、Dicerによって開始される。Dicerは長いdsRNA分子を約21ヌクレオチドからなる短いsiRNA二本鎖断片へと切断する酵素である。その後、siRNAは2つの一本鎖RNA(ssRNA)、すなわちパッセンジャー鎖とガイド鎖へと巻き戻される。パッセンジャー鎖は分解され、ガイド鎖はRNA誘導サイレンシング複合体(RISC)へと取り込まれる。RNAi経路の中で最もよく研究されているのは転写後段階での遺伝子サイレンシングであり、この過程ではガイド鎖がmRNA分子中の相補的な配列と対合し、RISCの触媒要素であるArgonaute2(Ago2)による切断が誘導される。一部の生物種では、当初のsiRNA濃度が限定的なものであっても、この過程が増幅し、全身に拡散する。
細胞に導入された合成dsRNAは目的の遺伝子の選択的かつ強固な抑制を誘導することとができるため、RNAiは培養細胞においても個体レベルにおいても有益な研究ツールである。RNAiは細胞内の各遺伝子を体系的にオフにする大規模スクリーニングに利用することができ、細胞分裂などのイベントや特定の細胞過程に必要な要素の同定のために活用することができる。また、この経路はバイオテクノロジーや医療、そして殺虫剤など実用的なツールとしても利用されている[2]。
細胞機構

RNAiは、RNA誘導サイレンシング複合体(RISC)によって制御されるRNA依存的遺伝子サイレンシング過程であり、細胞質に存在する短い二本鎖RNA(dsRNA)分子によって開始される。dsRNAが外因性のもの(RNAゲノムを持つウイルスの感染や実験室的操作に由来するもの)である場合には、RNAは直接細胞質に取り込まれ、Dicerによって短い断片へと切断される。ゲノム中のRNAコーディング遺伝子から発現したpre-miRNAなどのように、RNAi経路を開始するdsRNAは内因性のもの(細胞に由来するもの)である場合もある。こうした遺伝子に由来する一次転写産物は、まず核内でpre-miRNAに特徴的なステムループ構造を形成するようプロセシングされる。その後、外因性と内因性の2つのdsRNA経路はRISCへと集約される[4]。
外因性のdsRNAはリボヌクレアーゼタンパク質であるDicerによってRNAiを開始する[5]。Dicerは植物ではdsRNA、ヒトではshRNAと結合して切断を行い、3'末端に2ヌクレオチドの突出部を持つ20–25塩基対の二本鎖断片を形成する[6]。この長さは、標的遺伝子に対する特異性を最大化し、かつ非特異的な効果を最小化することが複数の生物種のゲノムに対するバイオインフォマティクス研究から示唆されている[7]。こうした短い二本鎖断片はsiRNAと呼ばれる。その後、RISCローディング複合体(RLC)によって、siRNAは一本鎖へと分離されて活性型のRISCへ取り込まれる。ショウジョウバエのRLCはDcr-2(Dicer2)とR2D2を含み、Ago2とRISCの一体化のために重要である[8]。TAF11はDcr-2とR2D2の四量体化を促進してRLCを組み立て、siRNAに対する結合親和性を10倍増加させる。TAF11との結合によって、R2D2/Dcr2-initiator(RDI)複合体はRLCへと変換される[9]。R2D2にはタンデムに並んだdsRNA結合ドメインが存在し、siRNA二本鎖の熱力学的に安定な末端を認識する。一方、Dcr-2は熱力学的安定性の低い末端を認識する。RISCへのRNAのローディングは非対称的であり、Ago2のMIDドメインはsiRNAの熱力学的に不安定な末端を認識する。そのため、5'末端がMIDドメインに認識されなかったパッセンジャー鎖は放出され、もう一方のガイド鎖はAgoと協調的にRISCを形成する[8]。
RISCへと取り込まれた後、siRNAは標的mRNAと塩基対を形成して切断を行い、そのmRNAが翻訳の鋳型として利用されることを防ぐ[10]。siRNAとは異なり、miRNAがロードされたRISCはmRNA上の相補性領域を探してスキャンする。 miRNAは通常不完全な相補性でmRNAの3' UTR領域に結合し、リボソームが翻訳のためにアクセスすることを防ぐ役割を果たす[11]。
miRNA

miRNAはゲノムにコードされたノンコーディングRNAであり、特に発生過程において遺伝子発現の調節を補助する[12]。広義のRNAiには、外来dsRNAから産生されるsiRNAによるものに加え、miRNAによる内因性の遺伝子サイレンシング効果も含まれる。成熟したmiRNAは外因性dsRNAから産生されたsiRNAと構造的に類似しているが、成熟するまでに広範囲の転写後修飾を必要とする。miRNAは最終産物よりもずっと長いRNAコーディング遺伝子からpri-miRNA(primary miRNA)と呼ばれる一次転写産物として発現し、核内でマイクロプロセッサー複合体によってステムループ構造を持つ約70ヌクレオチドの長さのpre-miRNA(precursor miRNA)へとプロセシングされる。マイクロプロセッサー複合体には、Droshaと呼ばれるRNase III酵素とdsRNA結合タンパク質DGCR8が含まれる。Dicerはこのpre-miRNAのdsRNA部分に結合して切断を行い、RISCに取り込まれる成熟型miRNA分子が産生される。これより下流では、miRNAとsiRNAは同様の装置を利用する[13]。ウイルス由来のmiRNAとしては、エプスタイン・バール・ウイルス(EBV)にコードされたものが最初に記載された[14]。それ以降、ウイルスでは多くのmiRNAの記載がなされている。VIRmiRNAは、ウイルス性のmiRNAとその標的、そして抗ウイルス性miRNAに関する総合的なカタログである[15]。
miRNAは長いdsRNA前駆体に由来するsiRNAとはいくつかの点で異なる。特に動物では、miRNAと標的mRNAとの塩基対形成は不完全であることが一般的であり、また類似した配列を持つ多くの異なるmRNAの翻訳を阻害する。対照的に、siRNAは通常は完全な塩基対形成を行い、唯一の特異的な標的に対してのみmRNAの切断を誘導する[16]。ショウジョウバエやC. elegansでは、miRNAとsiRNAはそれぞれ異なるArgonauteタンパク質とDicer酵素によってプロセシングされる[17][18]。
3' UTRとmiRNA
mRNAの3' UTRには、転写後にRNAiを引き起こす調節配列が存在することが多い。こうした3' UTRには、miRNAの結合部位と調節タンパク質の結合部位の双方が存在することが多い。miRNAは3' UTR内の特定の部位に結合することで、翻訳の阻害または転写産物の分解によって遺伝子発現を低下させる。また、3' UTRにはmRNAの発現を阻害するリプレッサータンパク質が結合するサイレンサー領域が存在する場合もある。
miRNAの配列とアノテーションがアーカイブされているウェブサイトmiRBase[19]には、2014年時点で233の生物種の28,645種類のエントリが登録されている。miRNAには平均して約400種類の標的mRNAが存在する(数百の遺伝子の発現に影響を与える)ことが予測されている[20]。ヒトのmRNAの3' UTRにはバックグラウンドレベルよりも高い水準で保存されている標的部位が45,000か所以上存在し、タンパク質コーディング遺伝子の60%以上に対してmiRNAとの対合を維持するような選択圧がはたらいていると推定されている[20]。
1種類のmiRNAが数百種類のmRNAの安定性を低下させる場合があることは、直接的な実験により示されている[21]。一方他の実験では、1種類のmiRNAが数百種類のタンパク質の産生を抑制する可能性があるものの、多くの場合こうした抑制は比較的弱いもの(2倍未満)であることが示されている[22][23]。
miRNAによる遺伝子発現の調節の異常は、がんにおいて重要であるようである[24]。一例として、消化器がんでは9種類のmiRNAにエピジェネティックな変化が生じ、DNA修復酵素をダウンレギュレーションする作用を示していることが同定されている[25]。
miRNAによる遺伝子発現調節の異常は、統合失調症、双極性障害、大うつ病、パーキンソン病、アルツハイマー病、自閉症スペクトラム障害など精神神経疾患にも重要であるようである[26][27][28]。
RISCの活性化と触媒
外因性のdsRNAには、C. elegansではRDE-4、ショウジョウバエではR2D2と呼ばれるエフェクタータンパク質が検知して結合し、Dicerの活性を刺激する[29]。このタンパク質は長いdsRNAにのみ結合するが、こうした長さに対する特異性を生み出す機構は不明である[29]。その後、このRNA結合タンパク質は切断されたsiRNAのRISCへの移行を促進する[30]。
C. elegansでは、Dicerによって産生された「一次性」のsiRNAを鋳型として「二次性」のsiRNAが合成されることで、この開始応答は増幅される[31]。こうした「二次性」のsiRNAはDicerによって産生されたsiRNAとは構造的に異なり、RNA依存性RNAポリメラーゼ(RdRP)によって産生されているようである[32][33]。


RISCの活性を担う構成要素はArgonauteタンパク質と呼ばれるエンドヌクレアーゼであり、結合したsiRNAに相補的な標的mRNAを切断する[34]。Dicerによって形成される断片は二本鎖であるため、理論上は双方の鎖が機能的なsiRNAとなることができる。しかしながら、2本の鎖のうちArgonauteタンパク質に結合し、遺伝子サイレンシングを指揮するのは、ガイド鎖と呼ばれる一方の鎖のみである。もう一方の鎖はパッセンジャー鎖またはアンチガイド鎖と呼ばれ、RISCの活性化の過程で分解される[35]。当初はATP依存的なヘリカーゼによって2つの鎖が分離されると考えられていたが[36]、この過程は実際にはATP非依存的であり、RISCの構成要素によって直接行われることが示されている[37][38]。しかしながら、in vitroにおけるATP存在下と非存在下でのRNAiの速度論的解析からは、触媒後の複合体から切断されたmRNAを巻き戻して除去するためにATPが必要である可能性が示されている[39]。ガイド鎖は5'末端の対合の安定性が低い傾向があるが[40]、RISCへの取り込みの前のDicerによるdsRNAの切断の方向には影響を与えない[41]。Dicerではなく、R2D2タンパク質がより安定性の高いパッセンジャー鎖の5'末端に結合することが鎖の識別因子として機能している可能性がある[42]。
Argonauteタンパク質のRNA結合の構造的基盤は、RNAが結合したArgonauteタンパク質のドメインのX線結晶構造解析によって研究されている。RNA鎖のリン酸化5'末端はArgonauteタンパク質の保存された塩基性ポケットに入り、マグネシウムなどの二価カチオンを介して、また5'末端のヌクレオチドと保存されたチロシン残基とのスタッキングによって接触を行っている。この部位はsiRNAが標的mRNAへ結合する際の核形成部位となると考えられている[43]。ガイド鎖の5'末端または3'末端のミスマッチによる阻害効果の解析からは、ガイド鎖の5'末端は標的mRNAとのマッチングと結合を担う一方で、3'末端は標的mRNAをRISCが切断を行いやすい領域へ物理的に配置をする役割を担っている可能性が高いことが示されている[39]。
活性化されたRISC複合体が細胞内のmRNA標的をどのように見つけているのかに関しては未解明である。切断過程と翻訳との関連性が提唱されているが、標的mRNAの翻訳はRNAiを介した分解に必要不可欠なものではない[44]。事実、RNAiは翻訳されていない標的mRNAに対してより効率的に働いている可能性がある[45]。Argonauteタンパク質はP-body(cytoplasmic body、GW bodyとも)と呼ばれる細胞質の特定の領域に局在している。この領域ではmRNAの分解が高率で行われており[46]、miRNAの活性もP-bodyに集中している[47]。P-bodyの破壊によってRNAiの効率が低下することから、この部位がRNAi過程に重要であることが示唆される[48]。
転写サイレンシング

RNAi経路の構成要素は、多くの真核生物においてゲノムの組織化と構造の維持に利用されている。ヒストンの修飾とそれに関連したヘテロクロマチン形成の誘導は、転写前段階で遺伝子をダウンレギュレーションする役割を果たす[50]。この過程はRNA誘導転写サイレンシング(RITS)と呼ばれ、RITS複合体と呼ばれるタンパク質複合体によって行われる。分裂酵母Schizosaccharomyces pombeでは、この複合体にはArgonaute、クロモドメインタンパク質Chp1、そしてTas3と呼ばれる機能未知タンパク質が含まれる[51]。ヘテロクロマチン領域の誘導と拡大にはArgonauteとRdRPタンパク質が必要である[52]。これらの遺伝子を欠失させた分裂酵母は、ヒストンメチル化とセントロメア形成が破壊され[53]、細胞分裂の進行は遅くなるか、もしくは後期の段階で停止する[54]。またある場合には、ヒストン修飾と関係した同様の過程によって遺伝子の転写がアップレギュレーションされることも観察されている[55]。
RITS複合体がヘテロクロマチンの形成や組織化を誘導する機構の詳細は未解明である。多くの研究は分裂酵母の接合型遺伝子座に焦点を当てているが、この遺伝子座における活性は他の生物やゲノム領域における活性を代表するものではない可能性もある。既存のヘテロクロマチン領域の維持に際しては、RITSはその領域の遺伝子に相補的なsiRNAと複合体を形成してその領域のメチル化ヒストンと安定に結合し、RNAポリメラーゼによって転写が開始されたpre-mRNA新生鎖を、転写と共役した形で分解している。ヘテロクロマチン領域の維持ではなく形成過程はDicer依存的であるが、それはおそらく転写産物を標的とするsiRNAが最初に形成される際にはDicerが必要であるためである[56]。新たなsiRNAは偶発的な転写による新生鎖からRdRPによって形成され、その領域に位置するRITS複合体へ取り込まれるため、ヘテロクロマチンの維持は自己強化型フィードバックループとして機能することが示唆されている[57]。分裂酵母の接合型遺伝子座やセントロメアにおける観察と哺乳類での現象との対応は明らかではなく、哺乳類細胞におけるヘテロクロマチンの維持はRNAi経路の構成要素とは無関係である可能性もある[58]。
RNA編集とのクロストーク
高等真核生物で最も広くみられるRNA編集は、ADARによるdsRNA中のアデノシンヌクレオチド(A)のイノシン(I)への変換である[59]。RNAiとA→IのRNA編集経路が共通したdsRNA基質をめぐって競合する可能性は2000年に提唱された[60]。一部のpre-miRNAはA→I RNA編集を受け[61][62]、この機構は成熟型miRNAへのプロセシングと発現を調節している可能性がある[62]。さらに、哺乳類のADARの少なくとも1種類に関してはsiRNAをRNAi経路の構成要素から隔離することが示されている[63]。ADARを持たないC. elegans系統を用いた研究からは、内在性遺伝子や導入遺伝子のRNAiによるサイレンシングにA→I RNA編集が対抗していることが示されており、このこともこのモデルを支持している[64]。

生物種間の多様性
外来dsRNAを取り込み、そしてそれらをRNAi経路で利用する能力は、生物によって差がある。RNAiの効果は、植物やC. eleganでは全身的かつ遺伝性のものであるが、ショウジョウバエや哺乳類ではそうではない。植物では、RNAiは原形質連絡(細胞間のコミュニケーションや輸送を可能にする、細胞壁に存在するチャネル)を介した細胞間でのsiRNAの輸送によって全身へ伝播していくと考えられている[36]。またその遺伝性はRNAiの標的となったプロモーターがメチル化されることによるものであり、メチル化パターンは新たな世代の細胞が生じるたびにコピーされる[66]。植物と動物のおおまかな違いは、内因的に産生されるmiRNAの標的性にある。植物では、通常miRNAはその標的遺伝子に対して(ほぼ)完全に相補的であり、RISCによる直接的なmRNAの切断が誘導されるのに対し、動物のmiRNAは標的となる配列がより多様である傾向があり、翻訳抑制が誘導される[65]。この翻訳抑制効果は、翻訳開始因子とmRNAのポリアデニル化テールとの相互作用の阻害によって行われている可能性がある[67]。
リーシュマニアLeishmania majorやトリパノソーマTrypanosoma cruziなど、一部の原生動物にはRNAi経路が全く存在しない[68][69]。一部の菌類でも大部分またはすべての構成要素が存在せず、そうした生物として最も有名なのはモデル生物でもある出芽酵母Saccharomyces cerevisiaeである[70]。Saccharomyces castelliiやカンジダCandida albicansなどの他の出芽酵母にはRNAiが存在し、S. castellii由来の2つのRNAi関連タンパク質を誘導することで、S. cerevisiaeでもRNAiが促進されることが示されている[71]。子嚢菌門や担子菌門の特定の種でRNAi経路が存在しないことは、RNAサイレンシングに必要なタンパク質が多くの菌類系統で独立に失われたことを示しており、おそらく類似した機能を持つ新たな経路の進化か、特定のニッチにおいて選択上の有利さが存在しなかったためであると考えられる[72]。
関連する原核生物のシステム
原核生物における遺伝子発現は、いくつかの面でRNAiと類似したRNAベースの系の影響を受ける。RNAをコードする遺伝子は、mRNAとアニーリングする相補的なRNAを産生することで、mRNAの存在量や翻訳を制御する。しかし、こうした調節性RNAにはDicerは関与せず、一般的にはmiRNAに類似したものとはみなされていない[73]。原核生物におけるCRISPR系が真核生物におけるRNAi系と類似したものであることも示唆されているが、どのタンパク質構成要素もオーソロガスではない[74]。
生物学的機能
免疫
RNAiはウイルスやその他の外来性遺伝物質に対する免疫応答の重要な部分をなしており、特に植物ではトランスポゾンの自己増殖も防いでいる[75]。シロイヌナズナArabidopsis thalianaなどの植物は複数のDicerホモログを発現しており、これらは植物が異なるウイルスに曝露した際には異なる反応を示すよう専門化されている[76]。RNAi経路が十分に理解される前から、植物における遺伝子サイレンシングの誘導がその植物全体に伝播したり、また接ぎ木によって台木から接ぎ穂へ移行したりすることがあることが知られていた[77]。この現象は、ウイルスに最初に遭遇した後、植物全体がウイルスへ応答することを可能にする、植物の免疫系の特徴として認識されていた[78]。一方、多くの植物ウイルスもRNAi経路を抑制する精巧な機構を進化させてきた[79]。こうしたものの中には、Dicerによって産生される一本鎖オーバーハング末端を持つ短い二本鎖RNA断片に対して結合するウイルスタンパク質などが含まれる[80]。一部の植物のゲノムは、特定種の細菌の感染に対する応答として、内因性のsiRNAを発現する[81]。これらは、感染過程の助けとなりうる宿主のあらゆる代謝過程をダウンレギュレーションする、病原体に対する全般的応答の一部である可能性がある[82]。
一般的に動物で発現しているDicerの種類は植物よりも少ないが、一部の動物ではRNAiが抗ウイルス応答を行っている。ショウジョウバエでは幼体と成体の双方において、RNAiは抗ウイルス免疫応答に重要であり、ショウジョウバエXウイルスなどの病原体に対して活性を示す[83][84]。免疫における同様の役割はC. elegansでも作用している可能性があり、ウイルスに応答してArgonauteタンパク質はアップレギュレーションされ、またRNAi経路のの構成要素を過剰発現した線虫はウイルス感染に対して耐性を示す[85][86]。
哺乳類の自然免疫におけるRNAiの役割の理解は進んでおらず、比較的わずかなデータしか存在しない。哺乳類細胞における機能的な抗ウイルスRNAi経路の存在を示す証拠は提示されており[87][88]、また哺乳類細胞のRNAi応答を抑制する遺伝子をコードするウイルスの存在は哺乳類におけるRNAi依存的な免疫応答を支持する証拠となる可能性がある[89][90]。しかしながら、この仮説には十分な証拠がないとして異議も唱えられている[91]。
哺乳類のウイルスにおけるRNAiには他の機能もあり、ヘルペスウイルスが発現するmiRNAはウイルス潜伏を媒介するヘテロクロマチン組織化の引き金として作用している可能性がある[92]。
遺伝子のダウンレギュレーション
内因的に発現しているmiRNAは、イントロン内に位置するのものも遺伝子間領域に位置するものも、翻訳抑制[65]そして発生の調節に最も重要であり、形態形成の時期の決定や、幹細胞などの未分化状態や不完全分化状態の細胞種の維持に特に重要である[93]。遺伝子発現のダウンレギュレーションにおける内因性miRNAの役割は、1993年にC. elegansで初めて記載された[94]。植物では、この機能はシロイヌナズナのmiR-JAWが植物の形状を制御するいくつかの遺伝子の調節に関与していることが示された際に発見された[95]。植物では、miRNAによって調節される遺伝子の大部分は転写因子である[96]。そのためmiRNAの活性は特に広範囲にわたり、転写因子やFボックスタンパク質など重要な調節遺伝子を調節することで発生時に遺伝子ネットワーク全体を調節する[97]。ヒトを含む多くの生物では、miRNAは腫瘍形成や細胞周期の調節異常と関連づけられている。miRNAはがん遺伝子としてもがん抑制遺伝子としても機能する[98]。
進化
最大節約法による系統学的解析に基づくと、全真核生物の最近共通祖先はすでに原始的なRNAi経路を持っていた可能性が極めて高く、特定の真核生物にRNAi経路が存在しないことは派生形質であると考えられている[99]。この祖先型のRNAi系は、少なくとも1つのDicer様タンパク質、1つのArgonauteタンパク質、1つのPiwiタンパク質、そしてRNA依存性RNAポリメラーゼを含んでいたと考えられ、これらは細胞中で他の役割を果たしていた可能性もある。大規模な比較ゲノミクス研究でも同様に、真核生物のクラウングループはすでにこれらの構成要素を持っており、エキソソームなどの一般的なRNA分解系とより密接な機能的関係を持っていた可能性があることが示唆されている[100]。またこの研究は、真核生物、大部分の古細菌、そして少なくとも一部の細菌(Aquifex aeolicusなど)に共通して存在するRNA結合性のArgonauteタンパク質ファミリーは、翻訳開始系の構成要素と相同であり、そしてそこから進化したものであることも示唆している[100]。
応用
遺伝子ノックダウンのためのRNAi経路
RNAi経路は実験生物学において、培養細胞やモデル生物におけるin vivoでの遺伝子機能の研究のためにしばしば利用される[34]。目的の遺伝子に対して相補的な配列を持つ二本鎖RNAが合成され、細胞または個体に導入される。そこで二本鎖RNAは外因性の遺伝子物質と認識され、RNAi経路が活性化される。この機構を用いて標的遺伝子の発現の劇的な低下を引き起こすことが可能であり、そしてこの低下の影響を研究することで遺伝子産物の生理学的役割を示すことができる。RNAiでは遺伝子の発現が完全には抑制されない場合があるため、遺伝子の発現が完全に除去される「ノックアウト」と区別して、「ノックダウン」と呼ばれることがある[101]。遺伝子アレイデータを用いたRNAiのサイレンシング効率の検証では、429の独立した実験において失敗率は18.5%であることが示されている[102]。
遺伝子ノックダウン効果を最大化しオフターゲット効果を最小化するdsRNAの設計法に関して、計算生物学では多くの研究がなされている。オフターゲット効果は、導入されたRNAの配列が複数の遺伝子と対合して発現を低下させるために生じる。ヒト、C. elegans、S. pombeのゲノム研究からは、可能なsiRNA配列のうち約10%で重大なオフターゲット効果が生じる可能性があると推定されている[7]。一般的[103][104]、哺乳類特異的[105]、そしてウイルス特異的[106]なsiRNAを設計し、自動的に交差反応性のチェックを行うアルゴリズムを備えたソフトウェアツールが多く開発されている。
生物種や実験系に応じて、外因性RNAはDicerによって切断されるように設計された長鎖RNAである場合や、siRNA基質として作用するよう設計された短鎖RNAである場合がある。ほとんどの哺乳類細胞では、長い二本鎖RNA分子に対しては外来遺伝物質に非特異的に作用する自然免疫の一種であるインターフェロン応答が誘導されるため、短いRNAが利用される[107]。マウスの卵母細胞や初期胚は外因性dsRNAに対するこの応答を欠くため、哺乳類の遺伝子ノックダウン効果を研究するための一般的なモデル系となっている[108]。siRNAが転写される適切な配列をコードしたプラスミドの安定トランスフェクションや[109]、より精巧なレンチウイルスベクターシステムによって転写の活性化や不活性化の誘導を可能にしたコンディショナルRNAi(conditional RNAi)と呼ばれる技術など[110][111]、siRNAの直接導入を回避することで哺乳類系におけるRNAiの有用性を改善した特殊な実験技術も開発されている。
機能ゲノミクス

ゲノムワイドRNAiライブラリの設計には、一定の実験条件のセットに対して単一のsiRNAを設計するよりも高度な技術が必要となる場合がある。siRNAライブラリの設計[112]や遺伝子ノックダウン時の効率の予測[113]には、ニューラルネットワークがよく利用される。マスゲノムスクリーニングはゲノムアノテーションのための有望な方法として広く知られており、マイクロアレイベースのハイスループットスクリーニングの開発の引き金となった[114][115]。しかし、こうしたスクリーニングの有用性や、モデル生物で開発された技術が近縁種にも一般化できるのか、例えばC. elegansの技術が関連する寄生性線虫に応用可能であるのかに関しては疑問視されている[116][117]。
医療
医療におけるRNAiの利用の歴史

動物におけるRNAサイレンシングの最初の例は1996年に記載された。線虫C. elegansにおいて、par-1 mRNAのセンス鎖とアンチセンス鎖のRNAを導入することで、par-1 mRNAの分解が引き起こされることが観察された[118]。この分解は一本鎖RNAによって開始されると考えられていたが、その2年後の1998年、ファイアーとメローによってこのpar-1遺伝子発現のサイレンシング能力は実際には二本鎖RNAによって開始されていることが発見された[118]。この発見によって、彼らはノーベル生理学・医学賞を受賞した[119]。この画期的な発見の直後、合成siRNAを用いることで、サイレンシングの標的は遺伝子全体ではなく遺伝子中の特定の配列とすることが可能であることが発見された[120]。そのわずか1年後、トランスジェニックマウスにおいてC型肝炎ウイルス配列を標的として、この配列特異的なサイレンシングの治療応用が実証された[121]。それ以降、RNAiの治療応用を広げる試みが多くの研究者によってなされ、具体的にはさまざまなタイプのがんを引き起こす遺伝子を標的とすることに関心が寄せられた[122][123]。2006年までに臨床試験に到達した最初の応用は、黄斑変性とRSウイルスの治療であった[124]。その4年後、ナノ粒子デリバリーシステムを用いて、固形腫瘍を標的としたヒトでの初めての第I相臨床試験が開始された[125]。現在のところ大部分の研究はがん治療へのRNAiの応用を試みるものであるが、可能な応用は広範囲にわたる。RNAiは、ウイルス[126]、細菌[127]、寄生虫による感染症[128]、不適応な遺伝子変異の治療[129]、薬物使用の制御[130]、疼痛管理[131]、さらには睡眠の調節[132]にも利用できる可能性がある。
治療応用
ウイルス感染
抗ウイルス治療はRNAiベースの医療応用として最初期に提唱されたものであり、2つの異なる種類のものが開発されている。1つはウイルスのRNAを標的とするものである。ウイルスのRNAを標的化することで、HIV[133]、HPV[134]、A型肝炎ウイルス[135]、B型肝炎ウイルス[136]、インフルエンザウイルス[137][138][139]、RSウイルス[140]、SARSコロナウイルス[140]、アデノウイルス[140]、麻疹ウイルス[141]など、多数のウイルスの複製を抑制できることが多くの研究により示されている。もう1つの戦略は、宿主細胞の遺伝子を標的とすることでウイルスの侵入を防ぐものである[142]。たとえば、宿主細胞のケモカイン受容体(CXCR4とCCR5)を抑制することでHIVの侵入を防ぐことができる[143]。
がん
伝統的な化学療法はがん細胞を効果的に殺すことができるが、正常細胞とがん細胞を区別する特異性を欠くために通常は重大な副作用を伴う。RNAiはがんと関連した遺伝子(がん遺伝子など)を標的とすることで腫瘍の成長を阻害する、より特異的なアプローチとなることが多くの研究で示されている[144]。RNAiはがん細胞の化学療法薬に対する感受性を高めることも可能であり、化学療法との併用療法も提唱されている[145]。細胞の浸潤や遊走の阻害も、RNAiベースの他の治療法となる可能性がある[146]。
神経疾患
RNAiは神経変性疾患の治療法となる可能性が示されている。細胞やマウスでの研究では、アミロイドβ(Aβ)を産生する遺伝子(BACE1やAPPなど)をRNAiで特異的に標的化することで、アルツハイマー病と関係するAβペプチドの量を大きく低下させることができることが示されている[147][148][149]。さらに、こうしたサイレンシングベースのアプローチは、パーキンソン病やポリグルタミン病の治療においても有望な結果をもたらしている[150][151][152]。
治療応用における困難
RNAiの臨床的可能性の実現のためには、siRNAが効率的に標的組織の細胞へ輸送される必要がある。しかしながら、臨床利用までに克服すべきさまざまな障壁が存在する。例えば、「裸」のsiRNAはその治療効力を低下させるいくつかの障害の影響を受けやすい[153]。いったんsiRNAが血流に移行すると、裸のRNAは血清中のヌクレアーゼによって分解されたり、自然免疫系を刺激したりする[153]。また、そのサイズと高いアニオン性のため、未修飾のsiRNA分子が細胞膜を通って細胞内へ移行するのは容易ではない。そのため、人工的なsiRNAやナノ粒子に封入したsiRNAを利用する必要がある。しかしながら、細胞膜を越えたsiRNAの輸送にはさらに固有の問題が存在する。siRNAが細胞膜を越えて輸送された場合、その量が最適化されていなければ意図しない毒性が生じたり、オフターゲット効果(部分的な配列相補性を持つ遺伝子に対する意図しないダウンレギュレーションなど)が生じたりする可能性がある[154]。細胞に移行した後も、その効果は細胞分裂ごとに希釈されるため繰り返し投与が必要である。また、dsRNAを運搬するベクターの一部には調節作用がある場合があるため、非特異的な副作用も考慮し、制御される必要がある[155]。
免疫応答の刺激
ヒトの免疫系は、自然免疫系と獲得免疫系の2種類に分類される[156]。自然免疫系は感染に対する第一の防御機構であり、病原体に対して一般的応答を行う[156]。一方、獲得免疫系は自然免疫系よりも後で進化した系であり、病原体の分子の特定の部分に反応するよう訓練された、高度に専門化されたB細胞とT細胞によって構成される[156]。
siRNAは自然免疫系によって制御されおり、自然免疫系による応答はさらに急性炎症応答と抗ウイルス応答に分類される[156]。炎症応答では、低分子シグナル伝達分子やIL-1、IL-6、IL-12、TNF-αなどのサイトカインが誘導される。こうした炎症性サイトカインは食作用を刺激し、侵入した病原体を破壊する[156]。抗ウイルス応答では、IFN-αやIFN-βなどのタイプIインターフェロンの放出や抗ウイルス遺伝子のアップレギュレーションが誘導される[156]。どちらの応答も、パターン認識受容体(PRR)の刺激を介して引き起こされる。複数のPRRによってRNA構造のさまざまな側面が認識されるため、免疫刺激を避けることは困難なものとなっている[156]。
治療技術としての展望
2015年から2017年にかけて行われたsiRNA治療の第I・II相試験では、肝臓での強力かつ持続的な遺伝子ノックダウン効果と臨床効果を示す一部の徴候がみられ、許容できない毒性はみられなかった[154]。トランスサイレチンの変異によって引き起こされる家族性神経変性・心筋症の治療へ向けた2つの第III相試験が進行中である[154]。多くの研究でin vivoデリバリーシステムの有望性は示されており、またそれらの多様な特性は無数の応用を可能にしている。最も有望なものはナノ粒子デリバリーシステムであるが、製品の安定した品質を確保するためには厳密に制御された混合過程が必要となることなど、製造過程のスケールアップにはさらなる課題が残されている[153]。
バイオテクノロジー
RNAiはバイオテクノロジー分野で応用されており、他の分野でも商業化が近い。ニコチンを含まないタバコ、カフェインを含まないコーヒー、栄養素を強化した野菜、低アレルゲンの作物など、RNAiを利用した新たな作物が発されている。遺伝子改変されたリンゴArctic Applesは2015年にFDAの承認を受けた[157]。このリンゴはPPO(ポリフェノールオキシダーゼ)遺伝子をRNAiによって抑制することで、果実を切った後の褐変が起こらないようになっている。PPOがサイレンシングされたリンゴは、クロロゲン酸を標準的なキノン産物へと変換することができないため、変色が起こらない[1]。
作物学におけるRNAiの応用には、ストレス耐性の付与や栄養素の強化などの改善などいくつかの可能性がある。RNAiはC3植物の生産性の向上のための光呼吸の阻害のほか、早期の開花、成熟や老化の遅れ、休眠の解除、ストレスに強い植物、自家不和合性の克服などの誘導に有用である可能性がある[1]。
食品
RNAiは食料生産における将来的有望性が示されているが、まだ若い技術であるためその利点と欠点に対する理解に欠けるところがある。そのため、よりよく理解し誤解を取り除く必要がある[158]。RNAiはすでに、天然毒素の産生が少ない遺伝子組み換え植物に利用されている。こうした技術は、植物においてRNAiの表現型が安定かつ遺伝性のものであることを利用している。ワタの種子はタンパク質に富むが、有毒テルペノイドであるゴシポールを含むためヒトの食物としては適さない。ゴシポール自体は害虫による損傷から植物を守るために重要であるため、植物の他の部分に影響を与えることなく種子でのみゴシポールの産生に重要な酵素δ-カジネンシンターゼを減少させるためにRNAiが利用されている[159]。同様に、キャッサバでシアン化物の原料となるリナマリンを減少させる試みも行われている[160]。
トマト類ではアレルゲンの減少[161]や抗酸化物質の強化[162]に成功している。Flavr Savrトマトやパパイヤリングスポットウイルス耐性パパイヤの2品種など、これまで商業化されている品種はもともとアンチセンス技術を用いて開発されたものであるが、実際にはRNAi経路が利用されている可能性が高い[163][164]。アスペルギルス・フラバスAspergillus flavusのα-アミラーゼを標的としたRNAiによるサイレンシングはトウモロコシ内でのこの菌類の増殖を低下せ、穀物を危険なアフラトキシンによる汚染から防ぐために利用されている[165]。タマネギでの催涙因子合成酵素(lachrymatory factor synthase)のサイレンシングは切っても涙の出ないタマネギの生産に、アブラナでのBP1遺伝子のサイレンシングは光合成の改善に利用されている[166]。コムギでは、アミロース含量の増大を目的としてSBEIIa、SBEIIb遺伝子が標的となっているほか[167]、六倍体品種の機能ゲノミクス研究にRNAiが、そしてLr21遺伝子によってもたらされるコムギ赤さび病耐性機構の研究のためにvirus-induced gene silencing(VIGS、RNAiの一種)が利用されている[168]。
その他の作物
タバコでは発がん性を有する可能性が高い物質の前駆体を減少させる取り組みが行われている[169]。また実験室レベルでは、一般的な植物ウイルスに対する耐性の付与などの改変が行われている[170]。ケシによる非麻薬性アルカロイドの産生も試みられている[171]。
殺虫剤
殺虫剤としてのRNAiの開発が行われており、遺伝子操作や外部からの投与など複数のアプローチがとられている[2]。一部の昆虫の中腸の細胞は、environmental RNAiと呼ばれる過程でdsRNA分子を取り込むことがある[172]。一部の昆虫では、その効果は昆虫の体中に広がり、全身に作用する[173]。ヒトがこうした殺虫性のdsRNAを発現する遺伝子組み換え作物を消費することで予想される曝露量の数百万倍の量を曝露した場合でも、動物に悪影響はみられない[174]。
RNAiの影響は鱗翅目(チョウやガ)の生物種によってさまざまに異なり、おそらくそれは唾液や消化液のRNA分解能力の違いによるものである。cotton bollworm、シロイチモジヨトウ、ニカメイガでは給餌によるRNAi感受性は示されていない[2]。
RNAiに対する耐性は広域的、すなわちある配列に対する耐性が他のdsRNA配列に対する耐性も付与する可能性が示唆されている。あるウエスタンコーンルートワームの実験室集団では、腸からのDvSnf7を標的としたdsRNAの取り込みが起こらないために耐性が生じている[175]。DvSnf7に対する他のdsRNA配列を試した際にも有効性は見られず、耐性管理は単純にdsRNAの配列を切り替えるだけでは困難であることが示唆される。バチルス・チューリンゲンシスBacillus thuringiensis由来のCryタンパク質とRNAiなど複数の戦略を併用することで、耐性の出現は遅らせることができると考えられている[2][176]。
ショウジョウバエ属Drosophila spp.、カイコガBombyx mori、トノサマバッタ属Locusta spp、スポドプテラ属Spodoptera spp.、コクヌストモドキTribolium castaneum、トビイロウンカNilaparvata lugens、オオタバコガHelicoverpa armigera、セイヨウミツバチApis melliferaは、昆虫の特定の系統内でRNAiがどのように機能するかを知るために広く利用されているモデルである。イエバエMusca domesticaはAgo2遺伝子を2つ持ち、ツェツェバエGlossina morsitansは3つ持つことが知られている[177][178]。miRNA経路に関しては、ロシアコムギアブラムシDiuraphis noxiaは2つのAgo1、M. domesticaは2つのDcr1、エンドウヒゲナガアブラムシAcyrthosiphon pisumはAgo1、Loqs、Dcr1を2つずつ持ち、Pashaを4つ持つ。piRNAに関しては、G. morsitansとA. pisumは2つまたは3つのAgo3を持つ[178]。こうした研究により、将来的な殺虫剤開発の標的や、作用機序、他の殺虫剤に対する耐性の理由などが明らかとなった[178]。
トランスジェニック植物
トランスジェニック作物はdsRNAを発現するように作製されており、その配列は標的害虫の重要な遺伝子をサイレンシングするよう慎重に選ばれたものである。こうしたdsRNAは、特定の遺伝子配列を発現する昆虫のみに影響を与えるよう設計されている。2009年の実証実験では、RNAが4種のショウジョウバエのうちいずれか1種のみに対して殺虫作用を示し、他の3種には害を及ぼさないことが示された[2]。
2012年、シンジェンタはベルギーのRNAi企業Devgenを5億2200万ドルで買収し、モンサントはアルナイラム・ファーマシューティカルズから知的財産権の独占権を2920万ドルで取得した。ペルー・リマの国際ポテトセンターでは、幼虫によるサツマイモの食害が世界的に問題となっているアリモドキゾウムシに対する標的遺伝子の探索が行われている。他にも、アリ、毛虫、pollen beetleなどの遺伝子のサイレンシングが試みられている。モンサントは、アメリカ合衆国だけで毎年10億ドルの被害をもたらしているウエスタンコーンルートワームのSnf7遺伝子を標的としたdsRNAを発現する、トランスジェニックトウモロコシの種子を初めて販売することとなる可能性が高い。2012年の論文では、Snf7のサイレンシングは幼虫の成長を止め、数日以内に死滅させることが示されている。2013年に同チームは、このRNAが他の生物種に影響を与えることはほとんどないことを示した[2]。
外部からの投与
dsRNAは遺伝子組み換え以外の方法でも供給することができる。1つのアプローチは、灌漑用水への添加である。RNA分子は植物の維管束系へ吸収され、その植物を食べる昆虫を殺す。他のアプローチは、従来の農薬のような形でのdsRNAの噴霧である。こうした方法は耐性の出現に対してより早く対応することができるが、dsRNAの低コストでの作製法を必要とし、そうした手法は現在のところ存在しない[2]。
ゲノムスケールでのスクリーニング
ゲノムスケールでのRNAi研究は、ハイスループットスクリーニング(HTS)技術に依存している。RNAi HTS技術はゲノムワイドな機能喪失スクリーニングを可能にし、特定の表現型と関係する遺伝子の同定に広く利用されている。この技術は、遺伝子発現マイクロアレイや一塩基多型発見プラットフォームといったゲノミクスの第一の波に続く、第二の波となる可能性があるとの評価がなされている[179]。ゲノムスケールでのRNAiスクリーニングの大きな利点の1つは、数千もの遺伝子を同時に調査することができる点である。ゲノムスケールでのRNAiスクリーニングでは1つの実験から大量のデータが生み出されるため、データ生成量の爆発的増加をもたらしている。こうした巨大なデータセットの処理は基本的な課題となっており、適切な統計学やバイオインフォマティクス的手法を必要とする。細胞ベースのRNAiスクリーニングの基本的過程は、RNAiライブラリや頑強で安定した細胞種の選択、RNAi試薬によるトランスフェクション、処理とインキュベーション、シグナル検出、重要な遺伝子または治療標的遺伝子の解析、同定などからなる[180]。
歴史

RNAi過程は、それがRNAと関連した機構であることが知られる前には、"co-suppression"や"quelling"と呼ばれていた。RNAiの発見に先立って、トランスジェニック植物で発現させたアンチセンスRNAによる転写阻害がまず観察され[182]、そして1990年代初頭のアメリカ合衆国とオランダの植物学者によって行われた実験による予想外の結果の報告[183]によってより直接的にRNAiの発見への道が開かれた。この実験ではペチュニアの花の色の変化が試みられ、研究者らは正常なピンクまたはスミレ色の花のペチュニアに対し、花の色素形成に重要な酵素であるカルコンシンターゼをコードする遺伝子のさらなるコピーを導入した。コピー数の増加による遺伝子の過剰発現によってより濃い色の花となることが予想されたが、実際には一部の花では紫色の色素は薄くなり、そして斑入りのパターンが形成されることもあった。このことは、カルコンシンターゼの活性は状況依存的に大きく低下するか、または抑制されていることを示していた。後に、一部の形質転換体のゲノム中のさまざまな位置で反対向きのプロモーターに隣接して導入遺伝子が挿入された結果、プロモーターの活性化によってアンチセンス転写産物が発現し、遺伝子がサイレンシングされたという説明がなされた。初期のRNAiの観察の他の例としてはアカパンカビNeurospora crassaの研究のものがあるが[184]、これが関連した現象であるとはすぐには認識されなかった。植物での現象のさらなる研究によって、ダウンレギュレーションはmRNAの分解率の上昇を介した、遺伝子発現の転写後阻害によるものであることが示された[185]。この現象は"co-suppression of gene expression"と呼ばれたが、その分子機構はいまだ不明のままであった[186]。
それから間もなく、ウイルス病に対する植物の耐性の改善に取り組んでいた植物ウイルス学者らによって、類似した予想外の現象が観察された。ウイルス特異的タンパク質を発現する植物はウイルス感染に対するトレランス(tolerance)や抵抗性(resistance)の向上がみられることは知られていたが、ウイルスRNA配列の短い非コード領域のみを持つ植物も同様の防御レベルを示すという予想外の結果が得られた。研究者らは導入遺伝子によって産生されるウイルスRNAがウイルスの複製を阻害すると考えた[187]。逆実験として、植物遺伝子の短い配列を導入したウイルスは、感染した植物で標的遺伝子を抑制することが示された[188]。この現象は"virus-induced gene silencing"(VIGS)と呼ばれ、これらの現象はまとめて"post transcriptional gene silencing"と呼ばれるようになった[189]。
こうした植物における初期の観察の後、他の生物種におけるこうした現象の探索が行われた[190][191]。クレイグ・メローとアンドリュー・ファイアーによる1998年のNature誌の論文では、C. elegansに二本鎖RNAを注入した後に強力な遺伝子サイレンシング効果がみられることが報告された[192]。彼らは筋タンパク質の産生の調節の研究の際に、mRNAやアンチセンスRNAの注入はタンパク質産生に影響を及ぼさないが、二本鎖RNAの注入によって標的遺伝子がサイレンシングされることを発見した。この研究をもとに、RNAiという用語を作った。この発見は、この現象の原因となる因子を初めて同定したこととなる。ファイアーとメローは2006年にノーベル生理学・医学賞を受賞した[34]。
出典
- ^ a b c “RNA interference: concept to reality in crop improvement”. Planta 239 (3): 543–64. (March 2014). doi:10.1007/s00425-013-2019-5. PMID 24402564.
- ^ a b c d e f g “A lethal dose of RNA”. Science 341 (6147): 732–3. (August 2013). Bibcode: 2013Sci...341..732K. doi:10.1126/science.341.6147.732. PMID 23950525.
- ^ “Structural basis for double-stranded RNA processing by Dicer”. Science 311 (5758): 195–8. (January 2006). Bibcode: 2006Sci...311..195M. doi:10.1126/science.1121638. PMID 16410517.
- ^ “RNA interference: the molecular immune system”. Journal of Molecular Histology 35 (6): 545–53. (August 2004). doi:10.1007/s10735-004-2192-8. PMID 15614608.
- ^ “Role for a bidentate ribonuclease in the initiation step of RNA interference”. Nature 409 (6818): 363–6. (January 2001). Bibcode: 2001Natur.409..363B. doi:10.1038/35053110. PMID 11201747.

- ^ “On the road to reading the RNA-interference code”. Nature 457 (7228): 396–404. (January 2009). Bibcode: 2009Natur.457..396S. doi:10.1038/nature07754. PMID 19158785.
“RNAi: double-stranded RNA directs the ATP-dependent cleavage of mRNA at 21 to 23 nucleotide intervals”. Cell 101 (1): 25–33. (March 2000). doi:10.1016/S0092-8674(00)80620-0. PMID 10778853.
“The contributions of dsRNA structure to Dicer specificity and efficiency”. RNA 11 (5): 674–82. (May 2005). doi:10.1261/rna.7272305. PMC 1370754. PMID 15811921.
“The promises and pitfalls of RNA-interference-based therapeutics”. Nature 457 (7228): 426–33. (January 2009). Bibcode: 2009Natur.457..426C. doi:10.1038/nature07758. PMC 2702667. PMID 19158789. - ^ a b “A computational study of off-target effects of RNA interference”. Nucleic Acids Research 33 (6): 1834–47. (2005). doi:10.1093/nar/gki324. PMC 1072799. PMID 15800213.
- ^ a b “Anatomy of RISC: how do small RNAs and chaperones activate Argonaute proteins?”. Wiley Interdisciplinary Reviews: RNA 7 (5): 637–60. (September 2016). doi:10.1002/wrna.1356. PMC 5084781. PMID 27184117.
- ^ “TAF11 Assembles the RISC Loading Complex to Enhance RNAi Efficiency”. Molecular Cell 59 (5): 807–18. (September 2015). doi:10.1016/j.molcel.2015.07.006. PMC 4560963. PMID 26257286.
- ^ “RNA-dependent RNA polymerases, viruses, and RNA silencing”. Science 296 (5571): 1270–3. (May 2002). Bibcode: 2002Sci...296.1270A. doi:10.1126/science.1069132. PMID 12016304.
- ^ “The microRNA Machinery”. Advances in Experimental Medicine and Biology 887: 15–30. (2015). doi:10.1007/978-3-319-22380-3_2. ISBN 978-3-319-22379-7. PMID 26662984.
- ^ “The functions of microRNAs in plants”. Frontiers in Bioscience 12: 3975–82. (May 2007). doi:10.2741/2364. PMC 2851543. PMID 17485351.
“A developmental view of microRNA function”. Trends in Biochemical Sciences 32 (4): 189–97. (April 2007). doi:10.1016/j.tibs.2007.02.006. PMID 17350266. - ^ “MicroRNA biogenesis: isolation and characterization of the microprocessor complex”. MicroRNA Protocols. Methods in Molecular Biology. 342. (2006). pp. 33–47. doi:10.1385/1-59745-123-1:33. ISBN 978-1-59745-123-9. PMID 16957365
- ^ “Identification of virus-encoded microRNAs”. Science 304 (5671): 734–6. (April 2004). Bibcode: 2004Sci...304..734P. doi:10.1126/science.1096781. PMID 15118162.
- ^ “VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets”. Database 2014: bau103. (1 January 2014). doi:10.1093/database/bau103. PMC 4224276. PMID 25380780.
- ^ “Repression of protein synthesis by miRNAs: how many mechanisms?”. Trends Cell Biol 17 (3): 118–26. (2007). doi:10.1016/j.tcb.2006.12.007. PMID 17197185.
- ^ “Distinct roles for Argonaute proteins in small RNA-directed RNA cleavage pathways”. Genes Dev 18 (14): 1655–66. (2004). doi:10.1101/gad.1210204. PMC 478188. PMID 15231716.
- ^ “Distinct roles for Drosophila Dicer-1 and Dicer-2 in the siRNA/miRNA silencing pathways”. Cell 117 (1): 69–81. (2004). doi:10.1016/S0092-8674(04)00261-2. PMID 15066283.
- ^ “miRBase”. mirbase.org. 2022年3月6日閲覧。
- ^ a b “Most mammalian mRNAs are conserved targets of microRNAs”. Genome Res. 19 (1): 92–105. (2009). doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ^ “Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs”. Nature 433 (7027): 769–73. (February 2005). Bibcode: 2005Natur.433..769L. doi:10.1038/nature03315. PMID 15685193.
- ^ “Widespread changes in protein synthesis induced by microRNAs”. Nature 455 (7209): 58–63. (September 2008). Bibcode: 2008Natur.455...58S. doi:10.1038/nature07228. PMID 18668040.
- ^ “The impact of microRNAs on protein output”. Nature 455 (7209): 64–71. (September 2008). Bibcode: 2008Natur.455...64B. doi:10.1038/nature07242. PMC 2745094. PMID 18668037.
- ^ “Mechanisms and role of microRNA deregulation in cancer onset and progression”. Genetics and Molecular Biology 34 (3): 363–70. (July 2011). doi:10.1590/S1415-47572011000300001. PMC 3168173. PMID 21931505.
- ^ “Epigenetic reduction of DNA repair in progression to gastrointestinal cancer”. World Journal of Gastrointestinal Oncology 7 (5): 30–46. (May 2015). doi:10.4251/wjgo.v7.i5.30. PMC 4434036. PMID 25987950.
- ^ “Micro spies from the brain to the periphery: new clues from studies on microRNAs in neuropsychiatric disorders”. Frontiers in Cellular Neuroscience 8: 75. (2014). doi:10.3389/fncel.2014.00075. PMC 3949217. PMID 24653674.
- ^ “The Emerging Role of microRNAs in Schizophrenia and Autism Spectrum Disorders”. Frontiers in Psychiatry 3: 39. (2012). doi:10.3389/fpsyt.2012.00039. PMC 3336189. PMID 22539927.
- ^ “MicroRNA and Posttranscriptional Dysregulation in Psychiatry”. Biological Psychiatry 78 (4): 231–9. (August 2015). doi:10.1016/j.biopsych.2014.12.009. PMID 25636176.
- ^ a b “RDE-4 preferentially binds long dsRNA and its dimerization is necessary for cleavage of dsRNA to siRNA”. RNA 12 (5): 807–18. (2006). doi:10.1261/rna.2338706. PMC 1440910. PMID 16603715.
- ^ “R2D2, a bridge between the initiation and effector steps of the Drosophila RNAi pathway”. Science 301 (5641): 1921–5. (September 2003). Bibcode: 2003Sci...301.1921L. doi:10.1126/science.1088710. PMID 14512631.
- ^ “Molecular biology. Amplified silencing”. Science 315 (5809): 199–200. (January 2007). doi:10.1126/science.1138030. PMID 17218517.
- ^ “Distinct populations of primary and secondary effectors during RNAi in C. elegans”. Science 315 (5809): 241–4. (January 2007). Bibcode: 2007Sci...315..241P. doi:10.1126/science.1132839. PMID 17124291.
- ^ “Secondary siRNAs result from unprimed RNA synthesis and form a distinct class”. Science 315 (5809): 244–7. (January 2007). Bibcode: 2007Sci...315..244S. doi:10.1126/science.1136699. PMID 17158288.
- ^ a b c “Advanced Information: RNA interference”. The Nobel Prize in Physiology or Medicine 2006. 2007年1月20日時点のオリジナルよりアーカイブ。2007年1月25日閲覧。
- ^ “Human RISC couples microRNA biogenesis and posttranscriptional gene silencing”. Cell 123 (4): 631–40. (November 2005). doi:10.1016/j.cell.2005.10.022. PMID 16271387.
- ^ a b Molecular Cell Biology (5th ed.). WH Freeman: New York, NY. (2004). ISBN 978-0-7167-4366-8
- ^ “Passenger-strand cleavage facilitates assembly of siRNA into Ago2-containing RNAi enzyme complexes”. Cell 123 (4): 607–20. (November 2005). doi:10.1016/j.cell.2005.08.044. PMID 16271386.
- ^ “Cleavage of the siRNA passenger strand during RISC assembly in human cells”. EMBO Reports 7 (3): 314–20. (March 2006). doi:10.1038/sj.embor.7400637. PMC 1456892. PMID 16439995.
- ^ a b “Kinetic analysis of the RNAi enzyme complex”. Nature Structural & Molecular Biology 11 (7): 599–606. (July 2004). doi:10.1038/nsmb780. PMID 15170178.
- ^ “Asymmetry in the assembly of the RNAi enzyme complex”. Cell 115 (2): 199–208. (October 2003). doi:10.1016/S0092-8674(03)00759-1. PMID 14567917.
- ^ “Short interfering RNA strand selection is independent of dsRNA processing polarity during RNAi in Drosophila”. Current Biology 16 (5): 530–5. (March 2006). doi:10.1016/j.cub.2006.01.061. PMID 16527750.
- ^ “A protein sensor for siRNA asymmetry”. Science 306 (5700): 1377–80. (November 2004). Bibcode: 2004Sci...306.1377T. doi:10.1126/science.1102755. PMID 15550672.
- ^ “Structural basis for 5'-end-specific recognition of guide RNA by the A. fulgidus Piwi protein”. Nature 434 (7033): 666–70. (March 2005). Bibcode: 2005Natur.434..666M. doi:10.1038/nature03514. PMC 4694588. PMID 15800629.
- ^ “mRNA translation is not a prerequisite for small interfering RNA-mediated mRNA cleavage”. Differentiation 73 (6): 287–93. (2005). doi:10.1111/j.1432-0436.2005.00029.x. PMID 16138829.
- ^ “Uncoupling of RNAi from active translation in mammalian cells”. RNA 11 (1): 38–44. (2005). doi:10.1261/rna.7158605. PMC 1370689. PMID 15574516.
- ^ “Argonaute 2/RISC resides in sites of mammalian mRNA decay known as cytoplasmic bodies”. Nat Cell Biol 7 (6): 633–6. (2005). doi:10.1038/ncb1265. PMID 15908945.
- ^ “GW bodies, microRNAs and the cell cycle”. Cell Cycle 5 (3): 242–5. (2006). doi:10.4161/cc.5.3.2410. PMID 16418578.
- ^ “Disruption of P bodies impairs mammalian RNA interference”. Nat Cell Biol 7 (12): 1267–74. (2005). doi:10.1038/ncb1334. PMID 16284622.
- ^ “An RNA-directed nuclease mediates post-transcriptional gene silencing in Drosophila cells”. Nature 404 (6775): 293–6. (2000). Bibcode: 2000Natur.404..293H. doi:10.1038/35005107. PMID 10749213.

- ^ “Chromosome organization and chromatin modification: influence on genome function and evolution”. Cytogenetic and Genome Research 114 (2): 96–125. (2006). doi:10.1159/000093326. PMID 16825762.
- ^ “RNAi-mediated targeting of heterochromatin by the RITS complex”. Science 303 (5658): 672–6. (January 2004). Bibcode: 2004Sci...303..672V. doi:10.1126/science.1093686. PMC 3244756. PMID 14704433.
- ^ “Argonaute slicing is required for heterochromatic silencing and spreading”. Science 313 (5790): 1134–7. (August 2006). Bibcode: 2006Sci...313.1134I. doi:10.1126/science.1128813. PMID 16931764.
- ^ “Regulation of heterochromatic silencing and histone H3 lysine-9 methylation by RNAi”. Science 297 (5588): 1833–7. (September 2002). Bibcode: 2002Sci...297.1833V. doi:10.1126/science.1074973. PMID 12193640.
- ^ “RNA interference is required for normal centromere function in fission yeast”. Chromosome Research 11 (2): 137–46. (2003). doi:10.1023/A:1022815931524. PMID 12733640.
- ^ “Small dsRNAs induce transcriptional activation in human cells”. Proceedings of the National Academy of Sciences of the United States of America 103 (46): 17337–42. (November 2006). Bibcode: 2006PNAS..10317337L. doi:10.1073/pnas.0607015103. PMC 1859931. PMID 17085592.
- ^ “RITS acts in cis to promote RNA interference-mediated transcriptional and post-transcriptional silencing”. Nature Genetics 36 (11): 1174–80. (November 2004). doi:10.1038/ng1452. PMID 15475954.
- ^ “RNA-dependent RNA polymerase is an essential component of a self-enforcing loop coupling heterochromatin assembly to siRNA production”. Proceedings of the National Academy of Sciences of the United States of America 102 (1): 152–7. (January 2005). Bibcode: 2005PNAS..102..152S. doi:10.1073/pnas.0407641102. PMC 544066. PMID 15615848.
- ^ “The assembly and maintenance of heterochromatin initiated by transgene repeats are independent of the RNA interference pathway in mammalian cells”. Molecular and Cellular Biology 26 (11): 4028–40. (June 2006). doi:10.1128/MCB.02189-05. PMC 1489094. PMID 16705157.
- ^ “RNA editing by adenosine deaminases that act on RNA”. Annual Review of Biochemistry 71: 817–46. (2002). doi:10.1146/annurev.biochem.71.110601.135501. PMC 1823043. PMID 12045112.
- ^ “Double-stranded RNA as a template for gene silencing”. Cell 101 (3): 235–8. (April 2000). doi:10.1016/S0092-8674(02)71133-1. PMID 10847677.
- ^ “RNA editing of a miRNA precursor”. RNA 10 (8): 1174–7. (August 2004). doi:10.1261/rna.7350304. PMC 1370607. PMID 15272117.
- ^ a b “Modulation of microRNA processing and expression through RNA editing by ADAR deaminases”. Nature Structural & Molecular Biology 13 (1): 13–21. (January 2006). doi:10.1038/nsmb1041. PMC 2950615. PMID 16369484.
- ^ “ADAR1 RNA deaminase limits short interfering RNA efficacy in mammalian cells”. The Journal of Biological Chemistry 280 (5): 3946–53. (February 2005). doi:10.1074/jbc.M407876200. PMC 2947832. PMID 15556947.
- ^ “Editor meets silencer: crosstalk between RNA editing and RNA interference”. Nature Reviews Molecular Cell Biology 7 (12): 919–31. (December 2006). doi:10.1038/nrm2061. PMC 2953463. PMID 17139332.
- ^ a b c “Anti-viral RNA silencing: do we look like plants ?”. Retrovirology 3 (1): 3. (2006). doi:10.1186/1742-4690-3-3. PMC 1363733. PMID 16409629.
- ^ “RNA-directed transcriptional gene silencing in plants can be inherited independently of the RNA trigger and requires Met1 for maintenance”. Current Biology 11 (10): 747–57. (May 2001). doi:10.1016/S0960-9822(01)00226-3. PMID 11378384.
- ^ “MicroRNAs control translation initiation by inhibiting eukaryotic initiation factor 4E/cap and poly(A) tail function”. Proceedings of the National Academy of Sciences of the United States of America 102 (47): 16961–6. (November 2005). Bibcode: 2005PNAS..10216961H. doi:10.1073/pnas.0506482102. PMC 1287990. PMID 16287976.
- ^ “Tests of cytoplasmic RNA interference (RNAi) and construction of a tetracycline-inducible T7 promoter system in Trypanosoma cruzi”. Molecular and Biochemical Parasitology 133 (2): 175–86. (February 2004). doi:10.1016/j.molbiopara.2003.10.005. PMID 14698430.
- ^ “Improvements in transfection efficiency and tests of RNA interference (RNAi) approaches in the protozoan parasite Leishmania”. Molecular and Biochemical Parasitology 128 (2): 217–28. (May 2003). doi:10.1016/S0166-6851(03)00079-3. PMID 12742588.
- ^ “Lineage-specific loss and divergence of functionally linked genes in eukaryotes”. Proceedings of the National Academy of Sciences of the United States of America 97 (21): 11319–24. (October 2000). Bibcode: 2000PNAS...9711319A. doi:10.1073/pnas.200346997. PMC 17198. PMID 11016957.
- ^ “RNAi in budding yeast”. Science 326 (5952): 544–550. (October 2009). Bibcode: 2009Sci...326..544D. doi:10.1126/science.1176945. PMC 3786161. PMID 19745116.
- ^ “Evolution and diversification of RNA silencing proteins in fungi”. Journal of Molecular Evolution 63 (1): 127–35. (July 2006). Bibcode: 2006JMolE..63..127N. doi:10.1007/s00239-005-0257-2. PMID 16786437.
- ^ “Translational repression is sufficient for gene silencing by bacterial small noncoding RNAs in the absence of mRNA destruction”. Proceedings of the National Academy of Sciences of the United States of America 103 (13): 4858–63. (March 2006). Bibcode: 2006PNAS..103.4858M. doi:10.1073/pnas.0509638103. PMC 1458760. PMID 16549791.
- ^ “A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action”. Biology Direct 1: 7. (March 2006). doi:10.1186/1745-6150-1-7. PMC 1462988. PMID 16545108.
- ^ “Inhibition of viruses by RNA interference”. Virus Genes 32 (3): 299–306. (June 2006). doi:10.1007/s11262-005-6914-0. PMC 7088519. PMID 16732482.
- ^ “Four plant Dicers mediate viral small RNA biogenesis and DNA virus induced silencing”. Nucleic Acids Research 34 (21): 6233–46. (2006). doi:10.1093/nar/gkl886. PMC 1669714. PMID 17090584.
- ^ “Systemic acquired silencing: transgene-specific post-transcriptional silencing is transmitted by grafting from silenced stocks to non-silenced scions”. The EMBO Journal 16 (15): 4738–45. (August 1997). doi:10.1093/emboj/16.15.4738. PMC 1170100. PMID 9303318.
- ^ “RNA silencing as a plant immune system against viruses”. Trends in Genetics 17 (8): 449–59. (August 2001). doi:10.1016/S0168-9525(01)02367-8. PMID 11485817.
- ^ “Suppression of post-transcriptional gene silencing by a plant viral protein localized in the nucleus”. The EMBO Journal 19 (7): 1672–80. (April 2000). doi:10.1093/emboj/19.7.1672. PMC 310235. PMID 10747034.
- ^ “Double-stranded RNA binding may be a general plant RNA viral strategy to suppress RNA silencing”. Journal of Virology 80 (12): 5747–56. (June 2006). doi:10.1128/JVI.01963-05. PMC 1472586. PMID 16731914.
- ^ “A pathogen-inducible endogenous siRNA in plant immunity”. Proceedings of the National Academy of Sciences of the United States of America 103 (47): 18002–7. (November 2006). Bibcode: 2006PNAS..10318002K. doi:10.1073/pnas.0608258103. PMC 1693862. PMID 17071740.
- ^ “Innate immune defense through RNA interference”. Science's STKE 2006 (339): pe27. (June 2006). doi:10.1126/stke.3392006pe27. PMID 16772641.
- ^ “RNAi is an antiviral immune response against a dsRNA virus in Drosophila melanogaster”. Cellular Microbiology 8 (5): 880–9. (May 2006). doi:10.1111/j.1462-5822.2006.00688.x. PMID 16611236.
- ^ “RNA interference directs innate immunity against viruses in adult Drosophila”. Science 312 (5772): 452–4. (April 2006). Bibcode: 2006Sci...312..452W. doi:10.1126/science.1125694. PMC 1509097. PMID 16556799.
- ^ “Animal virus replication and RNAi-mediated antiviral silencing in Caenorhabditis elegans”. Nature 436 (7053): 1040–1043. (August 2005). Bibcode: 2005Natur.436.1040L. doi:10.1038/nature03870. PMC 1388260. PMID 16107851.
- ^ “RNA interference is an antiviral defence mechanism in Caenorhabditis elegans”. Nature 436 (7053): 1044–7. (August 2005). Bibcode: 2005Natur.436.1044W. doi:10.1038/nature03957. PMID 16107852.
- ^ “Antiviral RNA interference in mammalian cells”. Science 342 (6155): 235–8. (October 2013). Bibcode: 2013Sci...342..235M. doi:10.1126/science.1241930. PMC 3853215. PMID 24115438.
- ^ “RNA interference functions as an antiviral immunity mechanism in mammals”. Science 342 (6155): 231–4. (October 2013). Bibcode: 2013Sci...342..231L. doi:10.1126/science.1241911. PMC 3875315. PMID 24115437.
- ^ “The interplay between virus infection and the cellular RNA interference machinery”. FEBS Letters 580 (12): 2896–902. (May 2006). doi:10.1016/j.febslet.2006.02.070. PMC 7094296. PMID 16563388.
- ^ “Interaction of viruses with the mammalian RNA interference pathway”. Virology 344 (1): 151–7. (January 2006). doi:10.1016/j.virol.2005.09.034. PMID 16364746.
- ^ “Is RNA interference involved in intrinsic antiviral immunity in mammals?”. Nature Immunology 7 (6): 563–7. (June 2006). doi:10.1038/ni1352. PMID 16715068.
- ^ “Antiviral silencing in animals”. FEBS Letters 579 (26): 5965–73. (October 2005). doi:10.1016/j.febslet.2005.08.034. PMC 1350842. PMID 16154568.
- ^ “Role of microRNAs in plant and animal development”. Science 301 (5631): 336–8. (July 2003). Bibcode: 2003Sci...301..336C. doi:10.1126/science.1085242. PMID 12869753.
- ^ “The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14”. Cell 75 (5): 843–54. (December 1993). doi:10.1016/0092-8674(93)90529-Y. PMID 8252621.
- ^ “Control of leaf morphogenesis by microRNAs”. Nature 425 (6955): 257–63. (September 2003). Bibcode: 2003Natur.425..257P. doi:10.1038/nature01958. PMID 12931144.
- ^ “Plant microRNA: a small regulatory molecule with big impact”. Developmental Biology 289 (1): 3–16. (January 2006). doi:10.1016/j.ydbio.2005.10.036. PMID 16325172.
- ^ “MicroRNAS and their regulatory roles in plants”. Annual Review of Plant Biology 57: 19–53. (2006). doi:10.1146/annurev.arplant.57.032905.105218. PMID 16669754.
- ^ “microRNAs as oncogenes and tumor suppressors”. Developmental Biology 302 (1): 1–12. (February 2007). doi:10.1016/j.ydbio.2006.08.028. PMID 16989803.
- ^ “On the origin and functions of RNA-mediated silencing: from protists to man”. Current Genetics 50 (2): 81–99. (August 2006). doi:10.1007/s00294-006-0078-x. PMC 2583075. PMID 16691418.
- ^ a b “Comparative genomics and evolution of proteins involved in RNA metabolism”. Nucleic Acids Research 30 (7): 1427–64. (April 2002). doi:10.1093/nar/30.7.1427. PMC 101826. PMID 11917006.
- ^ “Knockdown stands up”. Trends in Biotechnology 21 (1): 2–4. (January 2003). doi:10.1016/S0167-7799(02)00002-1. PMID 12480342.
- ^ “Validation of RNAi Silencing Efficiency Using Gene Array Data shows 18.5% Failure Rate across 429 Independent Experiments”. Molecular Therapy: Nucleic Acids 5 (9): e366. (September 2016). doi:10.1038/mtna.2016.66. PMC 5056990. PMID 27673562.
- ^ “dsCheck: highly sensitive off-target search software for double-stranded RNA-mediated RNA interference”. Nucleic Acids Research 33 (Web Server issue): W589–91. (July 2005). doi:10.1093/nar/gki419. PMC 1160180. PMID 15980542.
- ^ “DEQOR: a web-based tool for the design and quality control of siRNAs”. Nucleic Acids Research 32 (Web Server issue): W113–20. (July 2004). doi:10.1093/nar/gkh408. PMC 441546. PMID 15215362.
- ^ “siDirect: highly effective, target-specific siRNA design software for mammalian RNA interference”. Nucleic Acids Research 32 (Web Server issue): W124–9. (July 2004). doi:10.1093/nar/gkh442. PMC 441580. PMID 15215364.
- ^ “siVirus: web-based antiviral siRNA design software for highly divergent viral sequences”. Nucleic Acids Research 34 (Web Server issue): W448–50. (July 2006). doi:10.1093/nar/gkl214. PMC 1538817. PMID 16845046.
- ^ “Induction of the interferon response by siRNA is cell type- and duplex length-dependent”. RNA 12 (6): 988–93. (June 2006). doi:10.1261/rna.2340906. PMC 1464853. PMID 16611941.
- ^ “Absence of non-specific effects of RNA interference triggered by long double-stranded RNA in mouse oocytes”. Developmental Biology 286 (2): 464–71. (October 2005). doi:10.1016/j.ydbio.2005.08.015. PMID 16154556.
- ^ “A system for stable expression of short interfering RNAs in mammalian cells”. Science 296 (5567): 550–3. (April 2002). Bibcode: 2002Sci...296..550B. doi:10.1126/science.1068999. hdl:1874/15573. PMID 11910072.
- ^ “CRE recombinase-inducible RNA interference mediated by lentiviral vectors”. Proceedings of the National Academy of Sciences of the United States of America 101 (19): 7347–51. (May 2004). Bibcode: 2004PNAS..101.7347T. doi:10.1073/pnas.0402107101. PMC 409921. PMID 15123829.
- ^ “Cre-lox-regulated conditional RNA interference from transgenes”. Proceedings of the National Academy of Sciences of the United States of America 101 (28): 10380–5. (July 2004). Bibcode: 2004PNAS..10110380V. doi:10.1073/pnas.0403954101. PMC 478580. PMID 15240889.
- ^ “Design of a genome-wide siRNA library using an artificial neural network”. Nature Biotechnology 23 (8): 995–1001. (August 2005). doi:10.1038/nbt1118. PMID 16025102.
- ^ “Prediction of siRNA knockdown efficiency using artificial neural network models”. Biochemical and Biophysical Research Communications 336 (2): 723–8. (October 2005). doi:10.1016/j.bbrc.2005.08.147. PMID 16153609.
- ^ “High-Throughput RNA Interference in Functional Genomics”. RNA Towards Medicine. Handbook of Experimental Pharmacology. 173. (2006). pp. 97–104. doi:10.1007/3-540-27262-3_5. ISBN 978-3-540-27261-8. PMID 16594612
- ^ “Functional genomics using high-throughput RNA interference”. Drug Discovery Today 10 (3): 205–12. (February 2005). doi:10.1016/S1359-6446(04)03352-5. hdl:11858/00-001M-0000-0010-86E7-8. PMID 15708535.
- ^ “Testing the efficacy of RNA interference in Haemonchus contortus”. International Journal for Parasitology 36 (7): 801–10. (June 2006). doi:10.1016/j.ijpara.2005.12.004. PMID 16469321.
- ^ “RNA interference in parasitic helminths: current situation, potential pitfalls and future prospects”. Parasitology 134 (Pt 5): 609–19. (May 2007). doi:10.1017/S0031182006002071. PMID 17201997.
- ^ a b “A brief history of RNAi: the silence of the genes”. FASEB Journal 20 (9): 1293–9. (July 2006). doi:10.1096/fj.06-6014rev. PMID 16816104.
- ^ “The Nobel Prize in Physiology or Medicine 2006”. nobelprize.org (2006年10月2日). 2017年10月30日閲覧。
- ^ “Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells”. Nature 411 (6836): 494–498. (2001). Bibcode: 2001Natur.411..494E. doi:10.1038/35078107. PMID 11373684.
- ^ “RNA interference in adult mice”. Nature 418 (6893): 38–9. (July 2002). Bibcode: 2002Natur.418...38M. doi:10.1038/418038a. PMID 12097900.

- ^ “siRNA-based approaches in cancer therapy”. Cancer Gene Therapy 13 (9): 819–29. (September 2006). doi:10.1038/sj.cgt.7700931. PMID 16424918.
- ^ “Small RNA: can RNA interference be exploited for therapy?”. Lancet 362 (9393): 1401–3. (October 2003). doi:10.1016/s0140-6736(03)14637-5. PMID 14585643.
- ^ Sah D (2006). “Therapeutic potential of RNA interference for neurological disorders”. Life Sci 79 (19): 1773–80. doi:10.1016/j.lfs.2006.06.011. PMID 16815477.
- ^ “Evidence of RNAi in humans from systemically administered siRNA via targeted nanoparticles”. Nature 464 (7291): 1067–70. (April 2010). Bibcode: 2010Natur.464.1067D. doi:10.1038/nature08956. PMC 2855406. PMID 20305636.
- ^ “RNAi-mediated oncogene silencing confers resistance to crown gall tumorigenesis”. Proceedings of the National Academy of Sciences of the United States of America 98 (23): 13437–42. (November 2001). Bibcode: 2001PNAS...9813437E. doi:10.1073/pnas.241276898. PMC 60889. PMID 11687652.
- ^ “Schistosoma mansoni: evaluation of an RNAi-based treatment targeting HGPRTase gene”. Experimental Parasitology 118 (4): 619–23. (April 2008). doi:10.1016/j.exppara.2007.11.017. PMID 18237732.
- ^ “Lentiviral-mediated silencing of SOD1 through RNA interference retards disease onset and progression in a mouse model of ALS”. Nature Medicine 11 (4): 423–8. (April 2005). doi:10.1038/nm1207. PMID 15768028.
- ^ “Short interfering RNA (siRNA) targeting the Lyn kinase induces apoptosis in primary, and drug-resistant, BCR-ABL1(+) leukemia cells”. Nature Medicine 10 (11): 1187–9. (November 2004). doi:10.1038/nm1127. PMID 15502840.
- ^ “Effective relief of neuropathic pain by adeno-associated virus-mediated expression of a small hairpin RNA against GTP cyclohydrolase 1”. Molecular Pain 5: 1744–8069–5–67. (November 2009). doi:10.1186/1744-8069-5-67. PMC 2785765. PMID 19922668.
- ^ “Knockdown of orexin type 1 receptor in rat locus coeruleus increases REM sleep during the dark period”. The European Journal of Neuroscience 32 (9): 1528–36. (November 2010). doi:10.1111/j.1460-9568.2010.07401.x. PMC 3058252. PMID 21089218.
- ^ “Size selective recognition of siRNA by an RNA silencing suppressor”. Cell 115 (7): 799–811. (December 2003). doi:10.1016/s0092-8674(03)00984-x. PMID 14697199.
- ^ “RNA interference as an antiviral approach: targeting HIV-1”. Current Opinion in Molecular Therapeutics 6 (2): 141–5. (April 2004). PMID 15195925.
- ^ “Selective silencing of viral gene expression in HPV-positive human cervical carcinoma cells treated with siRNA, a primer of RNA interference”. Oncogene 21 (39): 6041–8. (September 2002). doi:10.1038/sj.onc.1205878. PMID 12203116.
- ^ “Silencing of hepatitis A virus infection by small interfering RNAs”. Journal of Virology 80 (11): 5599–610. (June 2006). doi:10.1128/jvi.01773-05. PMC 1472172. PMID 16699041.
- ^ “A retrovirus-based system to stably silence hepatitis B virus genes by RNA interference”. Biotechnology Letters 28 (20): 1679–85. (October 2006). doi:10.1007/s10529-006-9138-z. PMID 16900331.
- ^ “Construction of influenza virus siRNA expression vectors and their inhibitory effects on multiplication of influenza virus”. Avian Diseases 49 (4): 562–73. (December 2005). doi:10.1637/7365-041205R2.1. PMID 16405000.
- ^ “Gene silencing: a therapeutic approach to combat influenza virus infections”. Future Microbiology 10 (1): 131–40. (2015). doi:10.2217/fmb.14.94. PMID 25598342.
- ^ “Small interfering RNA targeting the nonstructural gene 1 transcript inhibits influenza A virus replication in experimental mice”. Nucleic Acid Therapeutics 22 (6): 414–22. (December 2012). doi:10.1089/nat.2012.0359. PMID 23062009.
- ^ a b c “Advancements in Nucleic Acid Based Therapeutics against Respiratory Viral Infections”. Journal of Clinical Medicine 8 (1): 6. (December 2018). doi:10.3390/jcm8010006. PMC 6351902. PMID 30577479.
- ^ “Inhibition of Measles virus multiplication in cell culture by RNA interference”. Acta Virologica 49 (4): 227–34. (2005). PMID 16402679.
- ^ “VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets”. Database 2014. (2014). doi:10.1093/database/bau103. PMC 4224276. PMID 25380780.
- ^ “Suppression of chemokine receptor expression by RNA interference allows for inhibition of HIV-1 replication, by Martínez et al”. AIDS 17 Suppl 4: S103–5. (2003). PMID 15080188.
- ^ “Silencing of disease-related genes by small interfering RNAs”. Current Molecular Medicine 4 (5): 507–17. (August 2004). doi:10.2174/1566524043360492. PMID 15267222.
- ^ “RNA interference is a functional pathway with therapeutic potential in human myeloid leukemia cell lines”. Cancer Gene Therapy 10 (2): 125–33. (February 2003). doi:10.1038/sj.cgt.7700544. PMID 12536201.
- ^ “CXCR4 knockdown by small interfering RNA abrogates breast tumor growth in vivo”. Cancer Gene Therapy 12 (1): 84–9. (January 2005). doi:10.1038/sj.cgt.7700770. PMID 15472715.
- ^ “Targeting BACE1 with siRNAs ameliorates Alzheimer disease neuropathology in a transgenic model”. Nature Neuroscience 8 (10): 1343–9. (October 2005). doi:10.1038/nn1531. PMID 16136043.
- ^ “Allele-specific RNAi mitigates phenotypic progression in a transgenic model of Alzheimer's disease”. Molecular Therapy 17 (9): 1563–73. (September 2009). doi:10.1038/mt.2009.123. PMC 2835271. PMID 19532137.
- ^ “Silencing of CDK5 reduces neurofibrillary tangles in transgenic alzheimer's mice”. The Journal of Neuroscience 30 (42): 13966–76. (October 2010). doi:10.1523/jneurosci.3637-10.2010. PMC 3003593. PMID 20962218.
- ^ “Viral-based modelling and correction of neurodegenerative diseases by RNA interference”. Gene Therapy 13 (6): 487–95. (March 2006). doi:10.1038/sj.gt.3302690. PMID 16319945.
- ^ “RNA interference improves motor and neuropathological abnormalities in a Huntington's disease mouse model”. Proceedings of the National Academy of Sciences of the United States of America 102 (16): 5820–5. (April 2005). Bibcode: 2005PNAS..102.5820H. doi:10.1073/pnas.0501507102. PMC 556303. PMID 15811941.
- ^ “RNAi medicine for the brain: progresses and challenges”. Human Molecular Genetics 20 (R1): R21–7. (April 2011). doi:10.1093/hmg/ddr137. PMC 3095054. PMID 21459775.
- ^ a b c “Delivery materials for siRNA therapeutics”. Nature Materials 12 (11): 967–77. (November 2013). Bibcode: 2013NatMa..12..967K. doi:10.1038/nmat3765. PMID 24150415.
- ^ a b c “Knocking down disease: a progress report on siRNA therapeutics”. Nature Reviews Genetics 16 (9): 543–52. (September 2015). doi:10.1038/nrg3978. PMC 4756474. PMID 26281785.
- ^ “RNA interference may result in unexpected phenotypes in Caenorhabditis elegans”. Nucleic Acids Research 47 (8): 3957–3969. (May 2019). doi:10.1093/nar/gkz154. PMC 6486631. PMID 30838421.
- ^ a b c d e f g “Silencing or stimulation? siRNA delivery and the immune system”. Annual Review of Chemical and Biomolecular Engineering 2: 77–96. (2011). doi:10.1146/annurev-chembioeng-061010-114133. PMID 22432611.
- ^ “FDA concludes Arctic Apples and Innate Potatoes are safe for consumption” 2017年9月29日閲覧。
- ^ Herrero, Mario; (ORCID 0000-0002-7741-5090); Thornton, Philip K.; (ORCID 0000-0002-1854-0182); Mason-D’Croz, Daniel; (ORCID 0000-0003-0673-2301); Palmer, Jeda; Benton, Tim G. et al. (2020). “Innovation can accelerate the transition towards a sustainable food system”. Nature Food (Nature Portfolio) 1 (5): 266–272. doi:10.1038/s43016-020-0074-1. ISSN 2662-1355.
- ^ “Engineering cottonseed for use in human nutrition by tissue-specific reduction of toxic gossypol”. Proceedings of the National Academy of Sciences of the United States of America 103 (48): 18054–9. (November 2006). doi:10.1073/pnas.0605389103. PMC 1838705. PMID 17110445.
- ^ “Generation of cyanogen-free transgenic cassava”. Planta 217 (3): 367–73. (July 2003). doi:10.1007/s00425-003-1005-8. PMID 14520563.
- ^ “Design of tomato fruits with reduced allergenicity by dsRNAi-mediated inhibition of ns-LTP (Lyc e 3) expression”. Plant Biotechnology Journal 4 (2): 231–42. (March 2006). doi:10.1111/j.1467-7652.2005.00175.x. PMID 17177799.
- ^ “Engineering plants with increased levels of the antioxidant chlorogenic acid”. Nature Biotechnology 22 (6): 746–54. (June 2004). doi:10.1038/nbt966. PMID 15107863.
- ^ “Tomato transgene structure and silencing”. Nature Biotechnology 23 (3): 287–9. (March 2005). doi:10.1038/nbt0305-287b. PMID 15765076.
- ^ “Comparative reactions of recombinant papaya ringspot viruses with chimeric coat protein (CP) genes and wild-type viruses on CP-transgenic papaya”. The Journal of General Virology 82 (Pt 11): 2827–36. (November 2001). doi:10.1099/0022-1317-82-11-2827. PMID 11602796.
- ^ “RNA interference-based silencing of the alpha-amylase (amy1) gene in Aspergillus flavus decreases fungal growth and aflatoxin production in maize kernels”. Planta 247 (6): 1465–1473. (June 2018). doi:10.1007/s00425-018-2875-0. PMID 29541880.
- ^ “RNA interference: a promising technique for the improvement of traditional crops”. International Journal of Food Sciences and Nutrition 64 (2): 248–59. (March 2013). doi:10.3109/09637486.2012.713918. PMID 22861122.
- ^ “Advances in RNA interference technology and its impact on nutritional improvement, disease and insect control in plants”. Applied Biochemistry and Biotechnology 169 (5): 1579–605. (March 2013). doi:10.1007/s12010-012-0046-5. PMID 23322250.
- ^ Downie, Rowena C.; Lin, Min; Corsi, Beatrice; Ficke, Andrea; Lillemo, Morten; Oliver, Richard P.; Phan, Huyen T. T.; Tan, Kar-Chun et al. (2021-07-27). “Septoria Nodorum Blotch of Wheat: Disease Management and Resistance Breeding in the Face of Shifting Disease Dynamics and a Changing Environment”. Phytopathology (American Phytopathological Society) 111 (6): PHYTO–07–20–028. doi:10.1094/phyto-07-20-0280-rvw. hdl:20.500.11937/83208. ISSN 0031-949X. PMID 33245254.
- ^ “Genetic engineering of Nicotiana tabacum for reduced nornicotine content”. Journal of Agricultural and Food Chemistry 54 (24): 9071–8. (November 2006). doi:10.1021/jf0610458. PMID 17117792.
- ^ “Transgenic resistance to tobacco ringspot virus”. Acta Virologica 48 (3): 145–52. (2004). PMID 15595207.
- ^ “RNAi-mediated replacement of morphine with the nonnarcotic alkaloid reticuline in opium poppy”. Nature Biotechnology 22 (12): 1559–66. (December 2004). doi:10.1038/nbt1033. PMID 15543134.
- ^ “Environmental RNAi in herbivorous insects”. RNA 21 (5): 840–50. (May 2015). doi:10.1261/rna.048116.114. PMC 4408792. PMID 25802407.
- ^ “Dissecting systemic RNA interference in the red flour beetle Tribolium castaneum: parameters affecting the efficiency of RNAi”. PLOS ONE 7 (10): e47431. (2012). Bibcode: 2012PLoSO...747431M. doi:10.1371/journal.pone.0047431. PMC 3484993. PMID 23133513.
- ^ “Corn rootworm-active RNA DvSnf7: Repeat dose oral toxicology assessment in support of human and mammalian safety”. Regulatory Toxicology and Pharmacology 81: 57–68. (November 2016). doi:10.1016/j.yrtph.2016.07.009. PMID 27436086.
- ^ “Development and characterization of the first dsRNA-resistant insect population from western corn rootworm, Diabrotica virgifera virgifera LeConte”. PLOS ONE 13 (5): e0197059. (2018-05-14). Bibcode: 2018PLoSO..1397059K. doi:10.1371/journal.pone.0197059. PMC 5951553. PMID 29758046.
- ^ “The EPA Quietly Approved Monsanto's New Genetic-Engineering Technology: It's the first time RNA interference will be used to kill insect pests.”. The Atlantic (2017年6月23日). 2022年4月4日閲覧。
- ^ Mongelli, Vanesa; Saleh, Maria-Carla (2016-09-29). “Bugs Are Not to Be Silenced: Small RNA Pathways and Antiviral Responses in Insects”. Annual Review of Virology (Annual Reviews) 3 (1): 573–589. doi:10.1146/annurev-virology-110615-042447. ISSN 2327-056X. PMID 27741406.
- ^ a b c Zhu, Kun Yan; Palli, Subba Reddy (2020-01-07). “Mechanisms, Applications, and Challenges of Insect RNA Interference”. Annual Review of Entomology (Annual Reviews) 65 (1): 293–311. doi:10.1146/annurev-ento-011019-025224. ISSN 0066-4170. PMID 31610134.
- ^ Matson RS (2005). Applying genomic and proteomic microarray technology in drug discovery. CRC Press. p. 6. ISBN 978-0-8493-1469-8
- ^ Zhang XHD (2011). Optimal High-Throughput Screening: Practical Experimental Design and Data Analysis for Genome-scale RNAi Research. Cambridge University Press. pp. ix–xiii. ISBN 978-0-521-73444-8
- ^ “Planting the Seeds of a New Paradigm”. PLOS Biol 2 (5): e133. (2004). doi:10.1371/journal.pbio.0020133. PMC 406394. PMID 15138502.
- ^ “Inhibition of gene expression in plant cells by expression of antisense RNA”. Proceedings of the National Academy of Sciences of the United States of America 83 (15): 5372–6. (August 1986). Bibcode: 1986PNAS...83.5372E. doi:10.1073/pnas.83.15.5372. PMC 386288. PMID 16593734.
- ^ “Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans”. The Plant Cell 2 (4): 279–289. (April 1990). doi:10.1105/tpc.2.4.279. PMC 159885. PMID 12354959.
- ^ “Quelling: transient inactivation of gene expression in Neurospora crassa by transformation with homologous sequences”. Molecular Microbiology 6 (22): 3343–53. (November 1992). doi:10.1111/j.1365-2958.1992.tb02202.x. PMID 1484489.
- ^ “Transgene-mediated suppression of chalcone synthase expression in Petunia hybrida results from an increase in RNA turnover”. Plant J 6 (6): 861–77. (1994). doi:10.1046/j.1365-313X.1994.6060861.x.
- ^ Antisense nucleic acids and proteins: fundamentals and applications. M. Dekker. (1991). pp. 4, 136. ISBN 978-0-8247-8516-1
- ^ “Plants combat infection by gene silencing”. Nature 385 (6619): 781–2. (1997). Bibcode: 1997Natur.385..781C. doi:10.1038/385781a0.
- ^ “Cytoplasmic inhibition of carotenoid biosynthesis with virus-derived RNA”. Proceedings of the National Academy of Sciences of the United States of America 92 (5): 1679–83. (February 1995). Bibcode: 1995PNAS...92.1679K. doi:10.1073/pnas.92.5.1679. PMC 42583. PMID 7878039.
- ^ “A similarity between viral defense and gene silencing in plants”. Science 276 (5318): 1558–60. (June 1997). doi:10.1126/science.276.5318.1558. PMID 18610513.
- ^ “par-1, a gene required for establishing polarity in C. elegans embryos, encodes a putative Ser/Thr kinase that is asymmetrically distributed”. Cell 81 (4): 611–20. (May 1995). doi:10.1016/0092-8674(95)90082-9. PMID 7758115.
- ^ “Cosuppression in Drosophila: gene silencing of Alcohol dehydrogenase by white-Adh transgenes is Polycomb dependent”. Cell 90 (3): 479–90. (August 1997). doi:10.1016/S0092-8674(00)80508-5. PMID 9267028.
- ^ “Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans”. Nature 391 (6669): 806–11. (February 1998). Bibcode: 1998Natur.391..806F. doi:10.1038/35888. PMID 9486653.
外部リンク
- Overview of the RNAi process, from Cambridge University's The Naked Scientists
- Animation of the RNAi process, from Nature
- NOVA scienceNOW explains RNAi – A 15-minute video of the Nova broadcast that aired on PBS, 26 July 2005
- Silencing Genomes RNA interference (RNAi) experiments and bioinformatics in C. elegans for education. From the Dolan DNA Learning Center of Cold Spring Harbor Laboratory.
- RNAi screens in C. elegans in a 96-well liquid format and their application to the systematic identification of genetic interactions (a protocol)
- 2 American ‘Worm People’ Win Nobel for RNA Work, from NY Times
- Molecular Therapy web focus: "The development of RNAi as a therapeutic strategy", a collection of free articles about RNAi as a therapeutic strategy.
- GenomeRNAi: a database of phenotypes from RNA interference screening experiments in Drosophila melanogaster and Homo sapians
- RNAi tools Archived 19 June 2018 at the Wayback Machine. Pre-designed and custom RNA Interference tools
- 塩見美喜子『RNA干渉』INCF Japan Node、2015年。doi:10.14931/bsd.3744。2022年4月17日閲覧。
- 『RNA干渉』 - コトバンク
